Title:Pf-Phospho: a machine learning-based phosphorylation sites prediction tool for Plasmodium proteins
期刊:BIB
中科院分区:2区
影响因子:13.994
发表时间:2022年6月27号
web服务器:http://202.54.249.134/DB/index.php
摘要
即使有几种有机硅工具可用于预测哺乳动物,酵母或植物蛋白的磷酸化位点,但目前尚无用于预测疟原虫蛋白磷脂的软件。然而,在过去的十年中,大量磷酸化 - 蛋白质数据的可用性以及机器学习的进展(ML)算法为质量系统破译磷酸化模式和开发基于ML的磷材料预测工具的机会开辟了机会。我们已经开发了PF磷酸,这是一种基于ML的方法,用于通过使用12 096个磷脂的大量数据集训练随机森林分类器,这是恶性疟原虫和Bergei疟原虫。在12个已知的磷脂中,有75%的地点已用于培训/验证分类器,而剩余的25%已被用作盲目测试的完全看不见的测试数据。令人鼓舞的是,PF磷酸可以预测具有84%敏感性,75%特异性和78%精度的非激酶磷脂材料。此外,它还可以预测五种质子激酶的激酶特异性磷酸材料 - PF PKG,恶性疟原虫,PF PKA,PF PK7,PF PK7和PBCDPK4,具有很高的精度。 PF-phospho(http://www.nii.ac.in/pfphospho。html)优于其他广泛使用的磷酸材料预测工具,这些工具已通过使用哺乳动物磷酸蛋白酶数据进行训练。它也已与其他广泛使用的资源(例如plasmodb,mpmp,PFAM)集成在一起,并通过Alphafold2进行了基于ML的预测结构。目前,PF磷酸是唯一用于基于ML的疟原虫信号网络的预测,是一个用户友好的平台,用于整合磷酸化信号的综合分析以及代谢和蛋白质 - 蛋白质 - 蛋白质相互作用网络。
1. 方法和数据集
数据集
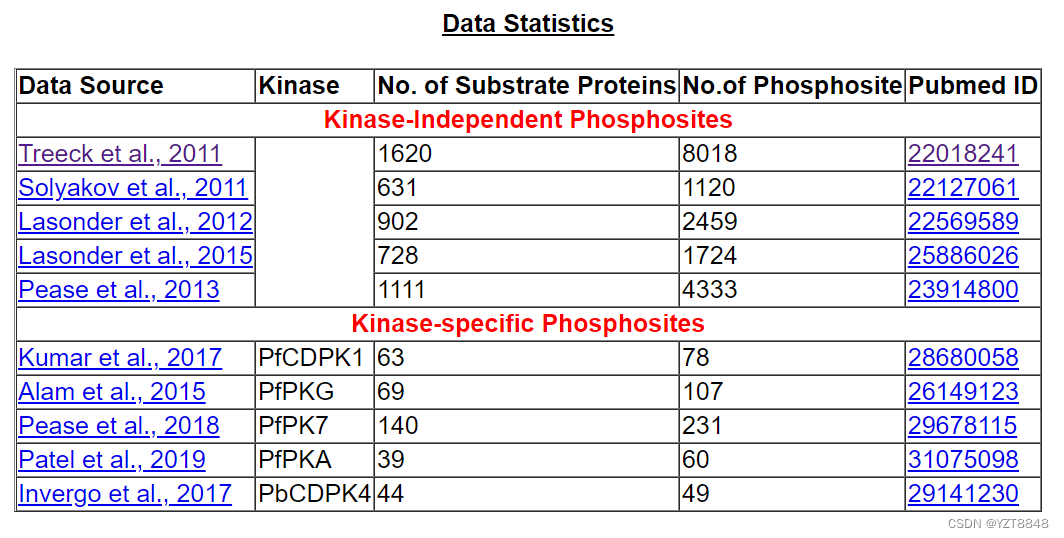
通过个别低通量实验以及磷蛋白组学研究确定的已知磷酸化点的现有数据被用来训练ML模型,以预测疟原虫蛋白中可能被磷酸化的S/T/Y残基。在文献中搜索疟原虫蛋白磷酸化的发生情况。由于实验验证的磷酸化位点的数据稀少,在不同的已发表的关于不同疟原虫物种的全球磷蛋白组学研究中确定的具有高置信度的磷酸化位点被编入目录并使用。1列出了文献中报道的野生型和基因敲除/封锁条件下的各种不同的全球磷蛋白组学研究,以及从每个研究中汇编的磷酸位点的数量。从Table 1[1, 24-33]中列出的九项不同的研究中,共汇编了1988种疟原虫蛋白质的12394个已知的磷酸酶位点的出现。这12394个来自1988年疟原虫蛋白的位点被用作建立独立于激酶的磷酸位点预测的ML模型的正面数据集。这1988个蛋白质中剩余的没有被磷酸化的丝氨酸/苏氨酸/酪氨酸残基被认为是阴性数据集。同样大小的阳性和阴性数据集被用于训练和测试(详见补充数据中的材料和方法)。以磷酸化残基为中心的13位多肽序列被用于训练和测试磷酸基点预测的ML模型。为了评估模型,我们从正反两方面分别保留了25%的13-mer序列,建立了一个基准数据集。其余75%的13-mer序列被用于训练模型。在特定激酶的正常与敲除/剔除条件下进行的差异性磷酸化蛋白质组学研究提供了有关被相应激酶磷酸化的位点的信息。基于这些研究,我们收集了PfPKG、PfCDPK1、PfPKA、PfPK7和PbCDPK4[1, 24, 27, 31, 32]的激酶特异性磷酸化位点数据(Table 1)。ML模型仅针对具有至少40个已知底物磷酸化位点的激酶建立,并且通过从S/T/Y中随机挑选被其他四种激酶磷酸化的相同数量的位点来创建特定激酶的负数据集(详见补充数据中的材料与方法)。在PfPKG、PfCDPK1、PfPKA、PfPK7和PbCDPK4[13, 14]的这些阳性和阴性数据集上,也评估了其他可用的公共领域磷酸化点预测工具NetPhorest-2.1和GPS 5.0的性能,使用其相应的激酶家族特定预测器(Table S3和Supple-mentary Data的材料和方法)。
方法
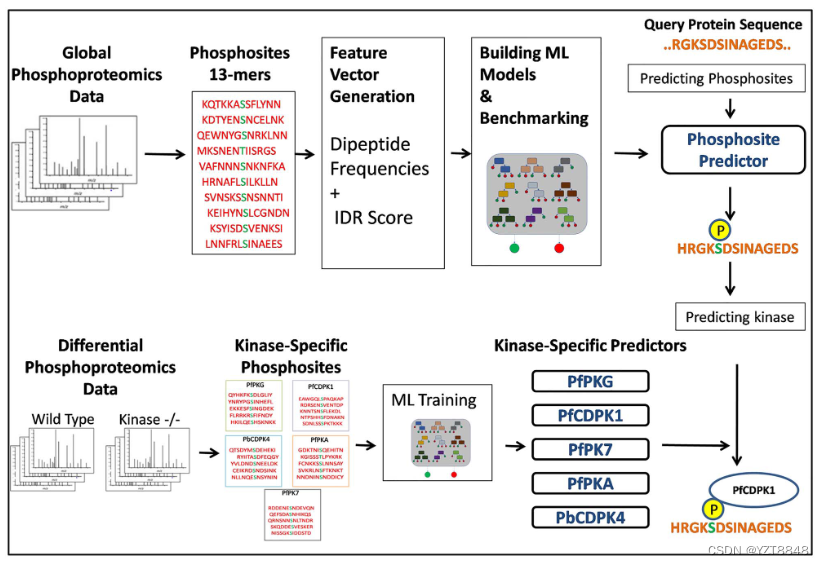
2.结果
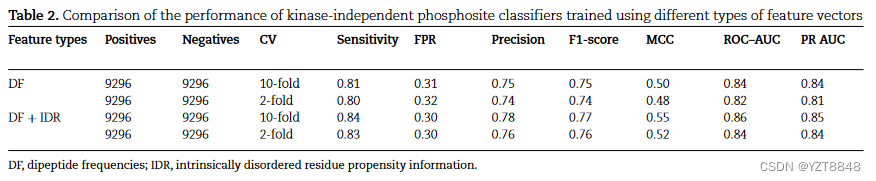
使用不同的算法(例如随机森林,Naivebayes,顺序最小优化(SMO)和深度学习)对ML分类器进行训练。随机森林是基于决策树的ML算法,而NaiveBayes是基于贝叶斯定理的简单算法。 SMO模型是基于输入数据点之间最大距离计算的优化培训的支持向量机(SVM)。深度学习分类器是WEKA工具箱中可用的DL4JMLP分类器。比较了10倍交叉验证(CV)中不同ML分类器的性能(表S2),并发现随机森林算法在ROCAUC(接收器操作特征曲线下的区域)值方面具有优于其他ML模型的性能。因此,在本研究和PF-Chospho Web服务器的实施中,随机森林被选为ML分类器。此外,还进行了2倍CV,以评估随机森林分类器的性能。 ROC和PR曲线显示了在整个得分截止范围内显示模型的性能,如图2所示,而在最佳分数截止下计算的混淆矩阵的其他统计参数的值如下2所示。从图2中的AUC值中可以看出,对于2倍和10倍CV分析,分类器的性能在特征向量中包括在二肽频率外,还包括13-Mer肽的固有疾病倾向时,分类器的性能略有增强。 。最佳性能模型,即包含IDP评分的10倍CV的ROC-AUC为0.86,PR-AUC为0.85,表明预测准确性略高。 2倍CV模型的相应AUC值分别为0.84和0.82,表明模型的收敛性在训练所需的数据量方面。表2显示了所有ML模型的最佳分数,显示了所有模型的灵敏度,特异性,精度,F1得分和Mathews的相关系数(MCC)。包含IDP评分的10倍CV模型的TPR(或灵敏度)为84%图3. ROC(A)和PR(B)曲线显示了与盲测的外部数据集对激酶无关的分类器的性能。 FPR为30%。相应模型的精度,F1得分和MCC值分别为0.78、0.77和0.55。受过二肽频率训练的模型在FPR值为31%的情况下显示为81%。这些结果表明,ML模型可以鉴定质子蛋白中的磷脂,其精度很高。基于这些结果,选择了所有进一步的磷材料预测工作和分析的特征,将二肽频率与IDR分数组合选择。
ML分类剂的盲试磷脂独立预测
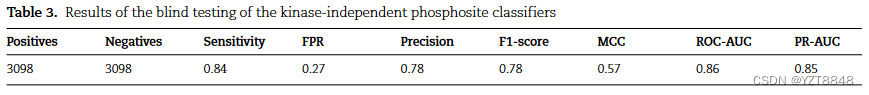
在CV分析之后,对具有二肽频率和IDR评分训练的激酶独立的随机森林分类器的性能进行了标准,该独立测试数据集被分开保存,不包括在训练模型中。该数据集由来自1192蛋白的3098个已知磷酸化位点组成。使用前面描述的相同方法,为该正集生成了相等大小的负数据集。然后在此组合的正面和阴性测试数据集上运行10倍CV激酶非依赖性的随机森林分类器,总计6196 s/t/y包含13-Mer肽,以检查模型是否可以正确识别已知的磷酸化位点。图3显示了对此独立数据集执行的盲测的ROC和精度 - 记录曲线。可以看出,ROC-AUC值为0.86,PR-AUC值为0.85。因此,即使在完全看不见的数据上,模型的性能也与CV数据集一样好。从表3可以看出,在最佳分数截止下,该模型在FPR时达到了84%的TPR,所有其他统计参数表明对独立测试数据的预测准确性很高。这些结果使我们有信心,我们的激酶非依赖性磷酸材料预测指标足以鉴定所有不同疟原虫蛋白质的磷脂,因此将是研究基于磷酸质子的信号传导的有用效用。






















 1463
1463











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








