如果你也想丰富文章图,或者想与你所研究的基因进行深入交流

那么你可能需要以下工具:进化树!!!简而言之,毕业无望,进化树来凑!
一分钟了解 进化树
进化树的全称叫系统发育树分析,
系统发育树分析:又名分子进化树,是生物信息学中描述不同生物之间的相关进化关系的方法。通过系统学分类分析可以帮助人们了解所有生物的进化历史过程,确定一个基因的进化历程。
它的样子是这样子的:

亦或者也是这个样子的:

进化树构建总体流程
以下流程为SunflowerTools:A package for Tree visualiazation and Data exploration的汉化:
这个工具还没有完全完成,它的定位是易操作的 易分析的 全面的 基因进化 分析工具
向日葵的构想来源:
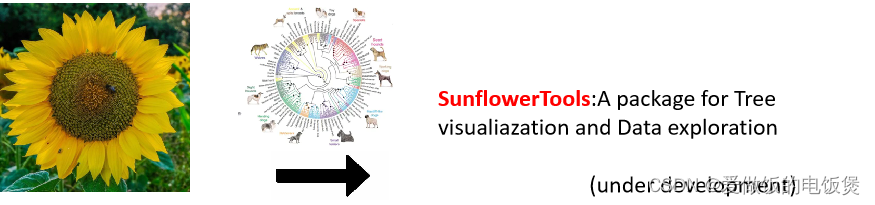
流程简介

基因组学有关知识点
中科大有门课,基因组学,有一部分也介绍了系统发育分析:
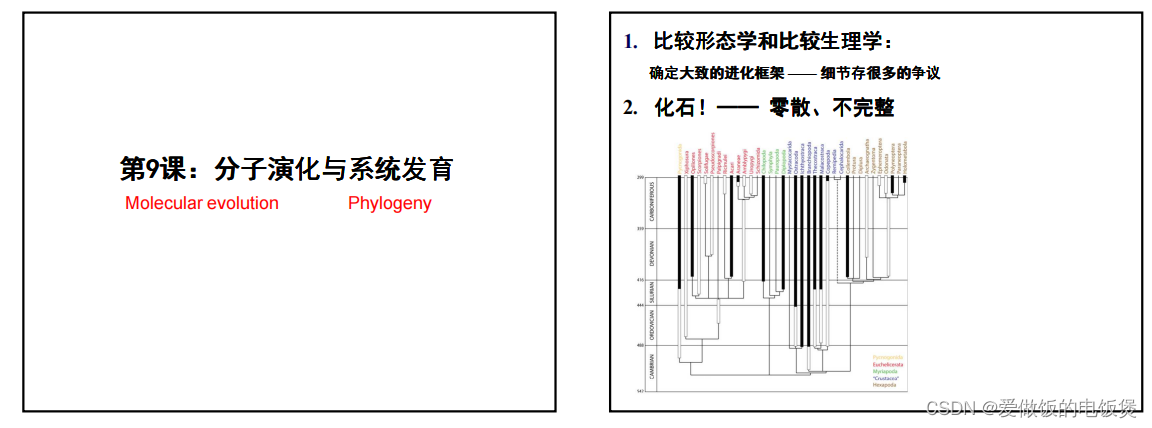
有关知识点如下:
1.第1个获得的个人基因组图谱:2007年Craig Venter 。
2.人类基因组单体型图 Haplotypemap
3.单体型(haplotype)
4.人类单体型图的主要结论的故事
5.连锁不平衡:LD linkage disequilibrium
6.国际千人基因组计划,在单碱基水平研究广泛人类变异。
7.人类基因组mapping图中的结构变异包括( )( )( )
8.人类基因组结构变异中的copy数变异包括( )、( / )
9.比较基因组杂交CGH:是将( 消减杂交 )和( 荧光原位杂交 )相结合,用于检测DNA序列的拷贝数变异,并将其定位在染色体上的方法。
10.CGH的限制在于不能够检测( )和( ),因为DNA的拷贝数没有发生改变,同时由于探针数量的限制,分辨率是100kb作用,相当数量的变异检测时取决于那组探针被使用。
11.深度测序检测SV的方法有①覆盖度深度的方法 ②( ) ③( )④( )
12.列出你所的疾病数据库(OMIM pubmed malacards )
13.研究生物进化的常见方法( )( )( )。
14.同源(homology):来源于共同祖先的( )。
15.直系同源 orthologs:不同物种中来源于共同祖先的同一基因。
16.旁系同源 paralogs:在同一物种中,由于( )而产生的基因,可能会进化出与原来功能相关的基因。
17.用于分子进化分析中的序列必须是( )的才可以反映其进化的过程。
18.异同源(xenolog)通过某一个基因( )得到的同源序列,来源于共生或病毒侵染所产生的相似基因
19.趋同进化(convergent evolution):不同的生物通过不同的进化途径获得相似的功能,如鲸鱼、海豚和鱼类。
20.同源分为3种( )、( )、( ),其中只有( )才可以用于分子进化分析以反映其进化的过程。
21.什么是分子钟假说(molecular clock hypothesis),中性理论为其提供了怎样的支持?
Pauling等认为某一蛋白在不同物种间的( )与所研究物种间的( )接近正线性关系,进而将分子水平的这种( )称为分子钟。
日本群体遗传学家木村资生(motookimura)提出中性理论:(1)进化过程中的核苷酸置换其绝大部分是( )或( )的突变随机固定的结果,而不是正向达尔文选择的结果。(2)许多蛋白质多态性必须在选择上为中性或近中性,并在群体中由突变引入和随机灭绝二者之间的维持平衡。
中性理论认为绝大部分氨基酸置换是中性或近乎中性,分子水平的恒速变异的分子钟假说在此理论下成立。
22.系统发育树 phylogenetic tree 用一种类似树分支的图像来概括一组对象( )之间的世系关系。
23.分子系统树 通过比较不同生物生物大分子序列差异的数值构建的系统树称为 分子系统树。
24.系统发育树是一种( ),由一系列节点nodes和分支branches组成,其中每个节点代表1个( )(物种或序列),而节点之间的连线则代表物种之间的( )。
25.系统发育树的组成结构,为什么系统发育树一般都是二叉树?
26.无根树的拓扑结构反映( ),有根数的拓扑结构反映( ),通过添加( ),无根树可以转变为有根树。
27.系统发育树的种类有哪2种?解释它们各自的含义,它们的不同之处在哪里?
答:系统发育树有基因树和物种树2种,基因树是指由来自各个物种的1个基因构建的系统发育树,表示基因分离的时间,物种树代表1个物种或群体进化历史的系统发育树,表示2个物种分歧的时间。物种树种的内节点分支代表物种形成,它代表着生殖隔离的形成。基因树种的基因蛋白可以在新物种形成前后复制或进化,基因树与物种树的拓扑结构不同。
28.简述构建系统发育树的方法
(1)挑选待分析的序列
(2)多序列比对
(3)选择建树模型
(4)构建系统发育树
(5)系统发育树检验可靠性分析、
(6)系统发育树的美化
一步一步的教程
ggtree
x <- read.tree("C:/Users/Newick") ##更换目录
library("ape")
library("ggtree")
groupInfo <- split(x$tip.label, gsub("_\\w+", "", x$tip.label))
y <- groupOTU(x, groupInfo)
ggtree(y, aes(color=group), layout='circular') + geom_tiplab(size=1, aes(angle=angle))
# 加载R包
# 读取树文件(ggtree针对不同工具生成的树文件有不同的函数进行读取,如没有对应的函数,可以使用通用的办法读取)
# 读取分组信息
# 按类分组
# 将分组信息添加到树中
tree <- groupOTU(x, groupInfo)
# 绘制进化树
ggtree(tree, layout="fan", ladderize = FALSE, branch.length = "none",aes(color=group)) + geom_tiplab2(size=3) + theme(legend.position = "right")
输出结果:

Mega 7.0
Mega使用起来简单,易上手,无需编程基础,下面将对其进行介绍:
MEGA软件作为一种用于分析物种鉴定、亲缘关系、分子功能等的工具,已经越来越成为生物学研究的常用软件。
- Step1: 序列比对
- Step2: 建立进化树
- Step3: 进化树美化
iTOL



























 701
701

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










