2、ClustalX进行多重序列比对
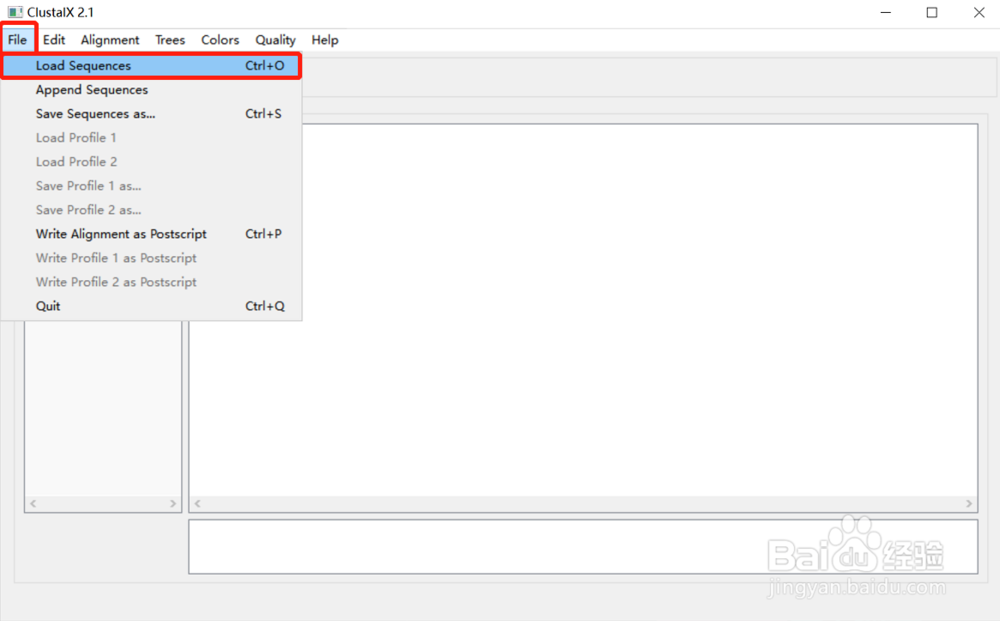
2.1 首先,打开ClustalX,依次在菜单栏选择“File”-“Load Sequences”导入我们的目的序列(fas格式)。
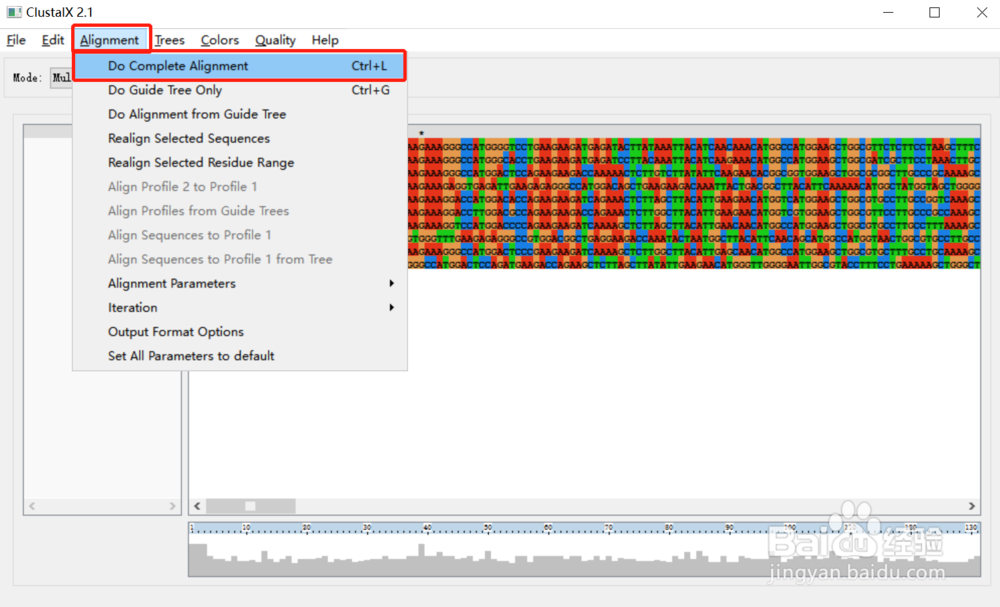
2.2 然后进行完全比对,依次选择“Alignment”-“Do Complete Alignment”
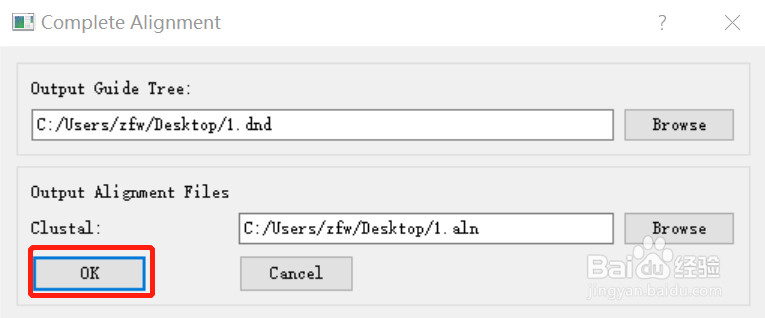
2.3 选择保存路径,点击“OK”。一般默认生成两个文件,分别是*.aln和*.dnd文件,可以用“记事本”打开。
-
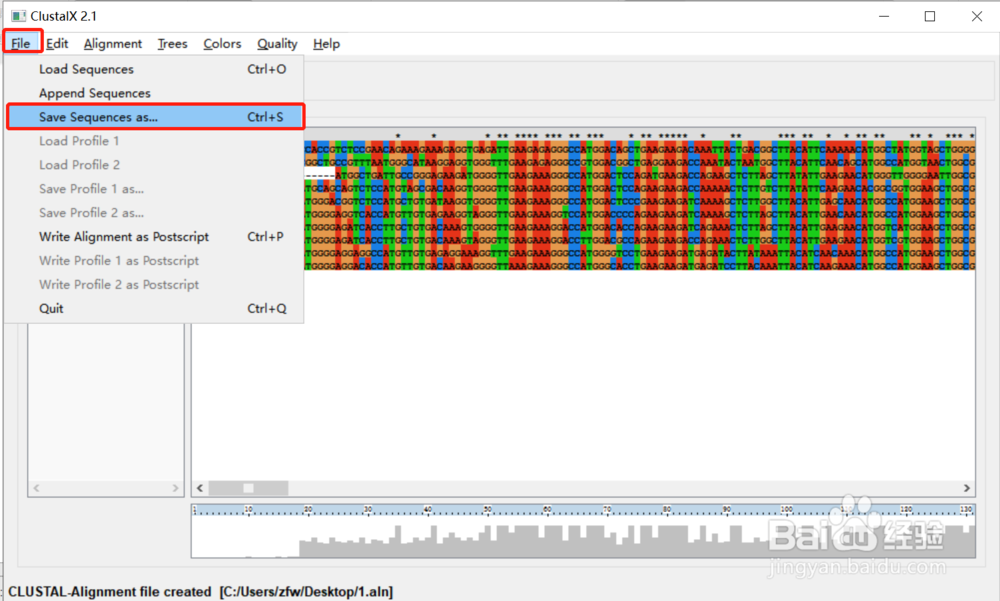
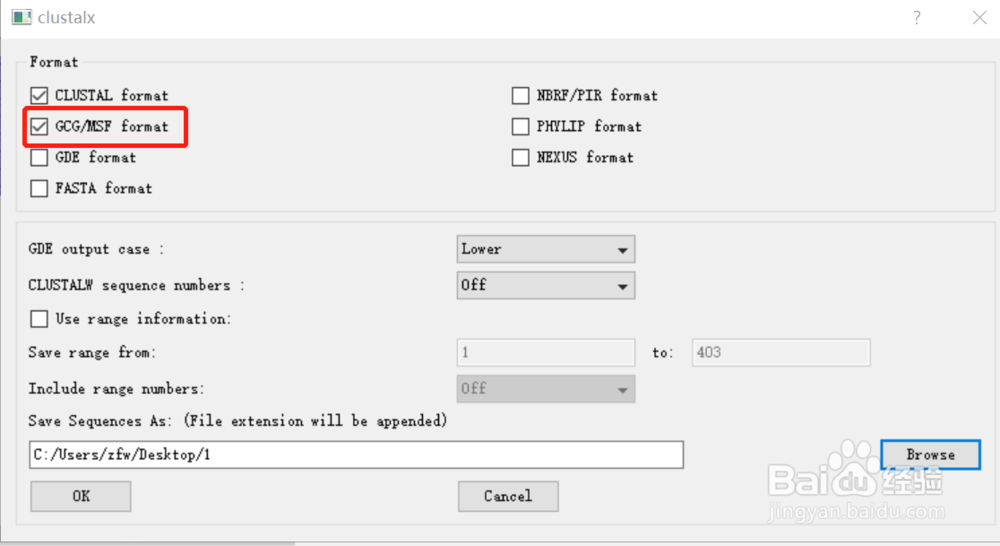
由于*.aln和*.dnd这两个文件的可读性不是太好,因此我们需要将文件保存成*.msf文件绘制多重比对效果图。依次点击“File”-“Save Sequences as”,然后选择“GCG/MSF format”,将多重序列比对结果保存成msf文件。
3. 利用GeneDoc对ClustalX的结果进行美化
GeneDoc是蛋白质和DNA序列同源比较的辅助软件,它不能对多个序列以某种算法进行自动比较,但是能对其他软件的比较结果进一步处理。如编辑和修改,用亮丽的色彩来区分相互间序列的同源性,并输出各种漂亮的图形格式。
GeneDoc下载地址:https://github.com/karlnicholas/GeneDoc。
3.1.导入
将之前ClustalX比对后保存的msf文件,依次选择“File”-“Open”,导入文件
3.2.美化设置
点击Project → Configure 弹出配置界面,这里可以对氨基酸的大小,每行氨基酸的个数、颜色以及显示模式等进行设置。
点击Project → Edit Sequences List,可以修改氨基酸序列的顺序或者修改序列名称。
如果发现基因ID无法完全显示,打开project的configure,右下角有一个“max 10”修改数字即可
3.3.导出
编辑好序列后,下面就是导出图片了。具体操作为:点击 Edit → Select Blocks for Copy,选择要导出的序列。
然后点一下序列,这样就选中了全部的序列,整个背景会变为黑色。
然后再点击 “Edit” -“Copy Select Blocks to”-“MetaFile”,等于已经将图片复制,然后打开电脑自带的画图或者PS,直接ctrl+V粘贴,然后保存图片即可。

参考链接:
https://jingyan.baidu.com/article/90808022869597bc91c80fd6.html
https://www.jianshu.com/p/f7efa9e1af35





















 3万+
3万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








