小罗碎碎念
本期文献主题:人工智能在病理组学中的系统研究进展
五月和六月分享了很多与病理组学相关的文章,为了进一步的缩小研究范围,在老板的指点下,整理了十篇文献。
这十篇文献里,第六、十篇是病理组学领域的通用基础模型,可以减少大家在训练阶段的工作量;其余的文献不一一介绍了,基本把目前病理组学领域的研究热点都包含进去了:
- 生物标志物
- 多模态
- 分子亚型分类
- 免疫治疗
- 预后预测/分析

PS:为了减轻大家工作量,以上十篇文献的原文,我会打包发到交流群里。
一、基于回归的深度学习方法,从病理切片中预测分子生物标志物

文献概述
这篇文章讨论了一种基于回归的深度学习方法,用于从病理切片中预测分子生物标志物。
研究团队开发并评估了一种自监督的、基于注意力机制的弱监督回归方法,该方法能够直接从11,671张覆盖九种癌症类型的患者图像中预测连续的生物标志物。
研究发现,使用回归方法可以显著提高生物标志物预测的准确性,并且与分类方法相比,能够更好地对应于已知的临床相关区域。
在结直肠癌患者的大型队列中,基于回归的预测分数比基于分类的分数提供了更高的预后价值。此外,该研究还探讨了如何通过数字病理学分析组织样本来提供有关肿瘤分级、亚型、分期和其他肿瘤生物标志物的信息。
研究结果表明,回归方法在计算病理学中为连续生物标志物分析提供了一个有希望的替代方案。
文章还提到了数字病理学的发展,以及如何利用深度学习技术从全切片图像(WSI)中预测基因变化和基因表达模式。
研究团队通过对比增强的聚类注意力多实例学习(CAMIL)回归方法与传统的分类方法进行了系统比较,发现CAMIL回归方法在多个数据集、器官和生物标志物上的表现优于传统方法。
此外,文章还讨论了回归方法在预测肿瘤微环境中的关键生物过程标志物方面的优势,包括肿瘤细胞、基质和免疫细胞的相关生物标志物。
研究结果表明,CAMIL回归方法能够以高AUROC值预测这些生物标志物,并且在与CAMIL分类方法和Graziani等人提出的回归方法相比时,表现出更好的性能。
最后,文章还探讨了CAMIL回归方法在预测结直肠癌患者总体生存方面的应用,并发现基于回归的生物标志物能够提供更好的预后预测。
研究团队认为,他们的开源回归方法为病理学中的连续生物标志物分析提供了一个有希望的替代方案,并对精准医疗产生重要影响。
重点关注
端到端实验工作流程的概览——图像预处理、建模、性能指标评估以及所使用的队列。
以下是详细分析:
A. 图像预处理流程和瓦片级特征提取:
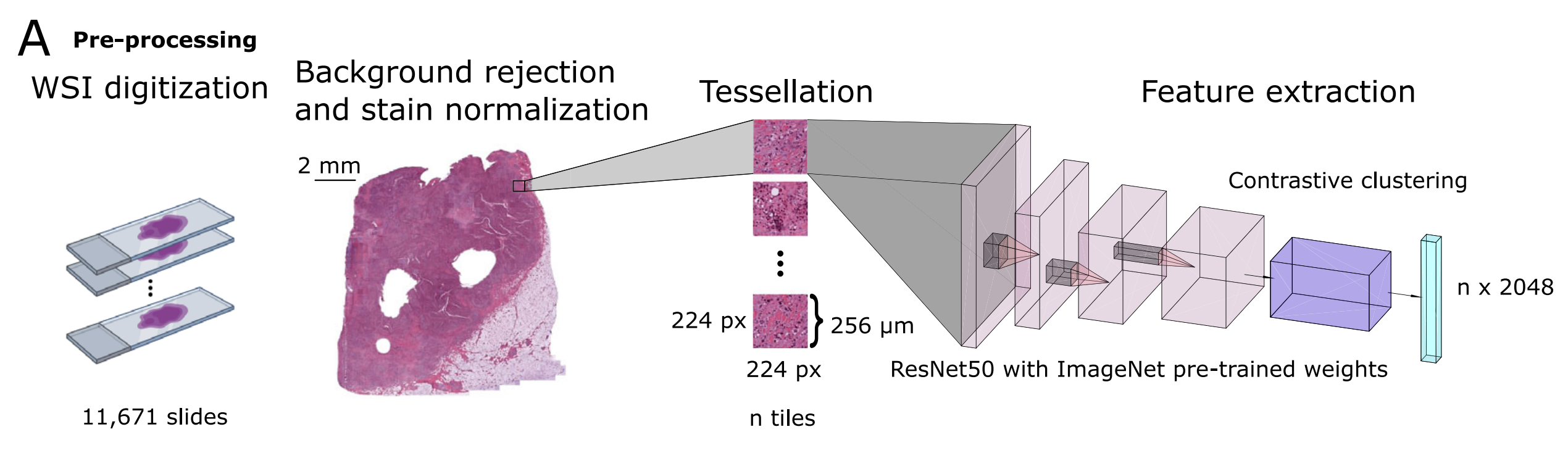
- 使用带有预训练的ImageNet权重的
ResNet50模型进行推理,以及检索对比聚类(RetCCL)模型,为每个患者生成特征矩阵。 - 这一步骤涉及将全切片图像(WSI)分割成小块(tiles),并对这些小块进行处理,以提取可用于后续分析的特征。
B. 建模架构:
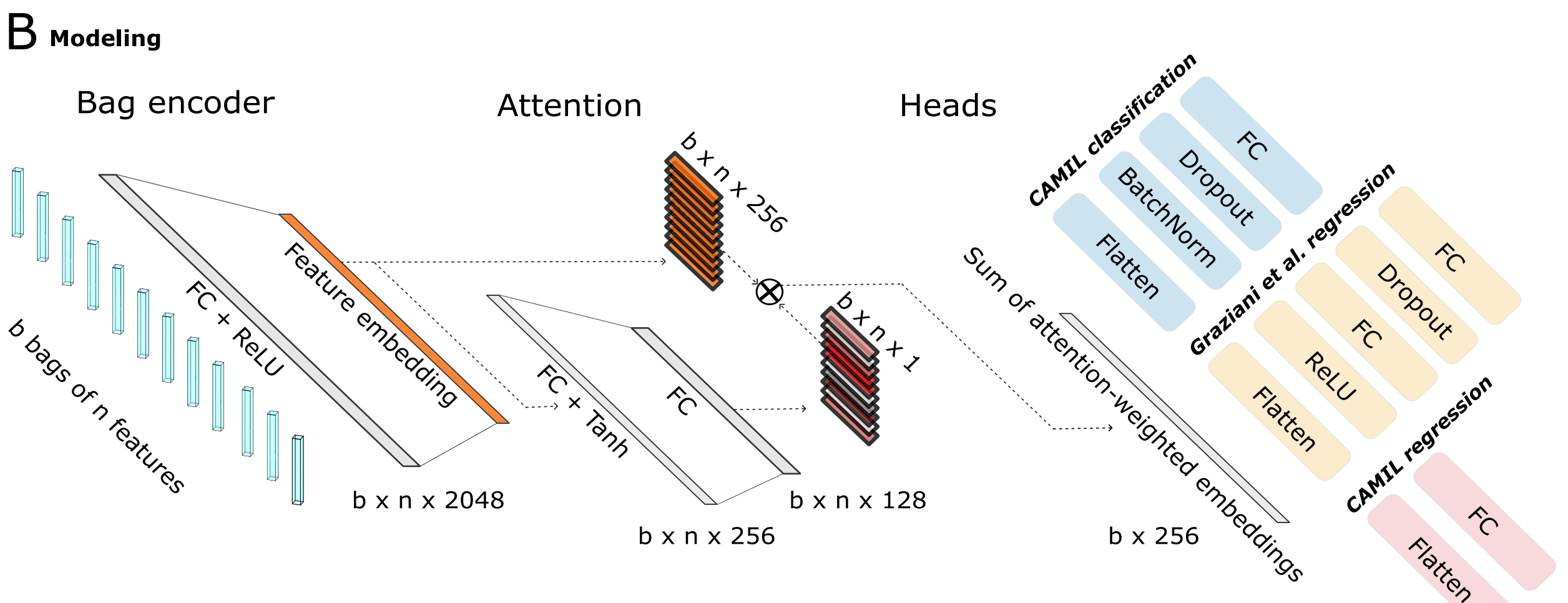
- 利用基于注意力的
多实例学习(attMIL)应用在自监督提取的特征上。 - 该架构包含三个独立训练的头(heads):
- 一个用于CAMIL分类。
- 一个用于回归,遵循Graziani等人提出的方法。
- 第三个用于本研究介绍的CAMIL回归方法。
C. 性能指标及其置信区间(CIs):
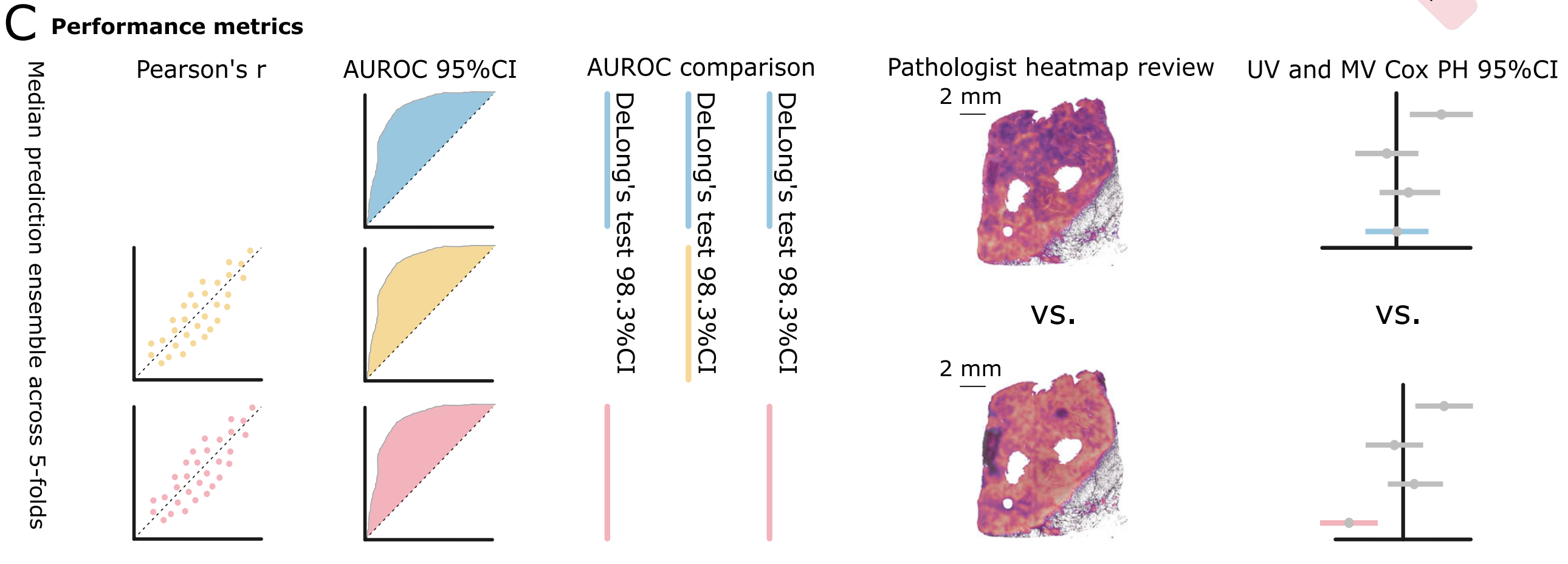
- 评估模型的三个独立训练头的性能指标包括:
- 回归模型使用皮尔逊相关系数(Pearson’s r)。
- 所有模型使用接收者操作特征曲线下面积(AUROC)。
- 对同源重组缺陷(HRD)和生物过程生物标志物进行了配对双尾DeLong测试。
- 进行了注意力热图的专家评审,以及对生物过程模型进行了单变量(UV)和多变量(MV)Cox比例风险(PH)模型分析。
D. 队列图表表示:
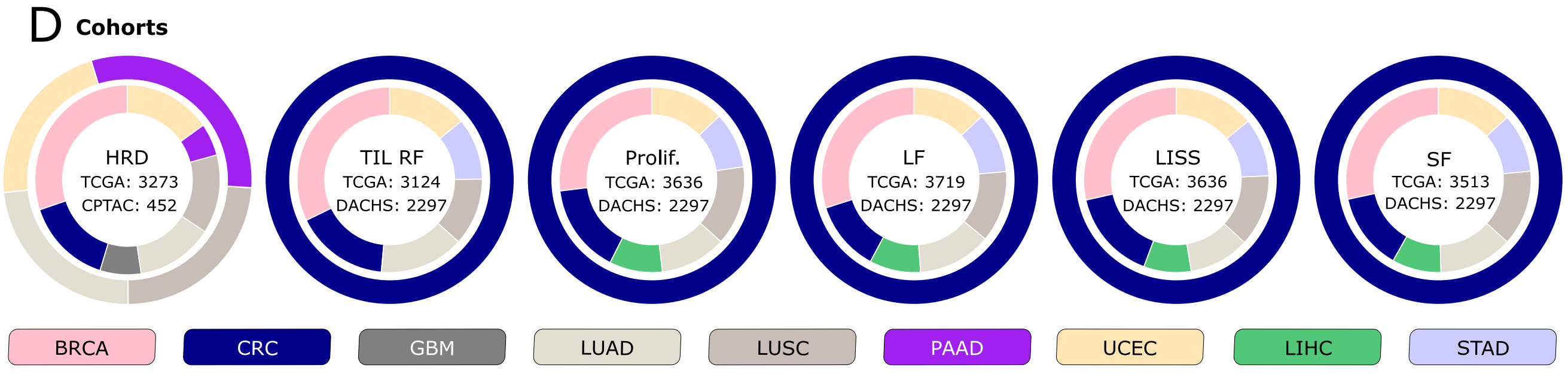
- 展示了本研究中使用的队列,内外圈分别表示用于训练和外部验证的队列。
- 训练队列来源于癌症基因组图谱(The Cancer Genome Atlas, TCGA)项目的所有临床目标。
- 外部验证队列来自临床蛋白质组肿瘤分析联盟(Clinical Proteomic Tumor Analysis Consortium, CPTAC)的努力,以及Darmkrebs: Chancen der Verhütung durch Screening (DACHS)研究,分别针对HRD目标和生物过程生物标志物。
- 考虑的
生物过程生物标志物包括肿瘤浸润淋巴细胞区域分数(TIL RF)、增殖(Prolif.)、白细胞分数(LF)、淋巴细胞浸润特征分数(LISS)和基质分数(SF)。 - 研究中考虑的
癌症类型包括乳腺癌(BRCA)、结直肠癌(CRC)、胶质母细胞瘤(GBM)、肺腺癌(LUAD)、肺鳞状细胞癌(LUSC)、胰腺腺癌(PAAD)、子宫内膜癌(UCEC)、肝细胞癌(LIHC)和胃癌(STAD)。
这个流程图提供了研究方法的清晰视觉表示,从图像预处理到模型评估,再到使用的队列,为读者呈现了研究的全貌。
二、深度学习+病理组学,对用于临床试验的患者进行预筛选

文献概述
这篇文章描述了一种基于组织病理学图像的深度学习算法的开发和部署,该算法用于临床试验中的患者预筛选。
作者提出的算法能够识别肿瘤中的遗传变异,例如成纤维细胞生长因子受体(FGFR),这对于靶向治疗至关重要。然而,分子测试可能因时间和组织需求量而延迟患者护理。该AI算法的开发、验证和部署能够降低筛选成本并加快患者招募。
研究团队使用了3000多张来自晚期尿路上皮癌患者的H&E染色全切片图像来训练深度学习算法,优化其高灵敏度以避免排除符合试验条件的患者。
该算法在一个包含350名患者的数据集上进行了验证,实现了0.75的曲线下面积(AUC)、在88.7%的灵敏度下具有31.8%的特异性,并预计可以减少28.7%的分子测试。
研究团队成功地将该系统部署在一个非干预性研究中,涵盖了89个全球研究临床场所,并展示了其在优先分配/取消分配分子测试资源以及在药物开发和临床环境中提供重大成本节约方面的潜力。
文章还讨论了将这种基于H&E图像的FGFR+筛查设备部署在临床试验或临床实践中的潜在价值,包括通过避免对不太可能携带遗传突变的患者进行分子测试来降低成本,以及通过为医生提供快速、可行的见解来减少患者入组或接受适当靶向治疗的时间(例如,通过丰富可能为FGFR+的患者队列)。
此外,文章还提到了算法的开发、验证和部署的具体过程,包括使用公共数据库、商业来源和内部临床试验的数据集,以及算法在多个独立大规模数据集和前瞻性实时临床环境中的稳健验证。研究团队还展示了该技术如何通过减少筛查负担和提高试验效率来改善临床护理,并推动精准医疗的发展。
重点关注
研究设计、数据集使用以及从算法开发到验证和部署的工作流程。
以下是对图内容的分析:
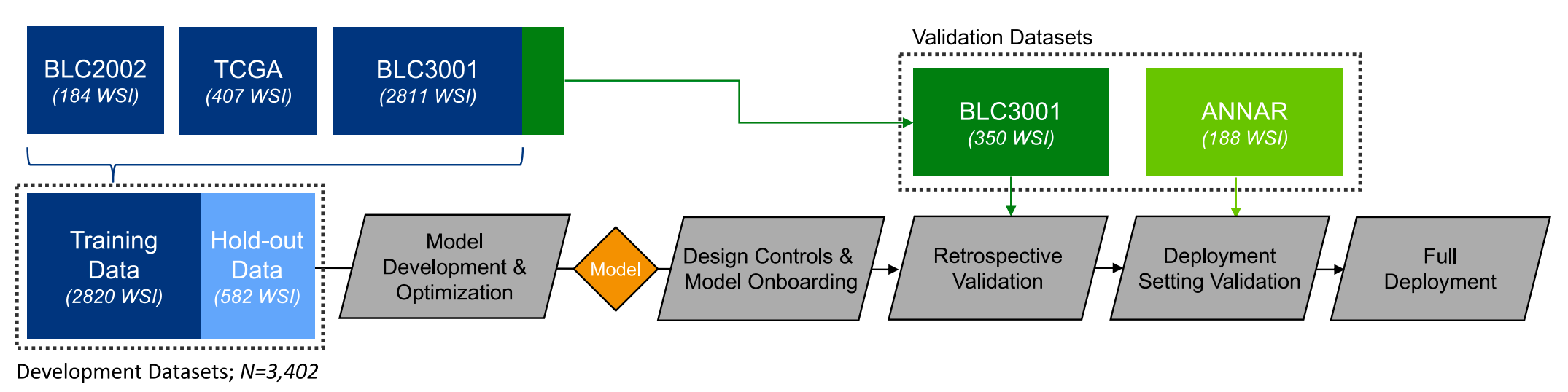
-
模型开发数据集:使用了来自三个不同队列的全切片图像(WSI)进行模型开发:
- 407张来自癌症基因组图谱(The Cancer Genome Atlas, TCGA)联盟。
- 3161张来自BLC3001(NCT03390504)的临床试验。
- 184张来自BLC2002(NCT03473743)的临床试验,这两个试验均与erdafitinib(一种药物)相关。
-
数据集的划分:从BLC3001队列中特别挑选了350个样本(150个FGFR阳性,200个FGFR阴性)用于模型的回顾性验证。这个子集被富集了FGFR阳性样本,以实现大约93%的统计功效。
-
独立验证数据集:此外,还从ANNAR(NCT03955913)试验中留出了188个样本,用于在算法打包成可部署设备并集成到部署平台后进行回顾性验证。
-
避免数据重复使用:确保没有患者样本同时在开发和回顾性验证中使用,以保证验证的独立性和结果的可靠性。
-
探索性分析:在工具部署后,为了评估算法在实体瘤上的性能,使用了来自不同肿瘤组织的361张切片组成的额外队列(即PAN-Tumor),这作为探索性分析。
-
算法部署:算法经过开发和优化后,被打包成一个部署设备,并集成到了部署平台上。
-
前瞻性应用:在ANNAR试验中,算法被前瞻性地应用于患者样本,以评估其在实际临床工作流程中的整合情况。
上图提供了一个清晰的视觉表示,说明了算法开发的严谨过程,包括数据的收集、模型训练、验证以及最终的部署。这个过程遵循了科学和临床验证的标准,确保了算法的可靠性和有效性。
三、HECTOR|用于预测子宫内膜癌患者远处复发风险的多模态模型

文献概述
这篇文章是发表在《Nature Medicine》上的一项研究,我前两天的推文也推荐过这篇文章,为什么要再次拿出来说呢?因为……我明天有一篇推文就是关于这篇文献的精析。
研究团队开发了一个名为HECTOR(histopathology-based endometrial cancer tailored outcome risk)的多模态深度学习预测模型,用于预测子宫内膜癌患者的远处复发风险。
HECTOR模型利用苏木精-伊红染色的全切片图像(whole-slide images, WSIs)和肿瘤分期作为输入,对来自八个子宫内膜癌队列的2072名患者进行了训练和测试,包括PORTEC-1/-2/-3随机试验。
HECTOR模型在内部测试集(353名患者)和两个外部测试集(分别为160名和151名患者)上展示了出色的预测性能,C指数(concordance index, C-index)分别为0.789、0.828和0.815,超过了当前的金标准。此外,HECTOR模型还能更好地预测辅助化疗的效果。通过对形态学和基因组特征的提取,研究团队发现了与HECTOR风险组相关的因素,其中一些具有治疗潜力。
HECTOR模型的开发为个性化治疗提供了新的工具,有助于改善子宫内膜癌患者的治疗决策。研究还探讨了HECTOR模型的解释性,通过分析模型风险评分与已知预后因素的关联,以及输入数据对预测的贡献,为理解HECTOR模型提供了更深入的生物学见解。
研究结果表明,HECTOR模型在预测远处复发风险方面具有潜力,并且可能成为未来临床实践中的一个有效工具。
重点关注
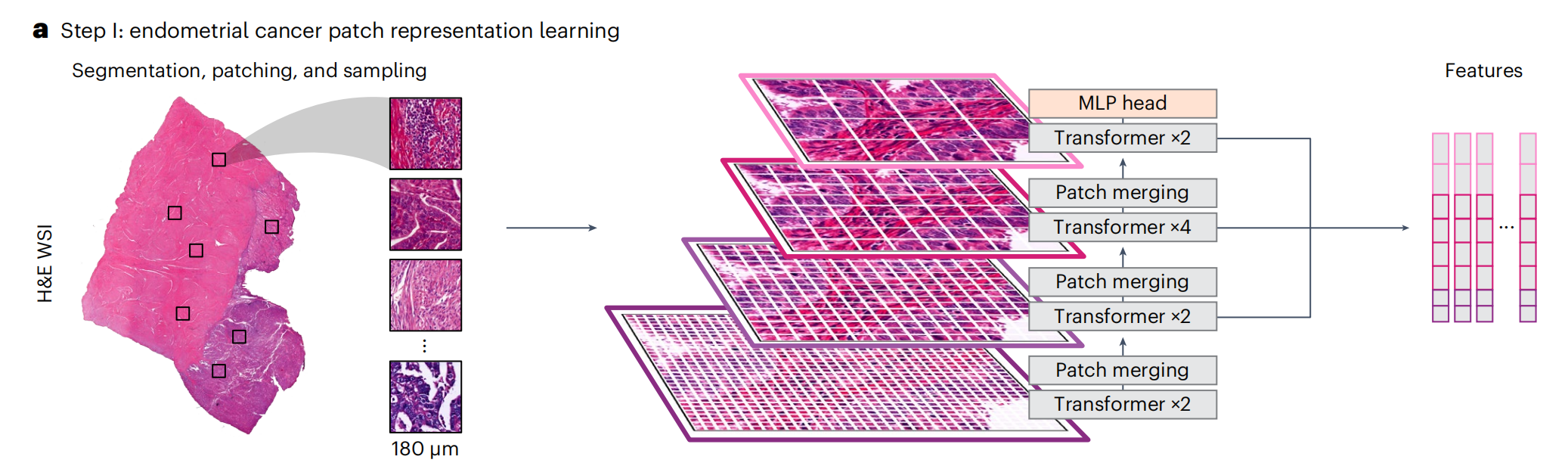
HECTOR模型的概览
a部分:
- 描述了从
子宫内膜癌(EC)的苏木精-伊红染色全切片图像(H&E WSI)中分割组织,并将其划分为180微米大小的区域(称为patches)的过程。 - 使用
多阶段视觉变换器(multistage vision transformer),通过自监督学习的方法,从1862名患者的WSIs中随机抽取图像块进行训练,这些患者不包括在内部和外部测试集中。 - 从变换器的最后八个块中提取图像块级别的特征。
b部分:
- 展示了HECTOR模型如何接受H&E WSI和根据FIGO 2009标准分类的解剖学分期(I-III期)作为输入。
- 提取的图像块级特征在空间和语义上进行了平均处理。
- 这些特征被输入到一个
基于注意力机制的多重实例学习模型(attention-based multiple instance learning model)和im4MEC深度学习模型中(所有层都是冻结的),后者能够从H&E WSI预测分子类别,如imPOLEmut、imMMRd、imNSMP或imp53abn。 - 解剖学分期和基于图像的分子类别都通过嵌入层(Embedding layers)进行处理。
- 使用
基于门控的注意力机制(gating-based attention)对这三个嵌入结果进行加权,然后通过Kronecker积进行融合。 - 使用
负对数似然损失函数(−log(likelihood loss))来预测在离散时间上的无远处复发概率函数。 - 风险评分定义为综合预测概率。
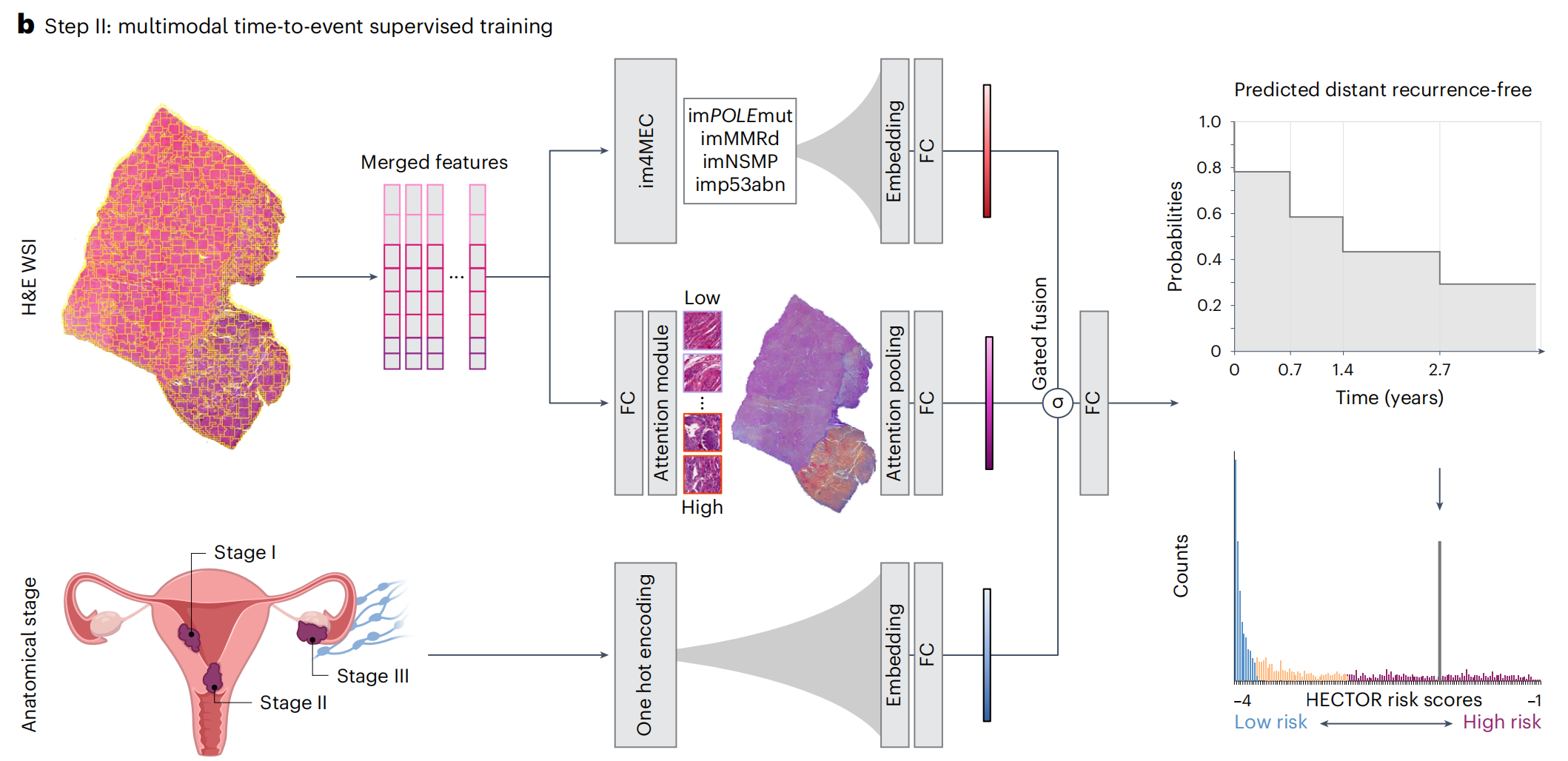
此外,图1还提到了多层面感知器(MLP, multilayer perceptron)和全连接层(FC, Fully Connected layer),这些是深度学习中常见的网络结构,用于处理和学习数据特征。
总体而言,HECTOR模型通过结合组织形态学特征、分子类别和解剖学分期信息,使用深度学习技术来预测子宫内膜癌患者的远处复发风险。
四、TORCH:基于细胞学图像的深度学习方法,区分未知原发部位的肿瘤起源

文献概述
这篇文章开发了一种基于细胞学图像的深度学习方法,名为TORCH(Tumor Origin differentiation using cytological histology),用于区分未知原发部位的肿瘤(CUP)的起源。
CUP是一种难以诊断的恶性肿瘤,因为其原发部位不明。研究者利用来自四家医院的57,220例患者的细胞学图像,训练了TORCH模型,以识别恶性肿瘤并预测肿瘤起源。
该模型在内部和外部测试集上的表现都非常好,癌症诊断的AUROC(接收者操作特征曲线下面积)值在0.953到0.991之间,肿瘤起源定位的AUROC值在0.953到0.979之间。TORCH在预测原发肿瘤起源方面表现出色,top-1准确率达到82.6%,top-3准确率为98.9%。
与病理学家的结果相比,TORCH显示出更好的预测效果,显著提高了初级病理医生的诊断分数。此外,与TORCH预测的肿瘤起源一致的患者,其总体生存期比接受不一致治疗的患者要好(27个月对比17个月)。研究强调了TORCH作为临床实践中有价值的辅助工具的潜力,尽管需要在随机试验中进一步验证。
研究还探讨了模型的解释性,使用注意力热图来解释模型预测结果,显示了模型预测中最重要的细胞学特征。此外,研究还对模型进行了消融实验,以评估临床变量与细胞学图像对模型性能的影响。最后,研究通过回顾性生存分析,评估了TORCH模型预测与患者治疗响应之间的关系。
整体而言,这项研究展示了深度学习在病理诊断领域的应用潜力,尤其是在处理未知原发肿瘤的诊断难题上。
重点关注
Fig. 1 展示了所提出的 TORCH 模型框架,该框架是一个深度学习系统,用于分析细胞学图像并预测未知原发肿瘤的起源。
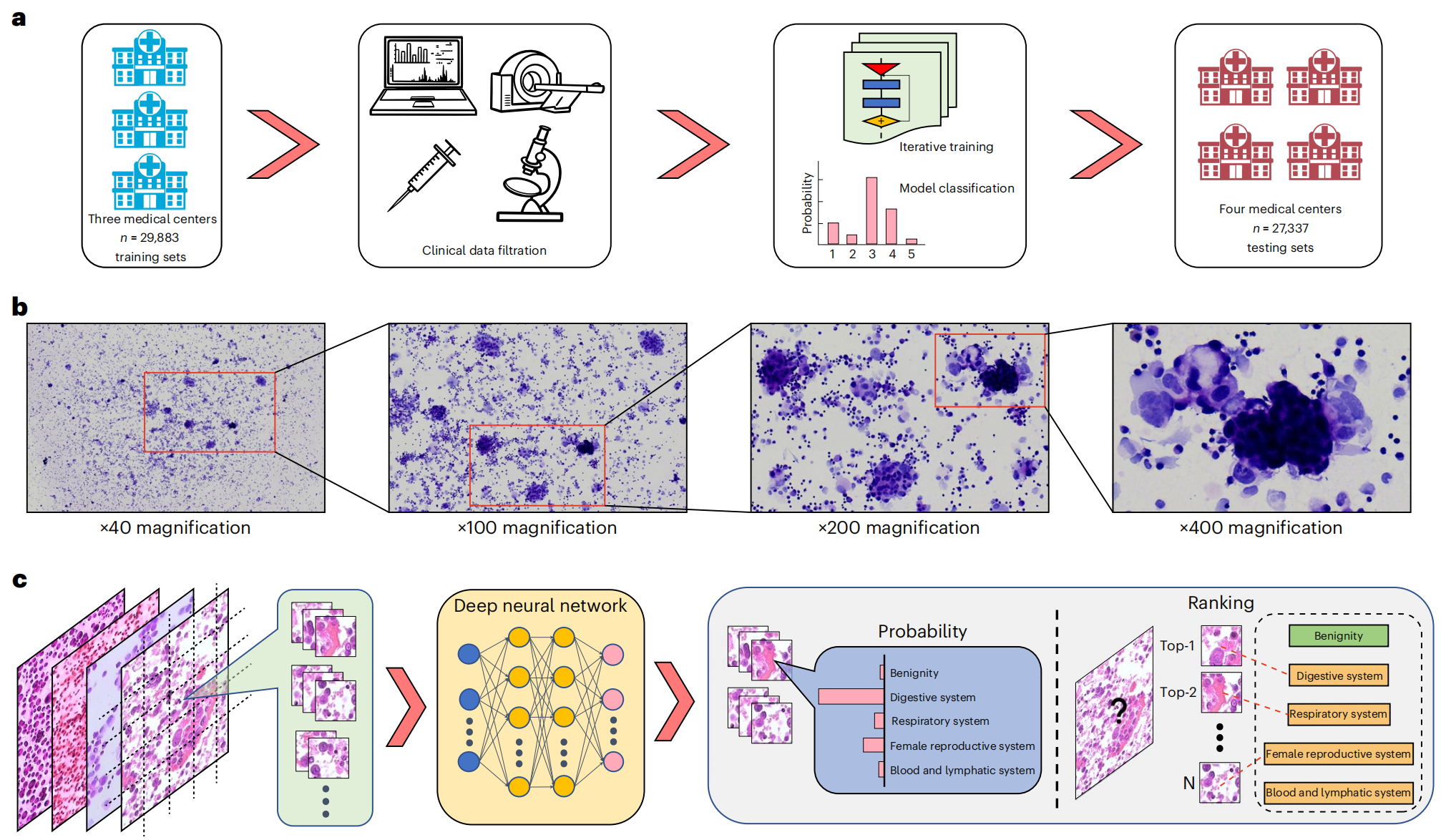
a. 模型训练集的构建:
研究者从三家大型三级转诊机构收集了总共 42,682 例病例数据,其中 70%(29,883 例)被用作训练集。这些临床病理数据是从放射影像科、病历系统和病理数字数据库中获取的。这表明研究者拥有一个大规模、多源的数据集来训练他们的深度学习模型。
b. 诊断过程中的图像放大:
在诊断过程中,大多数图像被放大到 ×200 或 ×400 的倍数进行观察。这通常是为了更清晰地观察细胞的形态特征,这些特征对于区分不同类型的肿瘤细胞至关重要。
c. 深度学习网络的目标和验证:
训练的深度学习网络使用细胞学图像,旨在根据最高预测概率分数将目标图像分为五个类别。这意味着模型将输出一个概率最高的类别,作为预测的肿瘤起源。分类结果在包括三个内部测试集(12,799 例)和两个外部测试集(14,538 例)的四个机构中进一步进行了验证。这显示了模型的泛化能力和在不同数据集上的可靠性。
N 代表第 N 个图像瓦片:在深度学习模型中,图像通常被分割成多个小的瓦片(tiles),每个瓦片都是模型输入的一部分。这有助于模型更细致地分析图像的不同区域,并提高整体的预测精度。
总体而言,Fig. 1 描述了一个综合的深度学习模型开发和验证流程,包括数据收集、图像处理、模型训练和多中心测试验证,旨在提高未知原发肿瘤诊断的准确性。
五、人工智能(AI)进行组织病理学图像分析,识别和区分子宫内膜癌(EC)的分子亚型


文献概述
这篇文章是一项关于使用人工智能(AI)进行组织病理学图像分析的研究,目的是识别和区分
子宫内膜癌(EC)的分子亚型。
研究团队运用AI技术对368名患者的组织病理学图像进行了分析,并在两个独立的验证队列中进一步验证了结果,这两个队列分别包含290名和614名来自其他中心的患者。
研究发现,通过AI分析能够区分p53异常(p53abn)和无特定分子特征(NSMP)的EC亚型,并且识别出一个NSMP EC患者亚群,这些患者的无进展生存期和疾病特异性生存率显著较差,被称为“p53abn样NSMP”。
研究还通过浅层全基因组测序发现,与普通NSMP相比,“p53abn样NSMP”群体的基因拷贝数异常负担更高,表明这一群体在生物学上与其他NSMP EC不同。该研究证明了AI在检测预后不同且传统分子或病理标准难以识别的EC亚群方面的强大能力,从而改进了基于图像的肿瘤分类。
此外,研究还指出,传统的临床病理参数在分类EC和指导治疗方面的可重复性不佳,特别是在高级别肿瘤中。AI的应用为识别具有侵袭性疾病的EC患者提供了新的工具,这些患者可能被过度治疗或未得到可能降低复发风险的适当治疗。研究结果表明,AI方法在区分p53abn和NSMP EC亚型方面具有较高的准确性和可靠性,并且可以推广到不同中心收集的数据集。
研究使用了多种深度学习模型以提高分类的准确性和鲁棒性,包括
- VarMIL
- Vanilla
- IDaRS
- HistogramBased
- Attention-based
- VLAD
- CLAM-MB
- CLAM-SB
- TransMIL
通过生存分析,研究还评估了不同亚群的疾病特异性生存期(DSS)和无进展生存期(PFS)。研究结果对于改善EC的诊断和治疗具有重要的临床意义,并且可以指导未来的临床试验和生物标志物的研究。
重点关注
图1展示了基于AI的组织病理学图像分析的工作流程,具体步骤如下:
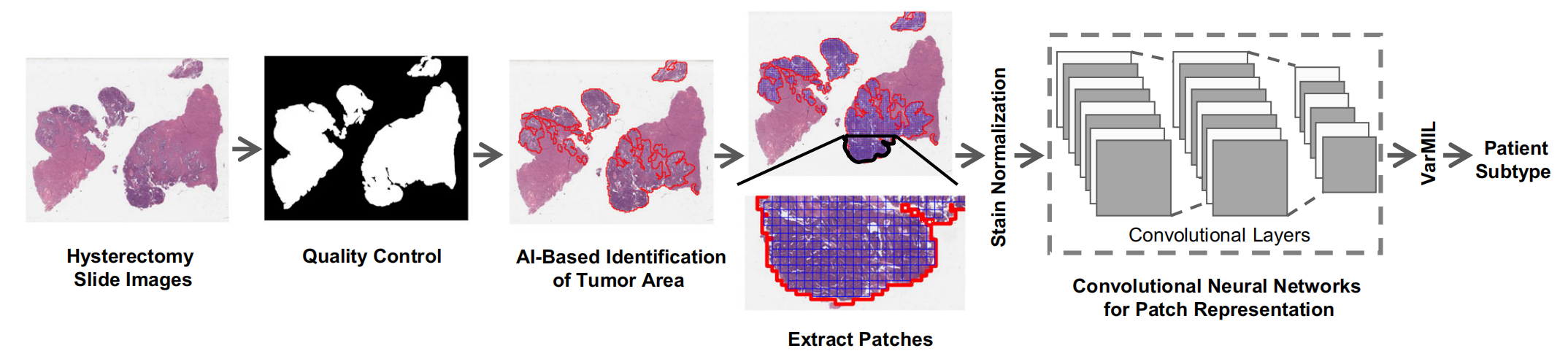
-
质量控制:首先使用名为HistoQC【81】的质量控制框架来生成一个掩膜,该掩膜仅包含组织区域,并去除图像中的伪影或异常。
-
肿瘤区域识别:接着,训练一个AI模型来识别组织病理学切片中的肿瘤区域。这个模型能够识别出哪些区域是肿瘤组织。
-
图像分割:识别出的肿瘤区域的图像被进一步分割成较小的块(patches),这些小块通常是512×512像素的大小。
-
颜色标准化:为了消除不同染色协议带来的颜色变化,对这些小块进行标准化处理,以确保颜色的一致性。
-
深度学习特征提取:将标准化后的图像小块输入到深度学习模型中,以提取每个小块的特征表示。这一步骤是深度卷积神经网络(CNN)发挥作用的地方,它能够学习图像块中与肿瘤分类相关的特征。
-
多实例学习模型预测:最后,使用基于多实例学习(Multiple Instance Learning, MIL)的模型VarMIL来预测患者的亚型。VarMIL模型能够综合考虑所有小块的特征,并对整个图像进行分类,从而预测患者的肿瘤亚型。
整个流程是一个从原始的组织病理学图像到肿瘤亚型预测的自动化过程,涉及到图像处理、深度学习和机器学习等多个步骤,旨在提高肿瘤分类的准确性和效率。
六、Prov-GigaPath:能够处理包含数十亿像素的全切片图像的全新数字病理基础模型

一作&通讯
| 作者类型 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Hanwen Xu | Microsoft Research | 微软研究院 |
| 第一作者 | Naoto Usuyama | University of Washington | 华盛顿大学 |
| 通讯作者 | Sheng Wang | University of Washington | 华盛顿大学 |
| 通讯作者 | Hoifung Poon | Microsoft Research | 微软研究院 |
文献概述
这篇文章是发表在《Nature》的一篇关于数字病理学领域的研究,介绍了一个名为Prov-GigaPath的全新数字病理基础模型,该模型在大规模真实世界数据上进行了预训练,能够处理包含数十亿像素的全切片图像。
主要内容包括:
-
模型介绍:Prov-GigaPath是一个在1.3亿个256×256病理图像瓦片上预训练的模型,这些图像来自美国大型医疗网络Providence的171,189个全切片,涵盖了超过30,000名患者的31种主要组织类型。
-
技术挑战:数字病理学面临的计算挑战包括处理大规模图像数据,以往的模型常常通过下采样来处理,但这样会丢失重要的全局上下文信息。
-
GigaPath架构:为了预训练Prov-GigaPath,作者提出了GigaPath,这是一种新的视觉变换器架构,适用于预训练千兆像素级别的病理切片。
-
性能评估:Prov-GigaPath在由Providence和TCGA数据组成的数字病理学基准测试中,包括9种癌症亚型任务和17种病理组学任务,表现出色,在26项任务中的25项上达到了最先进的性能。
-
多模态预训练:文章还探讨了Prov-GigaPath在病理报告的视觉-语言预训练方面的潜力,展示了其在标准视觉-语言建模任务中的性能。
-
开放性:Prov-GigaPath是一个开放权重的基础模型,包括源代码和预训练模型权重,以促进数字病理学研究的进展。
-
临床应用潜力:Prov-GigaPath在多种病理学任务上展示了其性能,表明了真实世界数据和全切片建模的重要性,有潜力协助临床诊断和决策支持。
文章还讨论了Prov-GigaPath在突变预测和癌症亚型预测方面的改进,以及在多模态视觉-语言处理方面的应用。作者强调了使用大规模预训练数据和大型模型架构的有效性,并提出了未来工作的方向,包括探索不同大小的模型架构、优化预训练过程以及将高级多模态学习框架整合到他们的工作中。
重点关注
图1提供了Prov-GigaPath模型架构的概览,具体内容可以分为以下几个部分:
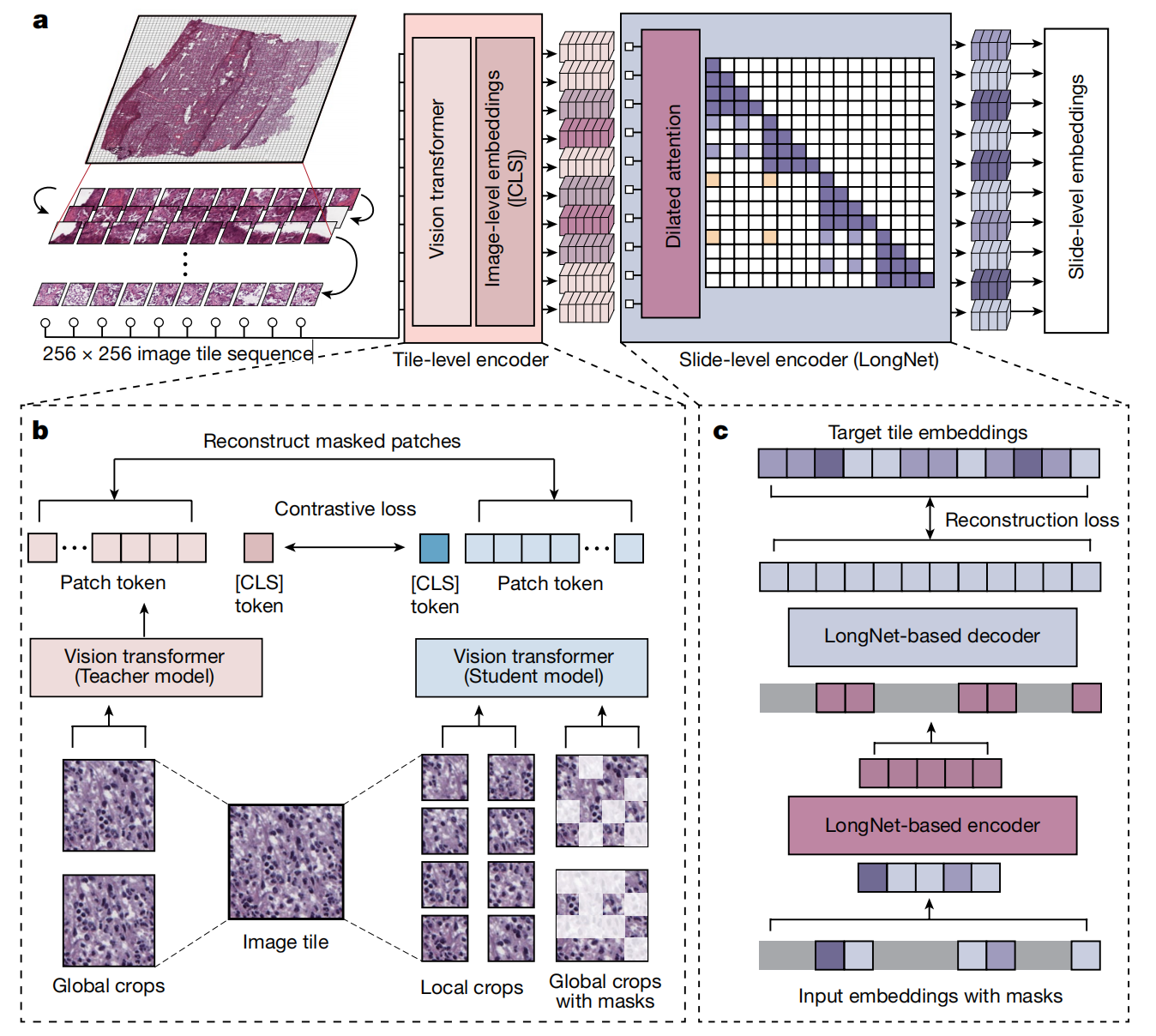
a. 模型架构流程图:
- Prov-GigaPath首先将每个输入的全切片图像(Whole Slide Image, WSI)序列化为按行主序排列的256×256像素图像瓦片序列。
- 使用图像瓦片级编码器(tile encoder)将每个图像瓦片转换成视觉嵌入(visual embedding)。
- 接着,Prov-GigaPath应用基于LongNet架构的切片级编码器(slide encoder),生成上下文化的嵌入(contextualized embeddings),这些嵌入可以作为各种下游应用的基础。
b. 图像瓦片级预训练:
- 使用DINOv2框架进行图像瓦片级的自监督预训练。DINOv2是一种先进的图像自监督学习框架,能够学习图像的特征表示。
c. 切片级预训练:
- 使用带有掩码的自编码器(masked autoencoder)和LongNet进行切片级的自监督预训练。LongNet是一种新开发的方法,适用于超长序列建模,能够适应数字病理学的需求。
[CLS]分类标记:
- 在模型中,[CLS]标记是分类(classification)的标记,通常用于Transformer模型中的序列分类任务。在训练过程中,这个标记会聚合整个序列的信息,以预测整个切片的标签。
总结:
Prov-GigaPath模型通过两阶段的预训练过程来学习图像瓦片和切片级别的特征。首先,它将全切片图像分割成小的图像瓦片,并学习每个瓦片的视觉特征。然后,它使用这些瓦片的特征来生成整个切片的上下文嵌入,这些嵌入可以捕捉到切片的全局模式和局部细节,为后续的病理分析任务提供支持。这种设计使得Prov-GigaPath能够有效地处理和分析大规模的病理图像数据,并在多种病理学任务中表现出色。
七、在晚期透明细胞肾细胞癌(aRCC)患者中,如何确定哪些患者可能从免疫检查点阻断(ICB)治疗中获益

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Lisa Kinget | 比利时鲁汶大学实验肿瘤学实验室 |
| 通讯作者 | Benoit Beuselinck | 比利时鲁汶大学大学医院鲁汶癌症研究所医学肿瘤科 |
| 通讯作者 | Abhishek D. Garg | 比利时鲁汶大学细胞应激与免疫实验室 |
文献概述
这篇文章的主要焦点是解决在晚期透明细胞肾细胞癌(aRCC)患者中,如何确定哪些患者可能从免疫检查点阻断(ICB)治疗中获益的问题。
研究团队进行了一项全面的多组学映射分析,涉及超过1000名患者的肿瘤组织,目的是在ICB治疗的背景下理解aRCC的免疫微环境。
研究的关键发现和方法包括:
-
多组学分析:研究者通过对aRCC患者的肿瘤组织进行多组学分析,发现了与免疫反应相关的特定基因和蛋白质的表达模式。
-
HLA分子的特异性:研究指出,具有对肿瘤新抗原具有高特异性的人类白细胞抗原(HLA)等位基因的患者,在接受ICB治疗后,其临床反应更为积极。
-
机器学习模型:研究者开发了一个基于机器学习的模型,用于从肿瘤的转录组数据中推导出一个新的HLA分子的表达特征,这个特征与ICB治疗后的阳性结果相关。
-
小鼠模型实验:使用RENCA肿瘤小鼠模型,研究者发现CD40激动剂与PD1阻断的联合使用可以增强肿瘤相关巨噬细胞和CD8+ T细胞的活性,从而实现最大的抗肿瘤效果。
-
空间映射技术:研究还利用空间转录组技术,对肿瘤微环境中的细胞相互作用进行了空间层面的分析,进一步证实了上述发现。
-
临床验证:研究结果在独立的临床队列中得到了验证,表明这些发现具有潜在的临床应用价值。
-
挑战与限制:尽管ICB治疗在aRCC中已获批准,但目前还没有公认的生物标志物能够合理地预先选择患者或指导“智能”免疫治疗组合。
-
研究意义:这项研究提供了新的多组学和空间地图,揭示了驱动ICB反应的免疫社区结构,有助于改善aRCC患者的临床管理。
文章的详细内容还包括了研究设计、实验方法、数据分析、以及对结果的深入讨论。研究结果不仅增进了对aRCC免疫微环境的理解,而且为开发新的生物标志物和治疗策略提供了科学依据。
重点关注
Fig. 1 提供了该研究的概览和Leuven RWD(真实世界数据)队列的分析结果。
a. 研究设计示意图:这部分描述了研究的发现阶段,研究者首先在aRCC(晚期透明细胞肾细胞癌)的Leuven RWD队列中进行了研究。随后,使用机器学习(ML)模型开发了一个特征标记(signature),并将其在外部(bulk)数据集以及单细胞和空间层面上进行了验证。此外,还通过小鼠RCC/RENCA模型进行了体内功能性验证。
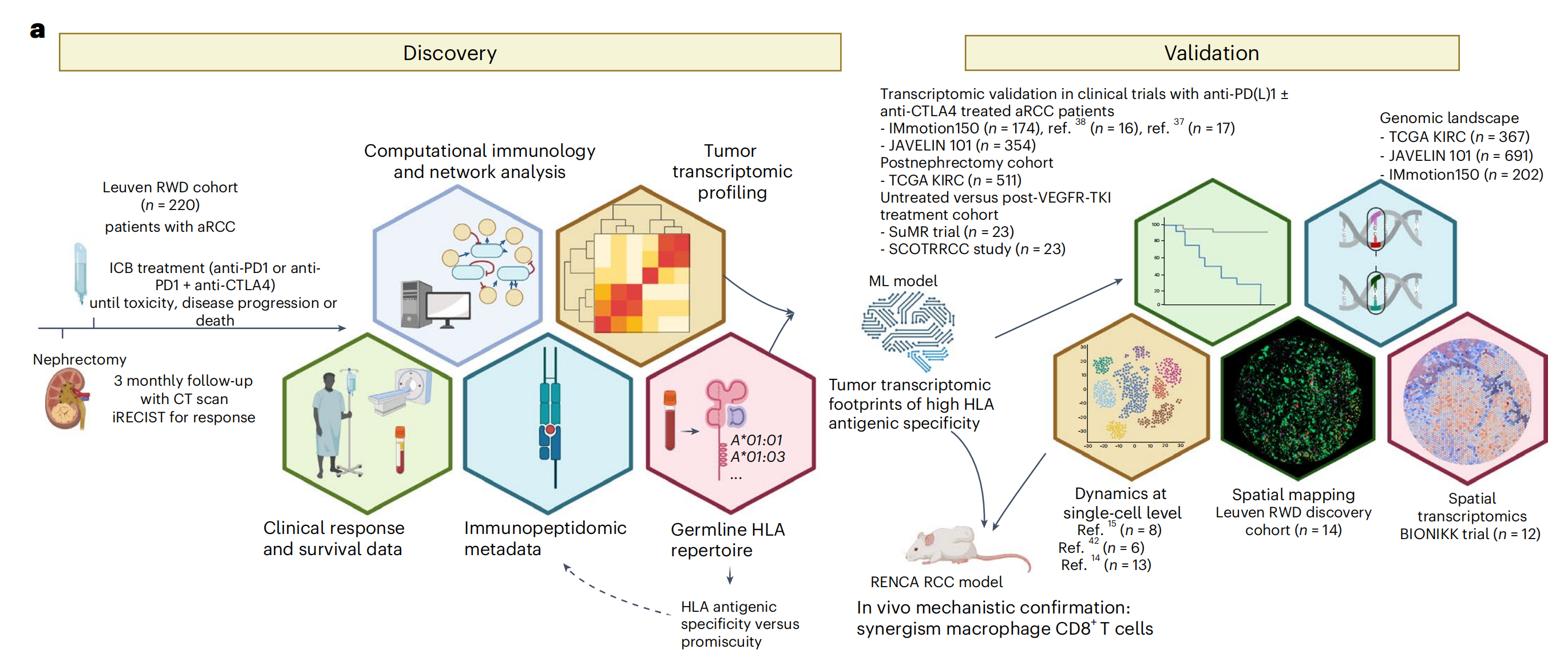
b. PFS的Kaplan-Meier曲线:这部分展示了Leuven RWD队列中,从开始接受ICB(免疫检查点阻断)治疗起,患者的无进展生存期(PFS)。根据ICB治疗的类型(ipilimumab-nivolumab组合治疗与单独nivolumab治疗)进行了分层。使用Cox比例风险回归模型计算了风险比(HR)和置信区间(CI),以比较两种治疗方案的效果。
c. OS的Kaplan-Meier曲线:与b部分类似,这部分展示了总生存期(OS)的Kaplan-Meier曲线,同样根据ICB治疗类型进行了分层,并使用Cox模型计算了HR和CI。
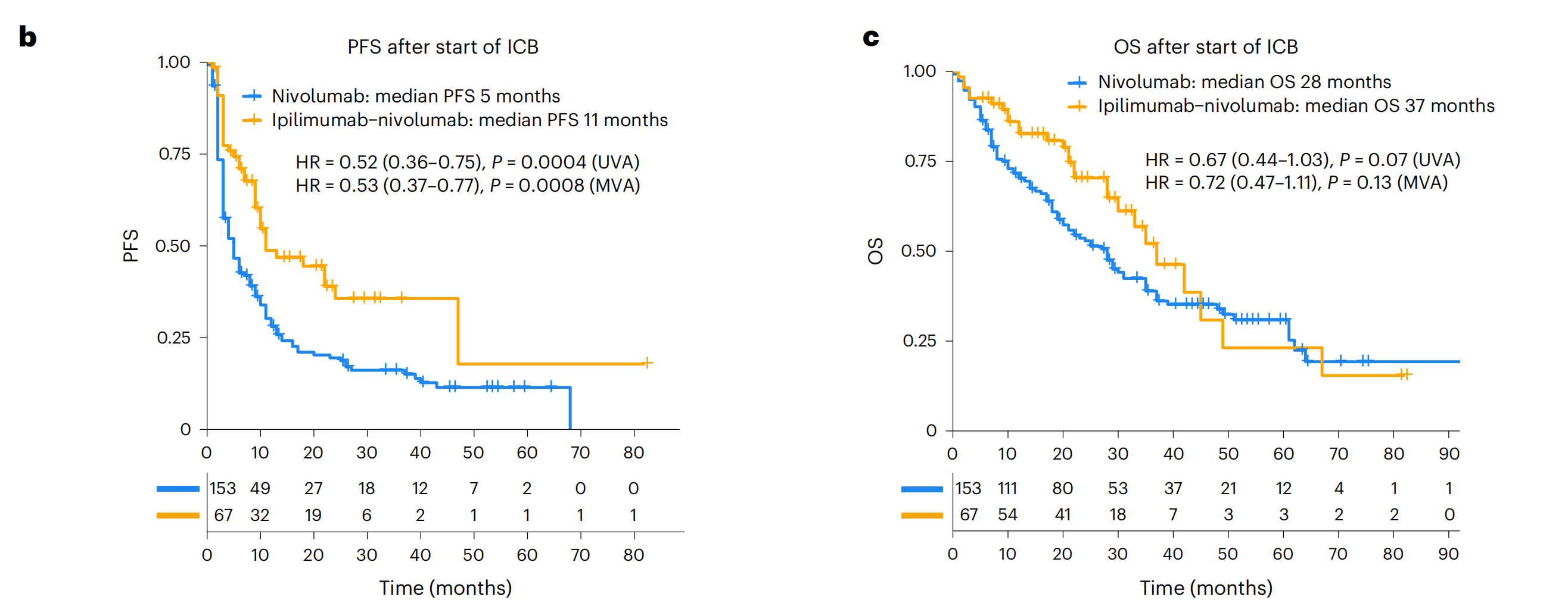
d. 最佳反应的堆叠条形图:这部分通过iRECIST标准(一种评估免疫治疗响应的标准),展示了ICB治疗后患者的最佳反应,包括完全缓解(CR)、部分缓解(PR)、疾病稳定(SD)、疾病进展(PD)和不可评估(NE)。使用Fisher精确检验计算了两种治疗方案间反应差异的P值。
e. 瀑布图:这部分通过iRECIST标准展示了每位患者肿瘤缩小的最大百分比。瀑布图是一种常用于展示肿瘤治疗反应的图表,可以直观地看出不同患者的治疗反应差异。
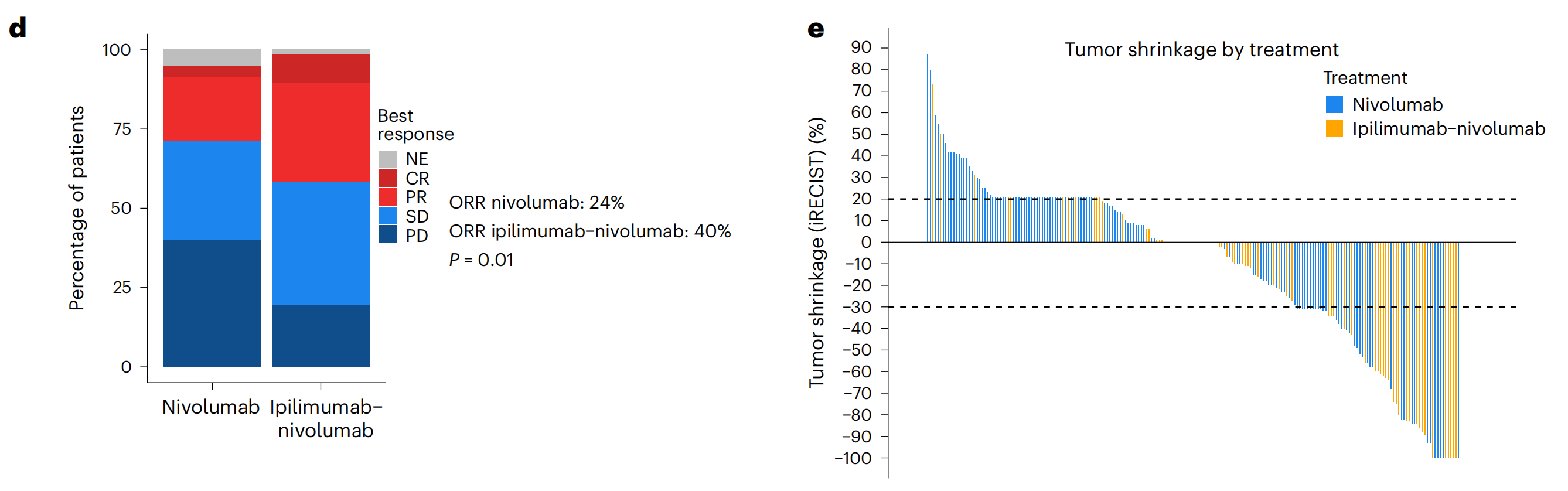
整体来看,Fig. 1 提供了关于ICB治疗在晚期透明细胞肾细胞癌患者中应用的全面分析,包括了研究设计、临床结果(PFS和OS)、治疗反应评估以及肿瘤缩小情况。通过这些数据,研究者能够评估不同ICB治疗方案的临床效果,并为未来的治疗策略提供科学依据。
八、一种名为组织学预后签名的新型数字组织学生物标志物,可提高侵袭性乳腺癌预后的能力

一作&通讯
| 角色 | 姓名 | 单位名称(英文) | 单位名称(中文) |
|---|---|---|---|
| 第一作者 | Mohamed Amgad | Department of Pathology, Northwestern University Feinberg School of Medicine, Chicago, IL, USA | 美国伊利诺伊州芝加哥市西北大学范伯格医学院病理学系 |
| 通讯作者之一 | Lee A. D. Cooper | Department of Pathology, Northwestern University Feinberg School of Medicine, Chicago, IL, USA | 美国伊利诺伊州芝加哥市西北大学范伯格医学院病理学系 |
| 通讯作者之二 | Lauren R. Teras | Department of Population Science, American Cancer Society, Atlanta, GA, USA | 美国癌症协会人口科学部,佐治亚州亚特兰大市 |
文献概述
这篇文章是发表在《Nature Medicine》上的一项研究,介绍了一种名为“组织学预后签名”(Histomic Prognostic Signature,简称HiPS)的新型数字组织学生物标志物,用于提高侵袭性乳腺癌预后的能力。
-
研究背景:乳腺癌是一种全球最常见的恶性肿瘤,具有高度异质性,其生存结果因肿瘤生物学、治疗方案和社会经济因素而有很大差异。传统的预后标准包括肿瘤-淋巴结-转移(TNM)分期、诺丁汉组织学分级和内在亚型。然而,这些标准主要依赖于病理学家的定性评估,并不包括肿瘤微环境中的非癌细胞元素。
-
HiPS开发:研究者利用深度学习技术开发了HiPS,这是一种全面的、可解释的评分系统,能够量化评估肿瘤微环境中的细胞和组织结构,测量上皮、基质、免疫和空间交互特征。HiPS的开发基于美国癌症协会的癌症预防研究-II(Cancer Prevention Study-II,CPS-II)的人群水平队列,并使用来自三个独立队列的数据进行验证,包括前列腺、肺、结直肠和卵巢癌试验(PLCO)、癌症预防研究-3(CPS-3)和癌症基因组图谱(The Cancer Genome Atlas,TCGA)。
-
研究方法:HiPS使用先进的深度学习基础的计算机视觉技术,对全切片图像(Whole-Slide Images,WSIs)进行定量评估,提供了一种客观的替代方法,减少了手动诺丁汉分级中的变异性,并捕捉到了无法可靠分级的潜在特征。HiPS还全面评估了整个肿瘤微环境(Tumor Microenvironment,TME),包括非肿瘤元素。
-
研究结果:HiPS在预测生存结果方面一致优于病理学家,且这一优势独立于肿瘤-节点-转移(TNM)分期和相关变量。HiPS的预后价值主要受基质和免疫特征的驱动。研究还发现,HiPS特征与高基因组不稳定性、缺氧免疫微环境、肌成纤维细胞(myCAF)表型和缺乏CD8+ T淋巴细胞的次优免疫反应相关。
-
结论:HiPS是一个经过稳健验证的生物标志物,可以支持病理学家的工作并改善患者的预后。HiPS的透明性和可解释性是其关键优势,它由对应于可识别的、已建立的生物学实体的特征组成。
-
研究意义:这项研究展示了如何利用人工智能系统提高乳腺癌非转移性侵袭性癌症患者的预后,为未来的癌症治疗和研究提供了新的方向和工具。
重点关注
图 4 展示了基质特征对 HiPS 分数的重要影响以及在 I 期癌症中如何改变风险分类。
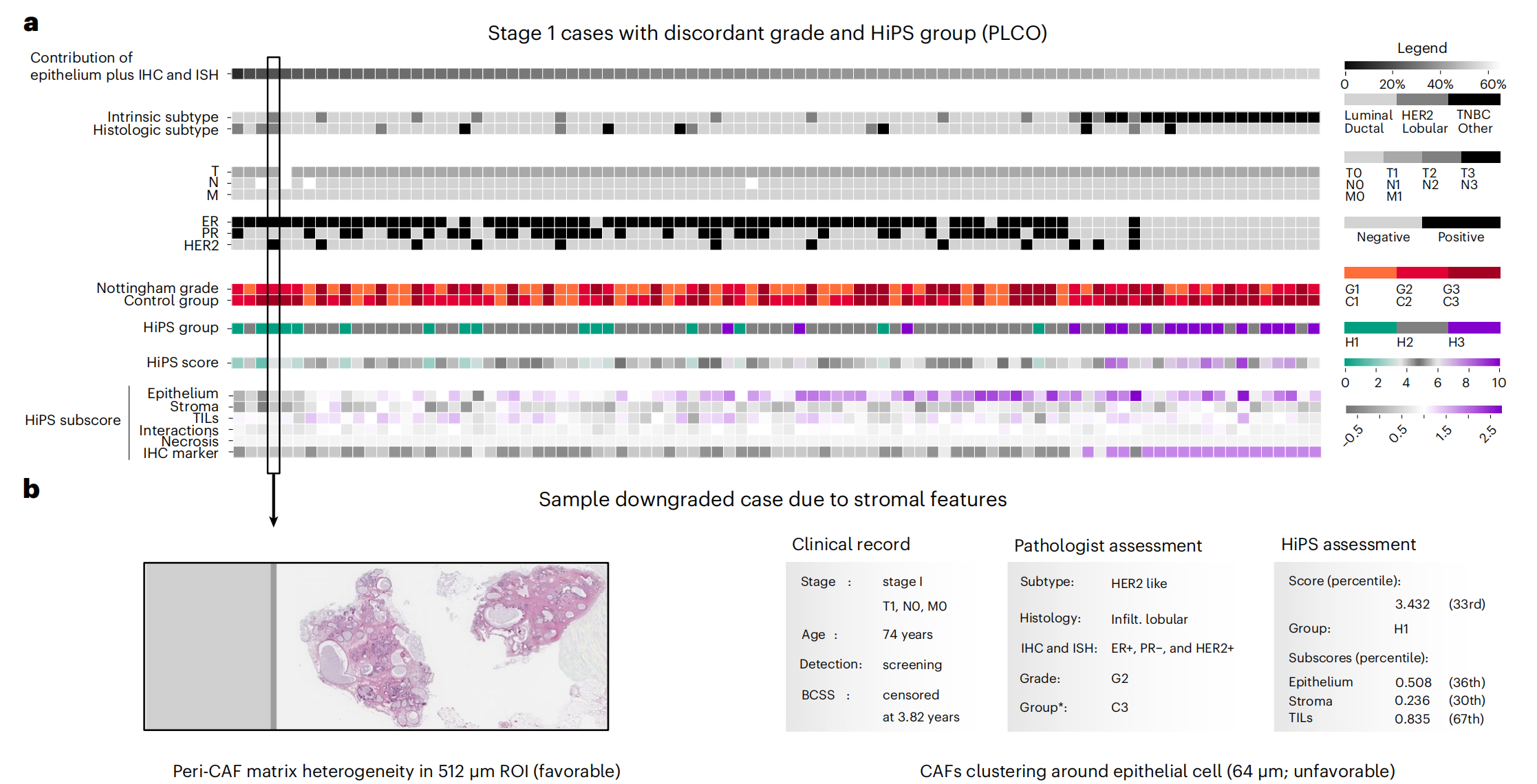
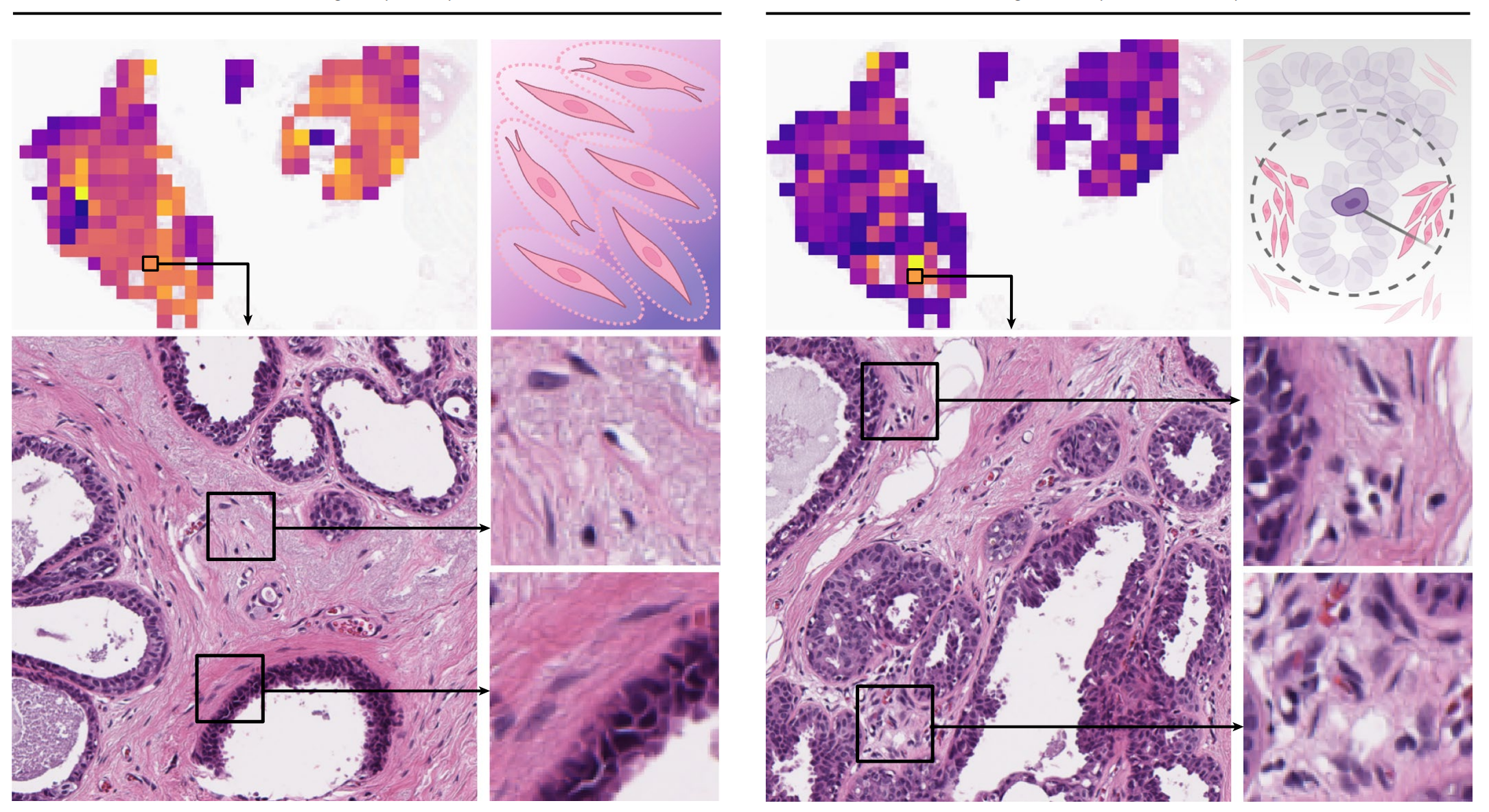
图 4a 分析:
- 研究对象:91 名 I 期癌症患者(共 231 名 I 期癌症患者)。
- HiPS 对风险分类的影响:HiPS 改变了诺丁汉风险分类,这表明在 I 期癌症中,组织学特征在指导临床决策中的重要性更高。
- HiPS 分数的组成:患者根据上皮和 ER、PR、HER2 特征对 HiPS 分数的贡献百分比进行排序。HiPS 分数由六个不同的生物学主题的子分数组成,这些子分数总结了每个主题内特征的影响。六个子分数的总和等于总 HiPS 分数。
- 非上皮特征的作用:在该队列中,非上皮特征对总 HiPS 分数的贡献很大,并且在改变患者的风险类别方面具有重要影响。
图 4b 分析(样本案例):
- 基质特征的重大影响:展示了基质特征对 HiPS 分数具有重大影响的两个例子:
- CAF 周围基质基质的异质性:反映了基质界面变化,如间质性变化(desmoplasia),这是一个有利的预后特征。
- CAF 在上皮细胞 64 微米半径内的聚集:这是一个不利的预后特征,因为它表明了基质细胞在肿瘤周围的密集分布,可能与肿瘤的侵袭性和预后不良相关。
总结:
图 4 强调了基质特征在 HiPS 分数中的重要性,尤其是在 I 期癌症中。基质特征不仅包括了肿瘤微环境中的细胞成分,还包括了非细胞成分,如基质基质和胶原蛋白。这些特征对于评估患者的预后和风险分类具有显著的影响,提示了在癌症治疗和研究中需要更多地关注肿瘤微环境的复杂性。
九、基于人工智能和MRI特征的非侵入性方法,预测小脑髓母细胞瘤的分子亚群

一作&通讯
| 角色 | 姓名 | 单位名称(中文) | 单位名称(英文) |
|---|---|---|---|
| 第一作者 | 王彦然 (Yan-Ran) | 安徽省生物医学成像与智能处理重点实验室,合肥综合性国家科学中心人工智能研究所,中国 | Anhui Province Key Laboratory of Biomedical Imaging and Intelligent Processing, Institute of Artificial Intelligence, Hefei Comprehensive National Science Center, China |
| 通讯作者1 | 龚建 | 北京天坛医院,首都医科大学附属北京神经外科研究所,中国 | Department of Pediatric Neurosurgery, Beijing Tiantan Hospital, Capital Medicine University, Beijing Neurosurgical Institute, China |
| 通讯作者2 | 龚建 (Jian Gong) | 斯坦福大学医学院,美国 | Department of Neurosurgery, Stanford School of Medicine, Stanford University, USA |
文献概述
这篇文章报道了一种基于人工智能和MRI特征的非侵入性方法,能够预测小脑髓母细胞瘤的分子亚群,为全球范围内的低资源地区提供了一种低成本、术前诊断的新途径。
-
研究背景:Medulloblastoma (MB) 是儿童和青少年中最常见的恶性中枢神经系统肿瘤,分子亚群测试是分层风险的唯一验证手段。但由于测试的复杂性和成本,全球许多医疗中心,特别是资源有限地区,难以进行这种测试。
-
研究目的:建立一个国际性的分子特征数据库,包含来自中国和美国的13个中心的934名MB患者,旨在通过基于MRI数据的机器学习策略,为MB的临床管理提供一种非侵入性、术前、低成本的分子亚群预测的替代途径。
-
研究方法:利用机器学习工作流程,从患者大脑MRI扫描中自动分割肿瘤区域,然后通过手动验证确保准确性。提取定量的放射组学特征和定性的肿瘤特征,以通知机器学习分类器。
-
研究结果:
- 建立了最大的分子特征化的MB数据库。
- 通过3折交叉验证,三类分类器(区分WNT、SHH和非WNT/非SHH,即G3/G4)显示出高AUC值。
- 在外部验证集中,AI模型能够泛化到不同的患者群体,包括在模型训练期间未涉及的医疗中心和种族。
- 通过前瞻性建立的独立测试集,进一步验证了AI模型在真实世界临床环境中的性能。
-
特征重要性分析:通过Shapley分析,研究了每个输入特征对机器学习模型预测准确性的贡献。发现肿瘤内特征对预测肿瘤分子亚群最为重要。
-
肿瘤位置与分子亚群的关联:研究发现肿瘤位置与分子亚群之间存在高度关联,这一发现与以往基于有限样本量的研究相矛盾。
-
研究意义:该研究展示了基于MRI的AI技术在MB分子诊断中的潜力,有助于减少全球健康差异,提高风险分层,并加速个性化治疗,特别是在资源受限地区。
-
数据和代码的可用性:研究提供了数据库和MRI成像特征的公开访问,以及用于肿瘤分割的深度学习模型的代码。
文章强调了通过AI和MRI特征实现术前分子亚群预测的可能性,并为全球MB研究提供了宝贵的资源。
重点关注
Figure 1展示了该研究的方法论概览和全球小脑髓母细胞瘤数据集的组装情况。
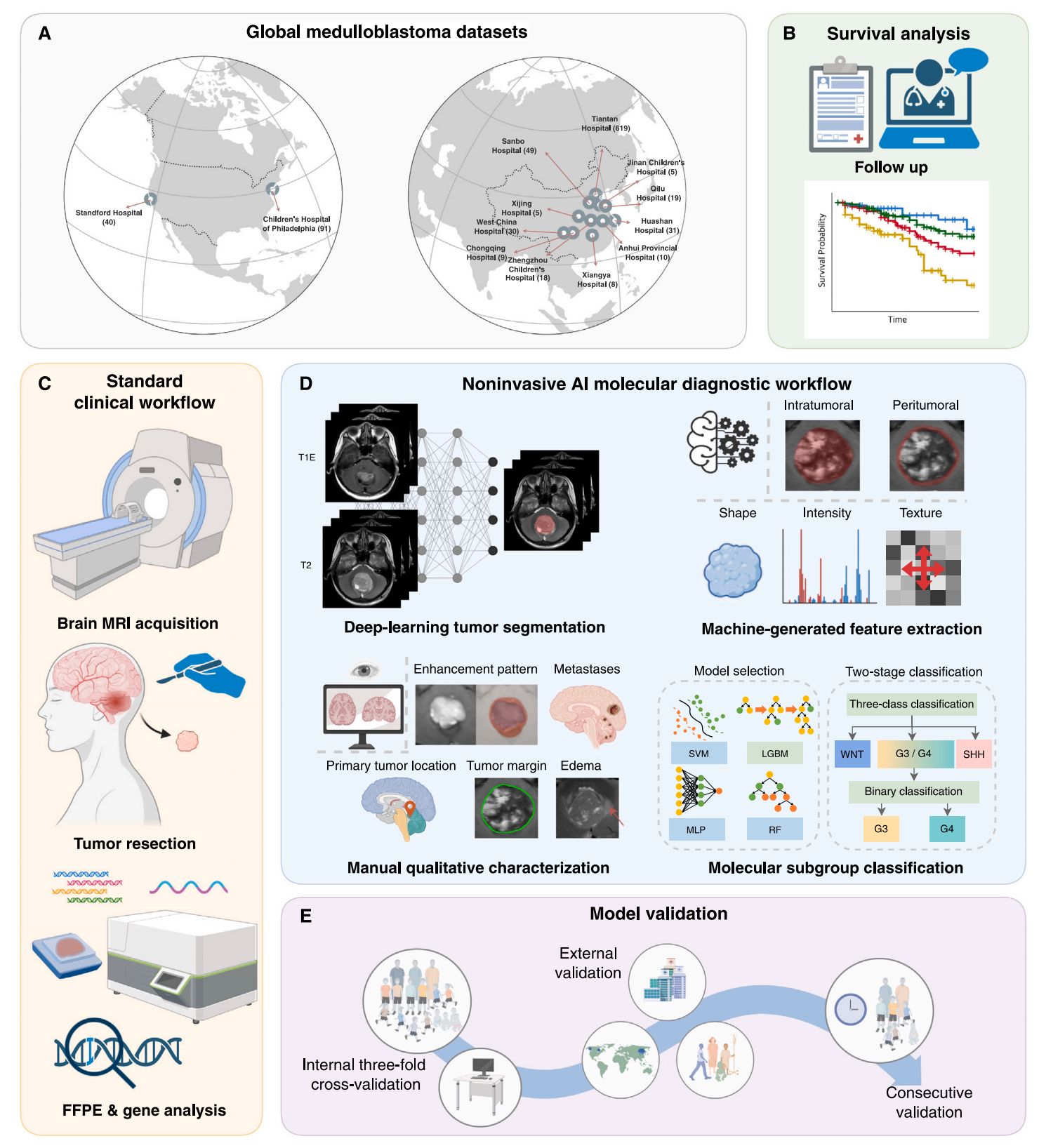
Figure 1A:
- 这部分提供了一个视觉化的全球小脑髓母细胞瘤数据集的组成部分,通过一张地图展示了参与组装这个全球数据集的中国和美国的城市。
- 地图上标注的城市包括斯坦福(Stanford)、费城(Philadelphia)、上海(Shanghai)、北京(Beijing)、济南(Jinan)、西安(Xi’an)、重庆(Chongqing)、成都(Chengdu)、郑州(Zhengzhou)、合肥(Hefei)和长沙(Changsha)。
- 这些城市代表的医疗中心为建立包含934名小脑髓母细胞瘤患者的国际分子特征数据库做出了贡献。
Figure 1B:
- 该部分说明了临床数据和预后信息是如何通过电话调查收集的。
- 这些数据作为本研究生存分析的主要终点。
Figure 1C:
- 这部分描述了小脑髓母细胞瘤的传统标准分子诊断工作流程。
- 这可能涉及到从手术后的肿瘤组织中进行RNA测序或DNA甲基化分析,以确定肿瘤的分子亚群。
整体来看,Figure 1强调了这项研究的全球合作性质,以及通过多中心合作收集的大量数据如何支持了该研究的深度和广度。通过这种方式,研究团队能够利用来自不同地区和种族背景的患者的MRI数据,开发和验证了一种基于人工智能的非侵入性分子亚群预测模型。
十、通用自监督病理模型:UNI

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Richard J. Chen | 哈佛医学院布里格姆和妇女医院病理科 |
| 第一作者 | Tong Ding | 哈佛医学院工程与应用科学学院 |
| 第一作者 | Ming Y. Lu | 哈佛医学院和麻省理工学院Broad Institute癌症项目 |
| 通讯作者 | Faisal Mahmood | 哈佛医学院布里格姆和妇女医院病理科 |
文献概述
这篇文章文章提出了一个名为UNI的通用自监督病理模型,该模型在超过100万张诊断级别的组织染色全切片图像(WSIs)上进行了预训练,涵盖了20种主要的组织类型。
UNI模型在34个不同的计算病理学(CPath)任务上进行了评估,包括肿瘤检测、分级和分类等,显示出在多个任务上超越了以往的最先进模型。
关键点概括如下:
-
背景:在计算病理学中,对组织图像的定量评估是至关重要的,但由于全切片图像(WSIs)的高分辨率和形态学特征的变异性,大规模注释数据存在挑战。
-
UNI模型:为了应对这一挑战,研究者提出了UNI,这是一个使用超过100 million图像进行预训练的自监督模型,这些图像来自超过100,000个诊断级别的H&E染色WSIs,数据量超过77TB。
-
评估:UNI在34个具有代表性的CPath任务上进行了评估,这些任务在诊断难度上各不相同。
-
性能:UNI不仅在多个任务上超越了先前的最先进模型,还展示了在CPath中新的建模能力,例如分辨率不可知的组织分类、使用少量类别原型的幻灯片分类,以及在OncoTree分类系统中对多达108种癌症类型的疾病亚型泛化。
-
临床意义:UNI通过大规模的无监督表示学习,推进了CPath领域的发展,使得人工智能模型能够高效地泛化和转移到具有挑战性的诊断任务和临床工作流程中。
-
数据集和方法:文章详细描述了用于评估UNI的数据集,包括OncoTree癌症分类系统、弱监督幻灯片分类任务等。同时提供了实验设置、实现细节和性能的补充信息。
-
结论:UNI作为一个基础模型,展示了其在多种临床任务中的潜力,特别是在分类罕见和代表性不足的疾病方面。
文章还包括了对模型和数据规模、自监督学习算法选择、以及模型泛化能力的深入分析,以及对UNI在不同图像分辨率下的鲁棒性的评估。研究者还讨论了UNI在实际临床应用中的潜在影响,以及未来在多模态能力和幻灯片级别模型开发方面的工作。
重点关注
图 4 描述了在少量样本(Few-shot)情况下,基于类原型(class prototypes)的感兴趣区域(ROI)和幻灯片级别的分类方法。
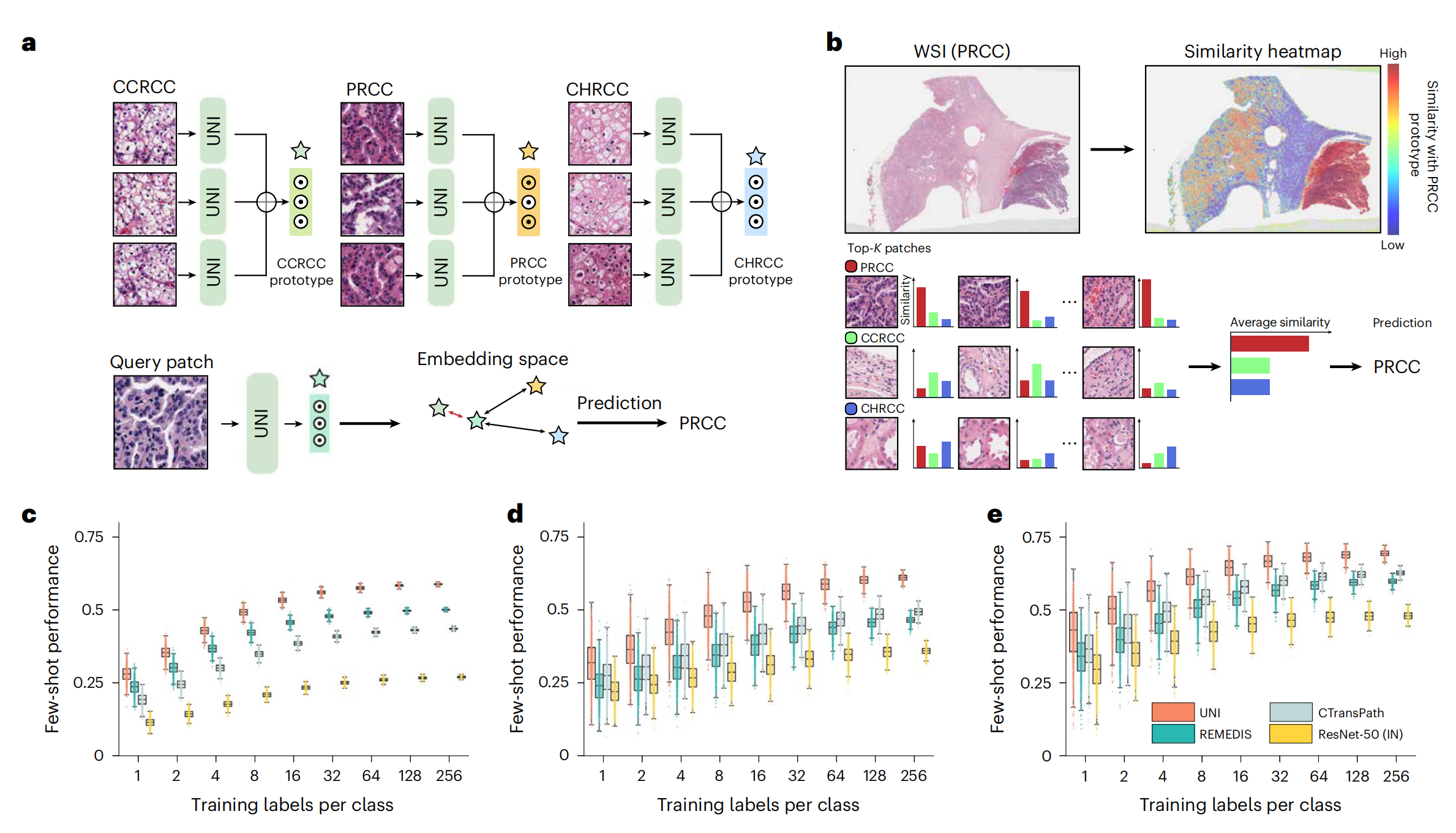
a. 通过 SimpleShot 进行的少量样本 ROI 分类:
- 通过平均同一类别的 ROI 提取的特征来构建类原型。
- 对于测试 ROI,SimpleShot 根据最小的欧几里得距离(即最相似的类原型)来预测其类别。
b. 通过 MI-SimpleShot 进行的少量样本幻灯片分类:
- 使用预先计算的 ROI 级别类原型集合(与幻灯片共享相同的类别标签)。
- MI-SimpleShot 根据来自 WSI 的 top-K 查询补丁与真实类别原型之间的平均相似性最高的类原型来预测幻灯片标签。
- 相似性热图显示了真实类别原型与 WSI 中每个补丁之间的相似性。
c-e. SimpleShot 在三个任务上的少量样本 ROI 分类性能:
- 展示了三个任务的模型性能四分位数(n = 1,000 次运行)和延伸至四分位数范围 1.5 倍内的数据点。
- 任务包括:
- c. 泛癌组织分类(TCGA, n = 55,360 个 ROIs)。
- d. 结直肠癌息肉分类(UniToPatho, n = 2,399 个 ROIs)。
- e. 前列腺癌组织分类(AGGC, n = 345,021 个 ROIs)。
- 所有任务的少量样本 ROI 性能在扩展数据图 8 中提供。
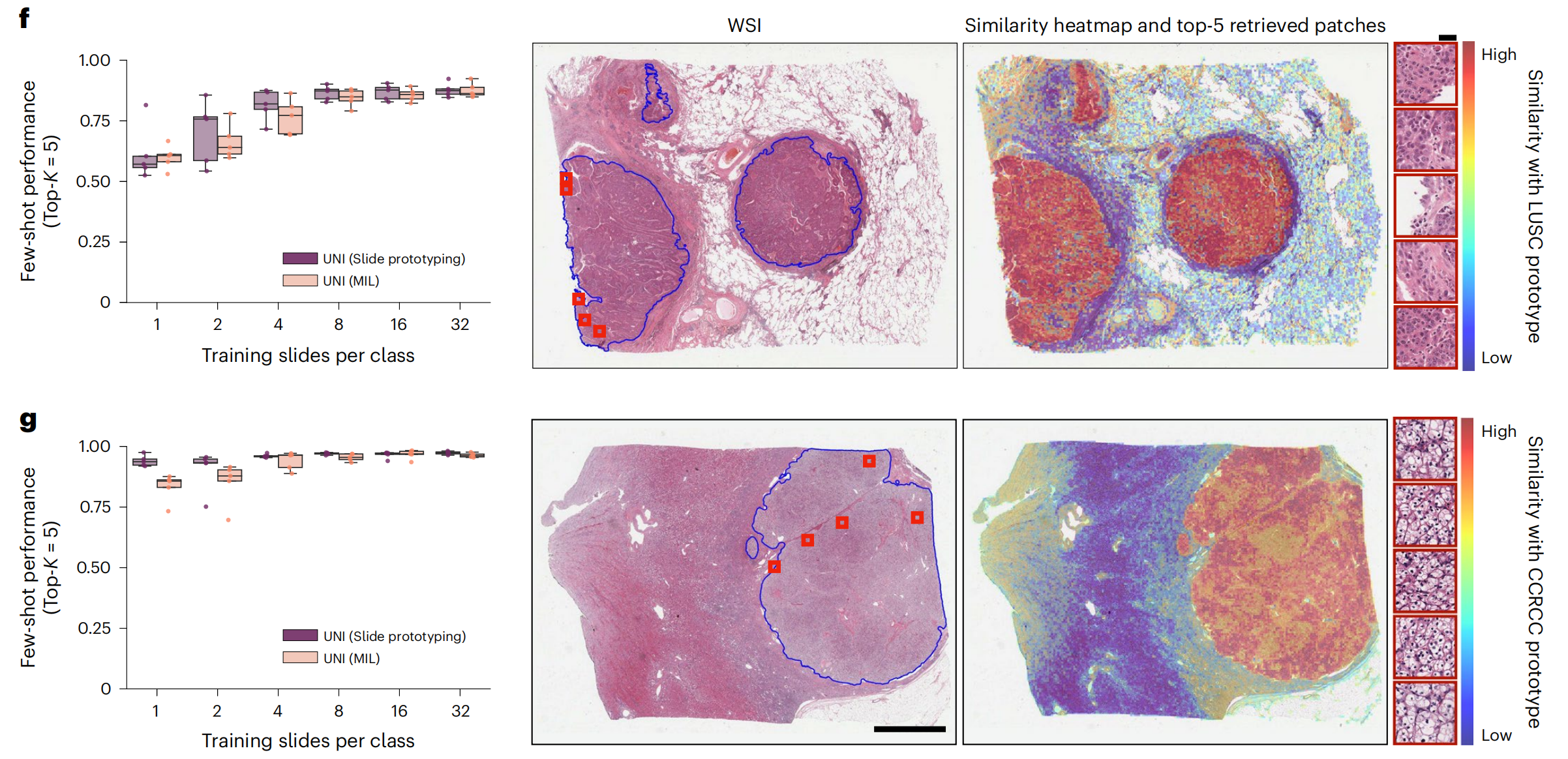
f,g. 通过 MI-SimpleShot 进行的少量样本幻灯片分类性能和相似性热图:
- f. 非小细胞肺癌(NSCLC)亚型分类(训练数据:TCGA;测试数据:CPTAC;n = 1,091 张幻灯片)。
- g. 肾细胞癌(RCC)亚型分类(训练数据:TCGA;测试数据:CPTAC-DHMC;n = 872 张幻灯片)。
- 在这两项任务中,使用 UNI 提取的预先特征,比较 MI-SimpleShot 与 ABMIL 在相同少量样本设置中的性能。
- 展示了与真实类别原型相似性最高的 top-5 补丁(用红色边框表示)的相似性热图,针对 LUSC(f)和 CCRCC(g)幻灯片。
- 尺度条:WSI 为 2 毫米;检索到的 top-5 补丁为 56 微米。
- 方法和扩展数据图 8-10 中提供了更多细节、比较和可视化。
整体而言,图 4 展示了 UNI 模型在少量样本学习方面的高效性,特别是在 ROI 和幻灯片分类任务中,通过构建类原型并利用相似性度量进行分类,展现了 UNI 在病理学图像分析中的潜力。























 1709
1709

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








