小罗碎碎念
这篇推文的主题是虚拟染色——包含【配对】和【无配对】两种方式,共计5篇文献。注意,前两篇是【配对】的,后三篇是【无配对】的,不要弄混啦。
另外,我觉得部分老师/同学看到“配对”这俩字是有点懵的,看下面这张图就明白了。

我们如果想实现HE到IHC的虚拟染色,那么早期的做法是准备两两配对的HE和IHC图像,最新的模型已经可以使用不是一一对应的HE和IHC图像了,直接学习底层的染色模式。(这样解释一下,应该就都清楚了,如果还是有点晕,看完后续的推文也就懂啦)
一、从H&E到特殊染色:AI改善非肿瘤性肾脏疾病诊断

一作&通讯
| 作者角色 | 姓名 | 单位名称 | 单位地址 |
|---|---|---|---|
| 第一作者 | Kevin de Haan | 加利福尼亚大学洛杉矶分校电气与计算机工程系 | 美国加利福尼亚州洛杉矶 |
| 加利福尼亚大学洛杉矶分校生物工程系 | |||
| 加利福尼亚纳米系统研究所 | |||
| 通讯作者 | Yair Rivenson | 加利福尼亚大学洛杉矶分校电子与计算机工程系 | |
| 加利福尼亚大学洛杉矶分校生物工程系 | |||
| 加利福尼亚纳米系统研究所 | |||
| W. Dean Wallace | 南加州大学凯克医学院病理学与实验室医学系 | 美国加利福尼亚州洛杉矶 | |
| Aydogan Ozcan | 加利福尼亚大学洛杉矶分校电子与计算机工程系 | ||
| 加利福尼亚大学洛杉矶分校生物工程系 | |||
| 加利福尼亚纳米系统研究所 | |||
| 加利福尼亚大学洛杉矶分校医学院外科 |
文献概述
该研究利用深度学习技术将常规的苏木精-伊红(H&E)染色组织切片转换成特殊的染色效果,以提高非肿瘤性肾脏疾病的诊断质量。
研究团队通过监督学习的方法,训练了一个基于深度学习的计算染色转换框架,将H&E染色转换为Masson’s Trichrome(MT)、周期酸-席夫(PAS)和Jones银染(JMS)等特殊染色。
这项技术在58个独立样本的肾脏活检组织切片上进行了测试,并通过三位肾脏病理学家的评估,发现使用虚拟特殊染色图像可以改善几种非肿瘤性肾脏疾病的诊断(P = 0.0095)。另一项研究发现,通过计算生成的特殊染色的质量在统计上与组织化学染色相当。
研究还展示了这种染色转换框架在初步诊断中的价值,尤其是在需要额外特殊染色时,可以节省时间和成本。此外,该技术还提供了额外的优势,如改善染色一致性、减少染色准备时间,并且可以避免对标本的破坏性额外切片和染色过程,从而节省资源并减少对患者的负担。
文章详细介绍了深度神经网络的设计和训练过程,包括使用CycleGAN进行数据增强,以及如何通过生成对抗网络(GAN)进行训练。
此外,还讨论了该技术在实际应用中的潜在优势,包括减少化学处理、节省时间和实验室成本,以及在不同实验室和设备间的良好泛化能力。
研究还指出,尽管该技术在当前研究中已经显示出潜力,但未来的研究需要更大的训练和测试数据集来进一步验证其适用性。
最后,文章还提供了一些实施细节,包括图像数据的获取、病理评估、统计分析方法以及代码和数据的可用性。
简要分析
Fig. 1 提供了一种基于深度学习的H&E染色向特殊染色转换技术的概览。在这个过程中,常规的苏木精-伊红(H&E)染色的组织切片通过深度神经网络被数字化转换成三种不同的特殊染色:Jones银染(JMS)、Masson三色染色(MT)和周期酸-席夫染色(PAS)。
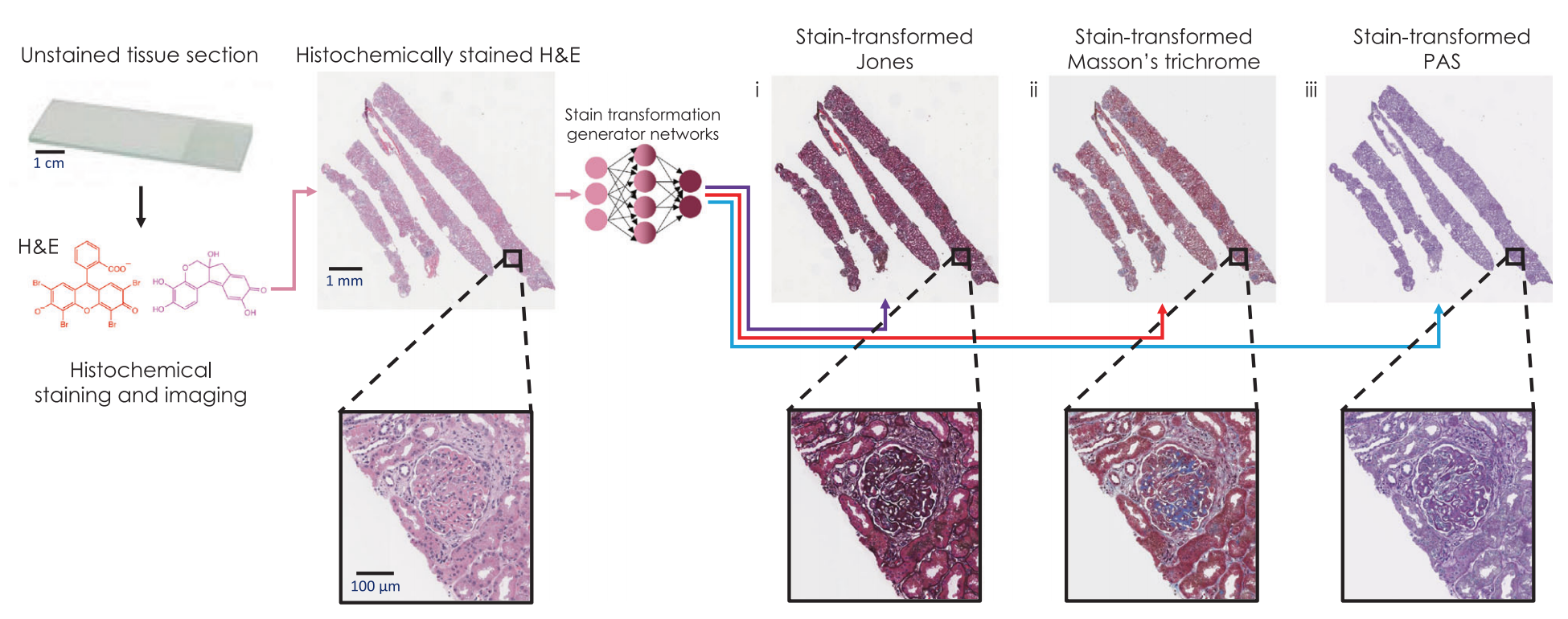
以下是对图1中描述的转换过程的分析:
-
起始点:组织切片首先经过H&E染色,这是一种常规的染色方法,用于突出细胞核(蓝色)和细胞质(红色)。
-
深度神经网络:使用一个深度学习模型,具体来说是一个生成对抗网络(GAN),来学习H&E染色图像和特殊染色图像之间的转换关系。
-
特殊染色的生成:
- JMS(Jones银染):通过深度神经网络的转换,H&E染色图像被转换成JMS染色图像。JMS染色主要用于突出肾脏和其他组织的基底膜,以及识别肾小球的形态结构。图中用紫色箭头(purple arrow)表示这一转换过程。
- MT(Masson三色染色):H&E染色图像也被转换成MT染色图像。MT染色用于观察结缔组织,特别是胶原蛋白。图中用红色箭头(red arrow)表示这一转换过程。
- PAS(周期酸-席夫染色):此外,H&E染色图像还能转换成PAS染色图像。PAS染色用于高亮显示多糖和粘液,常用于分析肾脏病变。图中用蓝色箭头(blue arrow)表示这一转换过程。
-
转换效果:通过这种深度学习技术,可以生成与实际经过特殊染色过程处理的组织切片图像在视觉上相似的虚拟染色图像,从而为病理学家提供更多的诊断信息。
-
应用价值:这种技术的应用可以减少实际特殊染色所需的时间和成本,同时加快病理诊断的过程,特别是在需要快速做出诊断和治疗决策的临床情况下。
总的来说,Fig. 1 展示了一个创新的深度学习框架,它能够将常规的H&E染色图像转换成多种特殊染色图像,为病理诊断提供了一种高效、低成本的新方法。
二、乳腺癌免疫组化图像合成:金字塔Pix2pix模型的应用

一作&通讯
| 姓名 | 角色 | 单位 |
|---|---|---|
| Shengjie Liu | 第一作者 | 北京邮电大学 |
| Chuang Zhu | 通讯作者 | 北京邮电大学 |
| Feng Xu | 通讯作者 | 首都医科大学 |
| Xinyu Jia | 作者 | 北京邮电大学 |
| Zhongyue Shi | 作者 | 首都医科大学 |
| Mulan Jin | 作者 | 首都医科大学 |
文献概述
这篇文章介绍了一种新的乳腺癌免疫组化图像生成方法,通过金字塔Pix2pix模型来实现。
研究团队首先提出了一个乳腺癌免疫组化(BCI)基准数据集,该数据集包含4870对配对的苏木精-伊红(HE)染色图像和免疫组化(IHC)图像,涵盖了不同HER2表达水平。这个数据集旨在通过图像到图像的翻译技术,直接从HE染色的全切片图像(WSI)合成IHC数据,以减少HER2评估的成本。
文章的主要贡献包括:
- 收集并构建了BCI数据集,这是第一个大规模公开可用的免疫组化图像生成数据集。
- 提出了一种基于HE的免疫组化图像生成的金字塔Pix2pix方法,与其他Pix2pix类似方法相比,该方法可以在多尺度上约束生成的图像,并在BCI数据集上取得更好的结果。
- 在BCI和LLVIP数据集上进行了广泛的实验,探索了不同尺度对模型的增益,展示了多尺度约束的灵活性和通用性。
研究还涉及了图像翻译算法的相关工作,包括无监督和监督的图像翻译方法,以及在图像翻译任务中常用的数据集。此外,文章还详细介绍了BCI数据集的建立过程,包括组织收集、切片准备、图像配准和后处理。
在提出的金字塔Pix2pix模型中,作者采用了多尺度损失函数,以适应结构对齐的数据,并在不同尺度上对生成的图像和真实图像进行比较,以提高生成图像的质量和准确性。
最后,文章通过与其他图像翻译算法的比较,展示了所提出方法的有效性,并进行了主观验证,邀请病理学家对生成的图像进行HER2表达评估。尽管当前方法在临床应用前还有很长的路要走,但BCI数据集对于进一步研究的重要性不言而喻。
未来的工作将探索合成IHC图像与真实IHC图像在HER2评估上的差异,并研究使用合成IHC图像为乳腺癌制定准确临床治疗方案的可能性。
简要分析
下图描述了建立BCI数据集的三个主要步骤
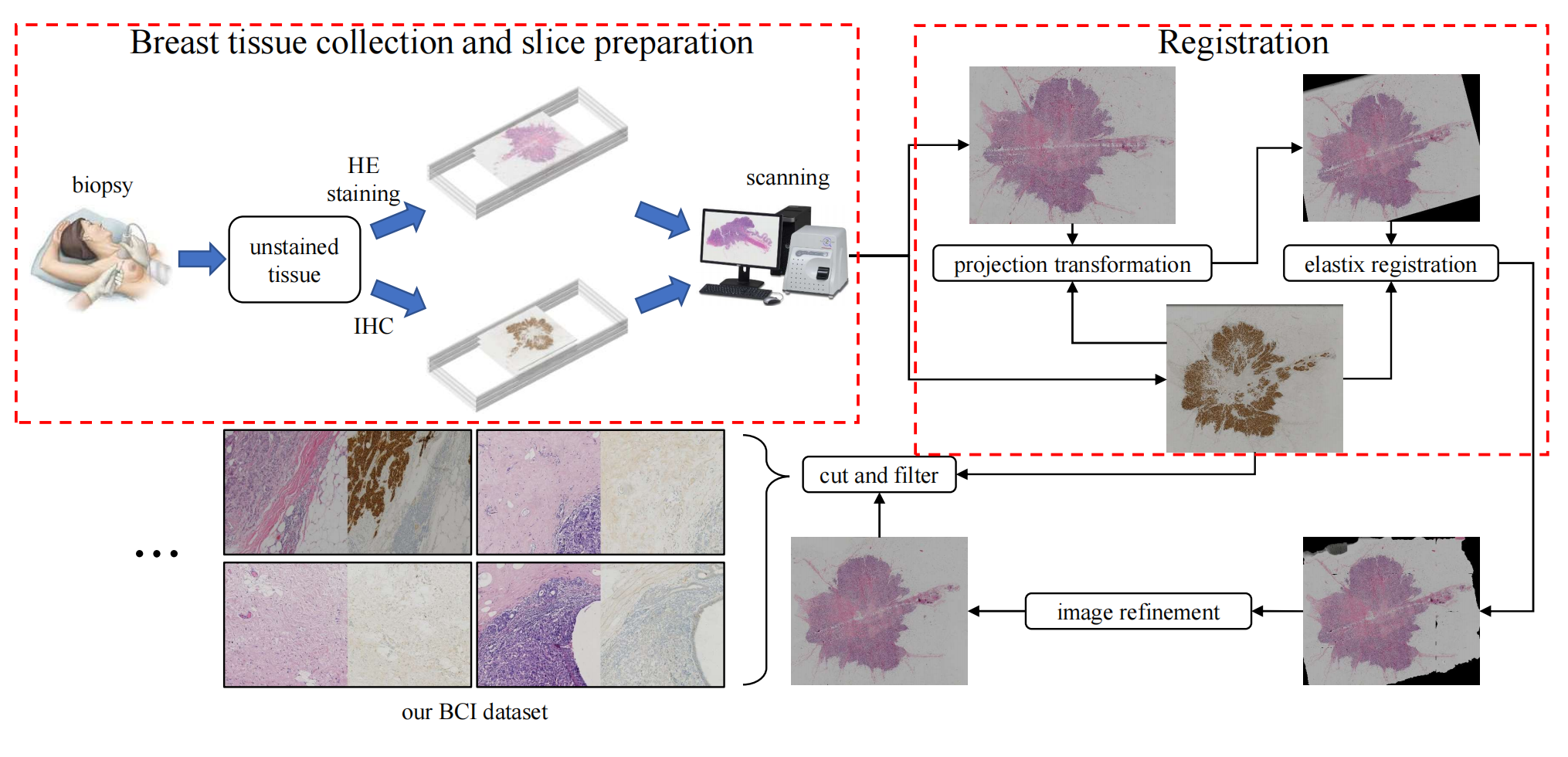
-
乳腺组织收集和切片准备:
- 这一步涉及实际的病理切片制作过程。首先,需要收集乳腺癌组织样本。
- 然后,这些组织样本被切成薄片,用于制作苏木精-伊红(HE)染色切片和免疫组化(IHC)染色切片。
- 这些切片是后续图像生成和分析的基础。
-
两个领域的图像配准:
- 由于IHC染色和HE染色的组织切片可能在形态上存在差异,且在切片制备过程中组织样本可能会被拉伸或压缩,因此需要对这两类图像进行配准处理。
- 图像配准的目的是使两个领域的图像在结构上对齐,以便进行准确的比较和分析。
- 配准过程包括初步的投影变换和更精细的非刚性区域配准。初步配准通过人工选择对应点并进行投影映射来实现大致对齐。然后,使用elastix工具箱进行更精细的局部非刚性配准,以尽可能地对齐两个领域图像的细节。
-
后处理,包括图像细化和切块:
- 在图像配准过程中,图像的扩张和收缩可能会在每个图像块的边缘留下间隙。因此,需要去除这些黑色边框,并用周围内容填充。
- 配准后的全切片图像(WSI)被切割成大小为1024×1024像素的小块,以便于后续的处理和分析。
- 最后,需要筛选出空白和对齐不良的区域,确保BCI数据集中的图像块质量。
整个流程的目的是为了创建一个结构对齐的数据集,该数据集可以用于训练和评估图像到图像的翻译模型,以生成准确的IHC图像,从而帮助评估乳腺癌组织中HER2的表达水平。
三、DeepLIIF算法优化免疫组化染色分析

一作&通讯
| 姓名 | 单位 | 单位中文翻译 |
|---|---|---|
| Parmida Ghahremani | Department of Computer Science, Stony Brook University | 石溪大学计算机科学系 |
| Travis J. Hollmann | Department of Medical Physics, Memorial Sloan Kettering Cancer Center | 纪念斯隆凯特琳癌症中心医学物理部 |
| Saad Nadeem | Department of Pathology, Memorial Sloan Kettering Cancer Center | 纪念斯隆凯特琳癌症中心病理学部 |
文献概述
这篇文章介绍了一种名为DeepLIIF的新型多任务深度学习算法,它能够提高免疫组化(IHC)染色组织切片中蛋白质表达评估的准确性和可重复性。
IHC染色是临床决策中的关键技术,尤其是在癌症分类、残留疾病检测和突变检测等方面。然而,传统的亮场染色技术动态范围有限,且不同染色通道之间存在重叠,需要复杂的数字化染色分离作为预处理步骤。
DeepLIIF算法利用了与IHC相同的组织切片上的独特共注册的多重免疫荧光(mpIF)训练数据,可以同时将低成本的IHC图像翻译成更昂贵、信息更丰富的mpIF表示,自动分割相关细胞,并量化蛋白质表达。这种方法不仅提高了IHC量化的准确性和可重复性,而且通过多任务学习提高了模型的通用性和鲁棒性。
文章还介绍了一种新的核膜染色剂LAP2beta,它具有超过95%的细胞覆盖率,有助于改善细胞的分离和蛋白质表达量的量化。DeepLIIF在训练时仅使用干净的IHC Ki67图像,但也能很好地推广到更嘈杂和有缺陷的图像,以及其他核和非核标记物,如CD3、CD8、BCL2、BCL6、MYC、MUM1、CD10和TP53。
研究者们使用多种数据集评估了DeepLIIF的性能,包括内部测试集、BC数据集和NuClick数据集,并与其他最先进的方法进行了比较。DeepLIIF在所有评估指标上均优于其他模型,包括像素准确率、Dice分数、交集比(IoU)和聚合Jaccard指数(AJI)。
此外,文章还讨论了DeepLIIF在不同数据集、染色技术和组织/癌症类型中的泛化能力,并展示了其在处理不同标记物的IHC图像时的性能。研究者们还对DeepLIIF进行了一系列消融研究,以评估每个组件对模型性能的影响。
最后,文章讨论了DeepLIIF在临床应用中的潜力,包括提高病理学家的解释能力以及为下游分析/算法提供更准确的细胞级分割、分类或评分。作者还提到了将DeepLIIF应用于H&E图像的意外成功,并展望了该技术在未来可能的发展方向。
简要分析
图1提供了DeepLIIF(深度学习推断多重免疫荧光)流程的概览,以及使用不同棕色/DAB标记物(包括BCL2、BCL6、CD10、CD3/CD8、Ki67等)的样本输入IHC(免疫组化)图像,以及对应的由DeepLIIF生成的苏木精/多重免疫荧光(haematoxylin/mpIF)模式和分类分割掩模。
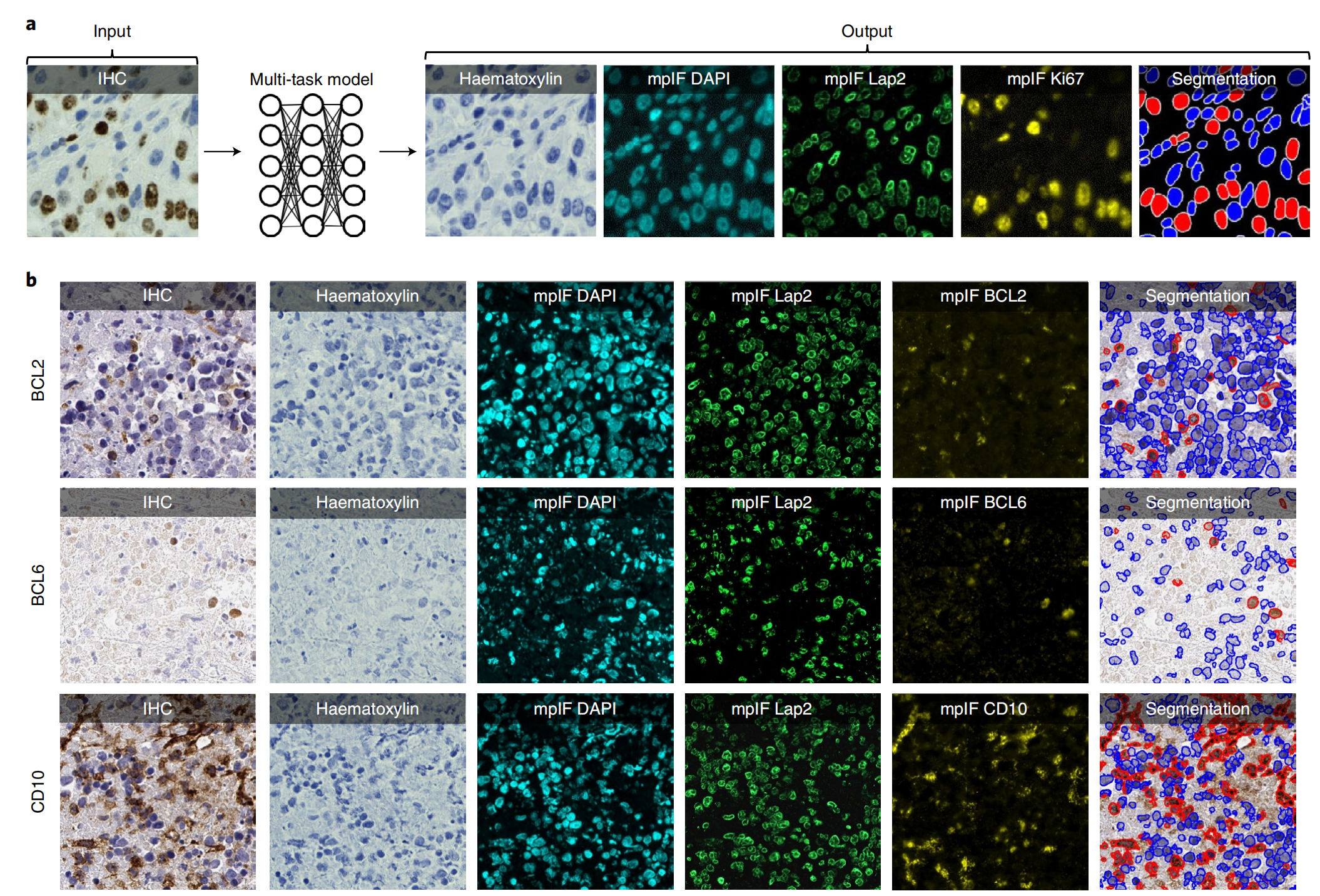
以下是对图1内容的分析:
a. DeepLIIF流程概述:
- DeepLIIF是一个多任务深度学习框架,它能够同时推断出对应的苏木精通道、mpIF DAPI(4′,6-二氨基-2-苯基吲哚)和mpIF蛋白质表达(例如Ki67、CD3、CD8等),以及阳性/阴性蛋白质细胞分割。
- 该模型内置了可解释性和可解释性,不依赖于粗糙的激活/注意力图。
- 在分割掩模中,红色细胞表示具有阳性蛋白质表达的细胞(输入IHC中的棕色/DAB细胞),而蓝色细胞代表阴性细胞(输入IHC中的蓝色细胞)。
b. 不同IHC标记物的DeepLIIF生成的haematoxylin/mpIF模式和分割掩模示例:
- DeepLIIF在清洁的IHC Ki67核标记图像上进行训练,能够泛化到更嘈杂的图像以及其他IHC核/细胞质标记图像。
- 图中展示了使用不同标记物的IHC图像,以及DeepLIIF生成的相应haematoxylin/mpIF模式和分割掩模。
- 这些模式和分割掩模有助于更准确地识别和量化蛋白质表达,提高了病理学分析的质量和可靠性。
总的来说,图1展示了DeepLIIF如何通过深度学习技术处理和分析IHC图像,以及如何生成有助于病理学家进行更准确诊断的模式和分割掩模。这种技术的进步对于提高疾病诊断和治疗的准确性具有重要意义。
四、PC-StainGAN:一种用于病理图像染色转换的无监督学习方法

一作&通讯
| 角色 | 姓名 | 单位 |
|---|---|---|
| 第一作者 | Shuting Liu | 清华大学深圳国际研究生院 生命与健康系 |
| 第一作者 | Baochang Zhang | 德国慕尼黑工业大学 计算机辅助医学程序组 |
| 通讯作者 | Tian Guan | 清华大学深圳国际研究生院 生命与健康系 |
| 通讯作者 | Yonghong He | 清华大学深圳国际研究生院 生命与健康系 |
文献概述
这篇文章是关于一种新的无监督学习方法,用于在病理学领域中实现苏木精-伊红(H&E)染色图像和Ki-67免疫组化(IHC)染色图像之间的转换。
这种方法被称为病理一致性约束生成对抗网络(PC-StainGAN)。
文章的主要贡献包括:
-
提出了一种新的对抗学习方法,用于从H&E染色图像生成Ki-67染色图像,这对于资源有限的地区尤其有用,因为IHC染色的可访问性较低。
-
该方法利用结构相似性约束和跳跃连接来提高结构细节的保留,并首次提出了病理一致性约束和病理表示网络,以确保生成的图像和源图像在不同染色域中具有相同的病理属性。
-
通过在两个不同的未配对的组织病理学数据集上进行实验,证明了该方法的有效性,并在不平衡数据集上展示了其稳定性和鲁棒性。
-
该方法在临床虚拟染色和计算机辅助多染色组织图像分析方面具有显著潜力。
文章还讨论了癌症的发病率和死亡率,以及病理学在癌症诊断中的重要性。作者指出,H&E染色虽然成本较低,但有时不足以区分正常细胞和癌细胞,而IHC检查可以提供更多的诊断证据。然而,IHC检查通常需要更多的时间和劳动力,并且在低收入和中等收入国家的应用受到成本和技术技能的限制。
文章最后讨论了未来的工作方向,包括改进模型架构、优化超参数、扩展到多域转换,并考虑使用联邦学习来解决数据隐私问题。
简要分析
Fig. 3 提供了文章中提出的 PC-StainGAN 方法的概述,该方法包含两个生成器(generators),每个生成器由编码器-解码器架构(encoder-decoder architecture)和一个病理表示网络(pathological representation network)组成。
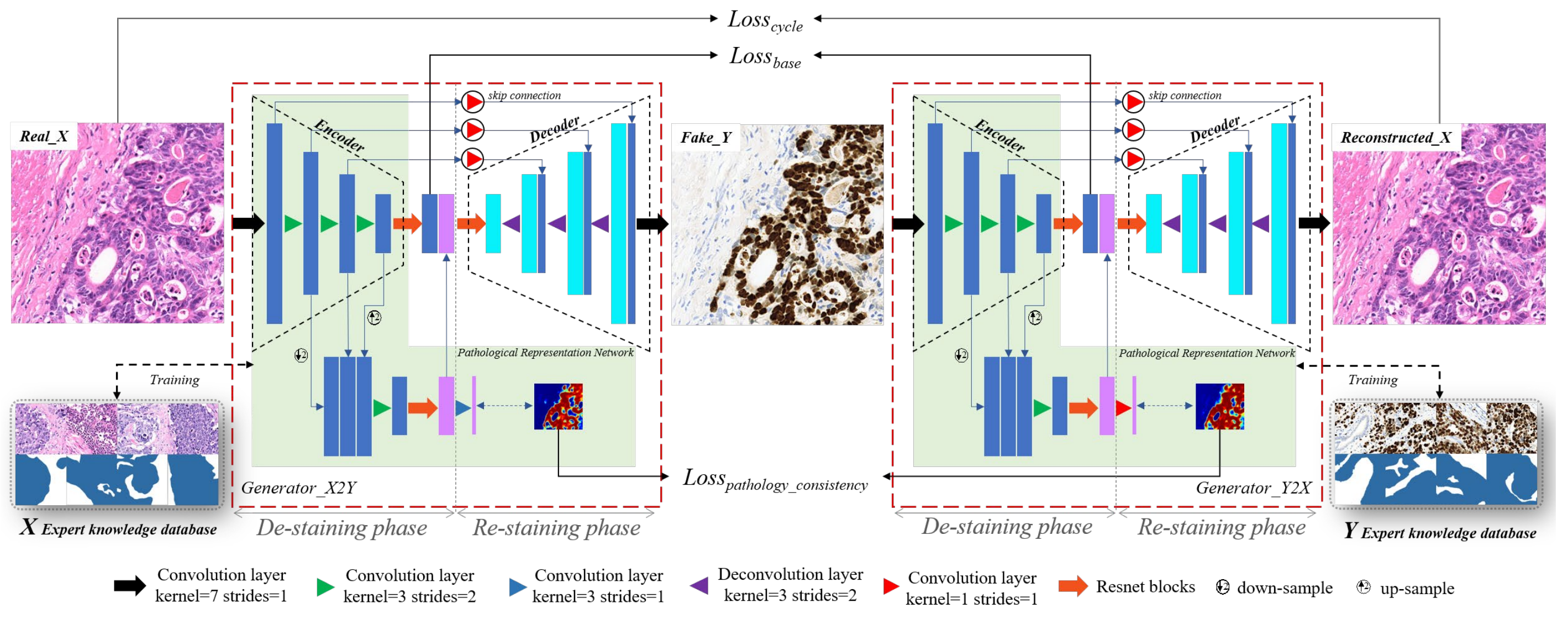
以下是对图中各个组成部分的分析:
-
编码器-解码器架构:每个生成器都包含一个编码器和一个解码器。编码器负责将输入图像(H&E染色图像或Ki-67染色图像)转换为一个中间特征表示,而解码器则将这个特征表示转换回图像域,生成目标染色域的图像。
-
病理表示网络:这个网络用于提取图像中的病理特征,并且通过专家知识数据库和训练数据集共同训练。这意味着网络不仅学习如何转换图像的染色风格,还学习如何识别和保留与病理相关的特征。
-
专家知识数据库:由经验丰富的病理学家标注的数据集,其中蓝色区域代表癌症病变区域,白色区域代表正常组织区域。这些标注提供了关于病理特征的专家级知识,帮助训练病理表示网络。
-
目标函数:模型的训练涉及到四种损失函数:
- 对抗损失(Adversarial loss):确保生成的图像在视觉上与目标染色域的图像无法区分。
- 循环一致性损失(Cycle consistency loss):包括L1损失和结构相似性约束(SSIM loss),确保转换过程中图像的结构信息得以保留。
- 病理一致性损失(Pathological consistency loss):确保生成的图像与原始图像在病理特征上保持一致。
- 基础空间对齐损失(Base space aligned loss):确保不同染色域的图像在基础特征空间中对齐,增强了模型对病理特征的捕捉能力。
总的来说,Fig. 3 描述了一个复杂的深度学习模型,它不仅能够转换图像的染色风格,还能够保留和学习图像中的病理特征,这对于提高病理图像分析的准确性和效率至关重要。通过结合专家知识数据库和训练数据集,模型能够更好地理解和模拟病理学家的诊断过程。
五、深度学习辅助的H&E至IHC染色转换技术

一作&通讯
| 角色 | 姓名 | 单位 |
|---|---|---|
| 第一作者 | Joseph Boyd | 中央苏佩莱克大学MICS实验室,巴黎-萨克雷大学,法国 |
| 通讯作者 | Maria Vakalopoulou⋆ (共同通讯作者) | 中央苏佩莱克大学MICS实验室,巴黎-萨克雷大学,法国 |
| 通讯作者 | Stergios Christodoulidis∗ (共同通讯作者) | 中央苏佩莱克大学MICS实验室,巴黎-萨克雷大学,法国 |
文献概述
这篇文章介绍了一种基于深度学习的图像处理技术,用于在全切片图像(Whole Slide Images,WSI)中进行染色转移。
研究团队提出了一种改进的循环生成对抗网络(CycleGAN)模型,通过引入感兴趣区域(Region of Interest,RoI)判别器,来提高染色转移的准确性。
这种方法特别适用于将苏木精-伊红(H&E)染色的图像转换为免疫组化(IHC)染色的图像,这对于病理学家解读组织切片中的转移性癌细胞非常有帮助。
文章的主要贡献包括:
- 提出了一种基于RoI的判别器网络,该网络可以从未配对的数据集中学习,并强制执行生物学一致性。
- 在两个数据集上展示了该方法在组织病理学patch上的染色转移性能优于现有技术。
- 该方法可以作为临床可视化工具,以及在诊断转移性细胞的分割流程中的潜在应用。
研究团队还讨论了如何通过自动图像处理流程构建用于训练RoI判别器的边界框库,以及如何实现和训练CycleGAN模型。他们在两个乳腺癌数据集上进行了实验,证明了他们的方法在定位DAB染色(一种用于标记癌细胞的染色)方面的准确性高于基线模型。
文章最后讨论了该方法的局限性和未来工作的方向,包括减少假阳性结果和进一步提高模型的准确性。研究工作得到了ANR Hagnodice和ARC SIGNIT的支持,并使用了CentraleSupélec和École Normale Supérieure Paris-Saclay的高性能计算资源。
简要分析
Fig. 3展示了使用不同方法进行染色转移的样本结果。
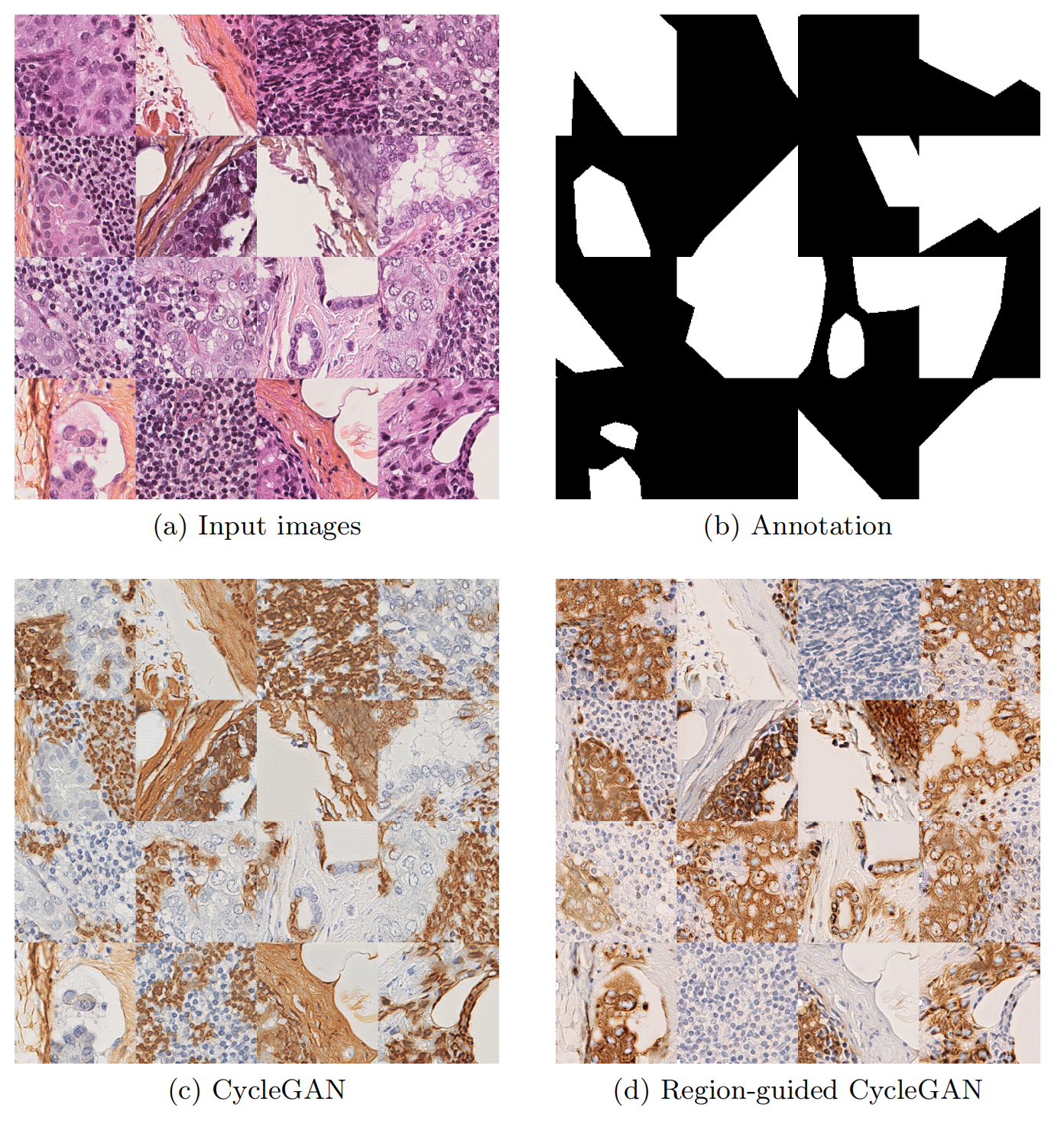
这个图包含了四部分,分别是:
(a) H&E样本:这部分展示了来自Gustave Roussy (GR) 数据集的苏木精-伊红(H&E)染色的组织切片图像。这些图像是输入数据,用于后续的染色转移过程。
(b) 真实标注掩膜(Ground Truth Annotation Masks):这部分展示了与(a)部分相对应的专家标注的掩膜图像,这些掩膜图像标识了组织切片中癌细胞的位置。这些标注作为染色转移的目标,即我们希望染色转移后的图像中,癌细胞的位置能够与这些掩膜图像相匹配。
© 基线CycleGAN染色转移:这部分展示了使用基线CycleGAN模型进行染色转移的结果。CycleGAN是一种用于图像到图像翻译的生成对抗网络,它能够在没有配对数据的情况下进行训练。然而,基线CycleGAN模型在定位DAB染色(一种用于标记癌细胞的染色)方面存在问题,这可能导致癌细胞的检测不准确。
(d) 提出的区域引导染色转移:这部分展示了使用文章中提出的区域引导CycleGAN模型进行染色转移的结果。与基线CycleGAN模型相比,区域引导模型通过引入感兴趣区域(RoI)判别器,显著提高了染色转移的准确性,特别是在定位DAB染色方面。这表明该方法能够更准确地识别和转移癌细胞的位置,从而提高了病理诊断的准确性。
总的来说,Fig. 3通过对比不同的染色转移方法,展示了区域引导CycleGAN模型在提高染色转移准确性方面的潜力,这对于病理学家在诊断癌症时识别癌细胞具有重要意义。























 1407
1407

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








