
近日,中国农业大学农学院小麦研究中心在GigaScience杂志上在线发表了题为“SnpHub: an easy-to-set-up web server framework for exploring large-scale genomic variation data in the post-genomic era with applications in wheat”的文章(https://doi.org/10.1093/gigascience/giaa060)。该研究提出了一种可在任意Linux服务器上通过简易配置并快速部署的大规模基因组变异数据库模型——SnpHub(主页:http://guoweilong.github.io/SnpHub)。该数据库模型适用于对大规模样本的VCF文件实现在线的快速检索和轻量级的分析和可视化功能。该工作目前同时提供了7套小麦及祖先种变异数据集的查询数据库:Wheat-SnpHub-Portal (主页:http://wheat.cau.edu.cn/Wheat_SnpHub_Portal/)。
随着高通量测序成本的持续下降,我们能够对“数以百计”甚至“数以千计”规模的样品进行全基因组重测序。目前,水稻、玉米和小麦等作物逐渐积累了大量的重测序数据,为相关研究提供了有重要价值的基因组学数据。然而,对几十T大小基因组变异数据进行检索和管理需要一定的生物信息学基础。虽然有一些物种已经有可公开查询的基因型数据库,但这些数据库通常是独立开发,功能有异;同时对于导入新的样本数据和更新数据库存在困难。本公众号在之前的推送“拿什么迎接你:即将到来的海量重测序数据?”中也讨论过,后基因组学时代的研究者还会遇到来自“超大规模数据的存储管理和读取”、“meta数据利用与数据再分析”、“对不同物种或基因组版本的支持”、“生信人才培养和队伍建设”等不同等方面的挑战。
为应对这些困难与挑战,该研究基于Shiny/R框架开发了SnpHub数据库模型。该数据库模型适用于大规模重测序数据“再分析”的数据库系统模型。利用该数据库模型,可在已有的VCF数据的基础上,在本地快速搭建一个高效率查询和快速轻量级分析的数据库。SnpHub的用户可以通过指定样本列表和特定的基因组区域在大量基因组变异数据中快速定位,并通过Shiny/R框架执行轻量级分析和可视化(图1)。用户只需打开浏览器访问数据集对应的网址,点击鼠标、输入一些基本信息就能得到分析与可视化结果,而繁杂的数据转换、处理任务将由SnpHub自动完成。
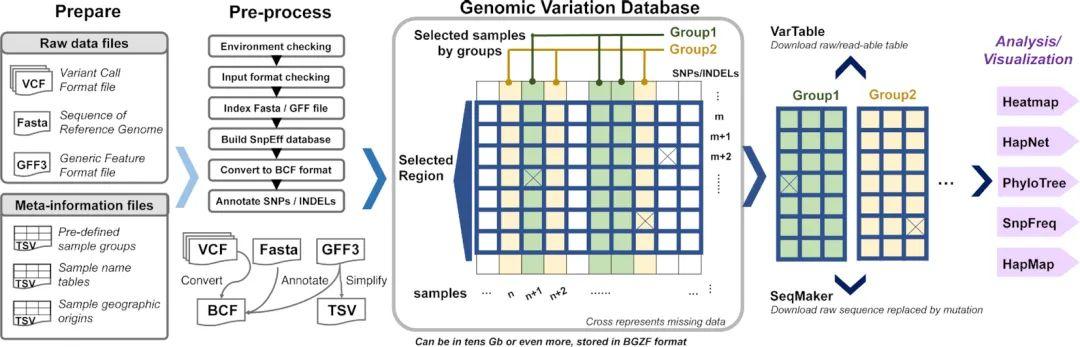
图1. SnpHub数据库模型的设计模式图
为了提升易用性,SnpHub在四个主要方面进行了设计和优化。(一)实现了基因型快速检索,极大的减少了磁盘的读写频率,提高了分析速度,使用户能在网页上即时得到分析结果。(二)考虑到样本在送测序、编号和展示分析时可能使用不同的名字,SnpHub采用了“三套样本名策略”,分别对应“源数据文件(VCF)”、供用户网页端查询的“编号名简短版本)”(和分析绘图输出“展示名(可读性好)”使用,使得数据管理分析更加轻松。(三)在一些分析需求中,需要对两组或多组样本进行比较展示(如不同地理来源、生态型,不同的抗逆性状等)。SnpHub支持多种方式样本分组,使得用户可以在群体水平进行比较分析。分组可由实例管理员预先定义,也可由用户临时定义。(四)为满足用户对分析结果和作图进行后续修改的需要,SnpHub支持将分析结果得到的表格下载为CSV格式(表格),图片保存为PNG格式(位图)或PDF格式(矢量图),以便进行个性化修改。
SnpHub提供的功能主要包括:查看当前数据集基本信息、变异数据表格查询(VarTable)(直接查询/下载原始变异数据)、变异数据的热图可视化(Heatmap)(查看指定区间的变异情况)、特定基因/区间的单倍型网络分析(HapNet)(研究指定区间的群体遗传构成)、样本列表或样本集合在指定区间的Phylogenetic Tree分析(PhyloTree)(研究指定区间的样本间关系)、变异信息的地图映射(HapMap)(不同基因型的地理分布信息)、将变异频率和功能在基因结构上显示(SnpFreq)(快速寻找重要突变位点,也可用于对比位点组间频率分化情况)、样本序列的替换生成等(SeqMaker)(图2)。在之前的推送中,我们已经对SnpHub的功能进行过整体性的概述(一千零一技 | Wheat-SnpHub-Portal数据库介绍及使用示例),并对其中的VarTable功能进行过详细介绍(一千零一技 | SnpHub:利用VarTable查询变异数据),今后我们还将继续推送每项功能的详细介绍、具体使用案例等内容。
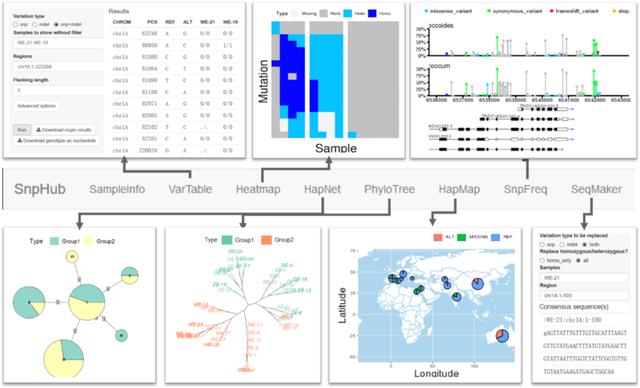
图2. SnpHub数据库模型支持的查询、分析和可视化功能示意图。
SnpHub的定位是对于大型基因组变异数据的数据库的轻量级分析框架,为了满足查询和分析的即时性,更侧重对特定区域或基因的分析,而非全基因组尺度的分析(如全基因组关联分析等)。为了进一步说明SnpHub的适用倾向,本工作将SnpHub与其它三种相关的数据库进行了比较(表1)。总体上,SnpHub允许用户交互地探索包含大量基因组多样性的数据集,并且在执行轻量级重分析方面更为强大。
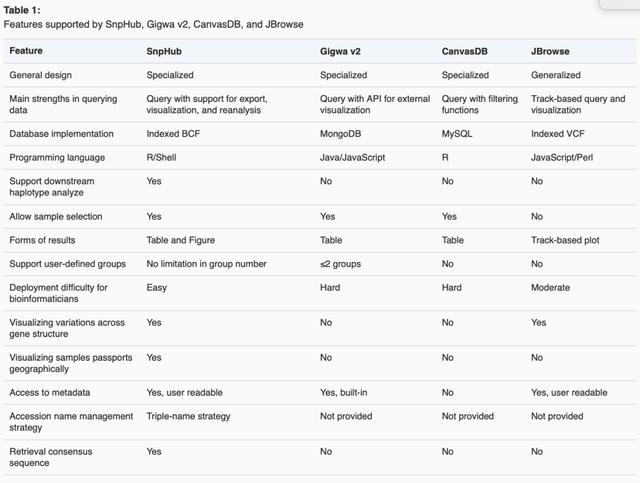
表1. SnpHub 与Gigwa v2, CanvasDB, JBrowse 功能比较。
包括普通小麦、粗山羊草、二粒小麦在内的小麦族中不同倍型物种的基因组和重测序数据已经陆续公布。利用这些已经公布的数据集(表2),我们构建7套SnpHub数据库实例,并搭建了“Wheat-SnpHub-Portal(小麦SnpHub门户)”网站,为广大小麦研究同行对这些数据的检索和利用提供便利。Wheat-SnpHub-Portal将在未来持续更新小麦及其祖先种的基因组变异数据集。(温馨提示:每个SnpHub实例需要10s-30s的加载时间,请耐心等待)。
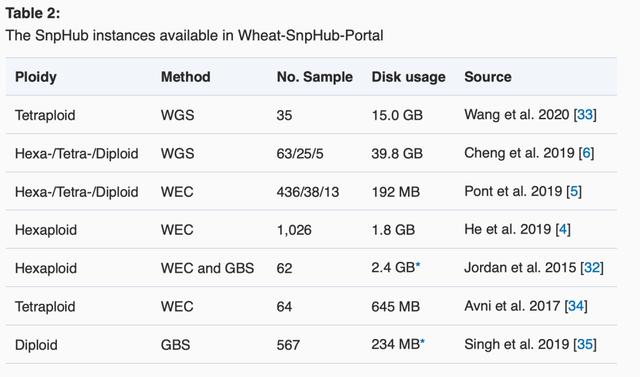
表2. Wheat-SnpHub-Portal中所包含的数据集。
中国农业大学农学院小麦研究中心的王文熙(本科生)与王梓豪(博士生)为该论文的共同第一作者,中国农业大学农学院的郭伟龙副教授为该论文的通讯作者。论文其他主要参与者还包括中国农业大学农学院李欣桐(本科生)、倪中福教授、胡兆荣副教授、辛明明教授、彭慧茹教授、姚颖垠教授和孙其信教授。相关工作得到了国家自然科学基金(31701415)、国家重点研发计划(2018YFD0100803 and 2016YFD0100801)和中央高校基本科研业务费的支持。
感谢西北农林科技大学的姜雨教授团队和山东农业大学孔令让教授团队提供相关数据集的VCF文件。感谢谢小明(本科生)、陈永明、杨正钊、余阔海等同学提供技术支持。感谢中国农业大学农学院小麦研究中心的各位师生等初期用户对该数据库功能的反馈和建议。
一作心声:
小麦研究已经进入了后基因组时代,包括基因组学在内的各种组学数据已成为了这个时代的“基础设施”,如何利用和挖掘这些海量数据是一个绕不开的话题。最初想到做这样的数据库,是因为小麦研究中心各位同学对测序数据的利用中有极大的需求,但又存在诸多困难。在开发过程中,我们逐渐意识到,虽然每位研究者关注的问题不尽相同,但涉及的数据分析方法和策略存在一定的相似性;设计一套既能满足共性需求,同时又能通过调整参数、样本等进行个性化分析工具可能会研究工作和科研合作起到事半功倍的效果。后来,随着功能的增加与对类似软件文章的阅读,我们发现这样一款封装了多种较为成熟的可视化手段的数据库软件可以帮助到更多研究者,从而决定走向了投稿的漫漫长路。很高兴这个工具最终能够成功发表,能够让更多人看到、用到。也非常感谢“小麦研究联盟”公众号提供平台,让我们可以在文章正式发表前就对这项工作进行介绍。
和很多生物信息学软件/数据库的工作一样:“文章的正式发表也意味着相关工作的才刚正式开始”。我们非常欢迎专家、同行、朋友同行反馈!
(联系方式:王文熙,wangwenxi20@gmail.com)
Citation:
Wenxi Wang, Zihao Wang, Xintong Li, Zhongfu Ni, Zhaorong Hu, Mingming Xin, Huiru Peng, Yingyin Yao, Qixin Sun, Weilong Guo. (2020) SnpHub: an easy-to-set-up web server framework for exploring large-scale genomic variation data in the post-genomic era with applications in wheat , GigaScience , 9(6):giaa060
(引用SnpHub的同时,也请引用对应的数据集的来源文章

)


近日,中国农业大学农学院小麦研究中心在GigaScience杂志上在线发表了题为“SnpHub: an easy-to-set-up web server framework for exploring large-scale genomic variation data in the post-genomic era with applications in wheat”的文章(https://doi.org/10.1093/gigascience/giaa060)。该研究提出了一种可在任意Linux服务器上通过简易配置并快速部署的大规模基因组变异数据库模型——SnpHub(主页:http://guoweilong.github.io/SnpHub)。该数据库模型适用于对大规模样本的VCF文件实现在线的快速检索和轻量级的分析和可视化功能。该工作目前同时提供了7套小麦及祖先种变异数据集的查询数据库:Wheat-SnpHub-Portal (主页:http://wheat.cau.edu.cn/Wheat_SnpHub_Portal/)。
随着高通量测序成本的持续下降,我们能够对“数以百计”甚至“数以千计”规模的样品进行全基因组重测序。目前,水稻、玉米和小麦等作物逐渐积累了大量的重测序数据,为相关研究提供了有重要价值的基因组学数据。然而,对几十T大小基因组变异数据进行检索和管理需要一定的生物信息学基础。虽然有一些物种已经有可公开查询的基因型数据库,但这些数据库通常是独立开发,功能有异;同时对于导入新的样本数据和更新数据库存在困难。本公众号在之前的推送“拿什么迎接你:即将到来的海量重测序数据?”中也讨论过,后基因组学时代的研究者还会遇到来自“超大规模数据的存储管理和读取”、“meta数据利用与数据再分析”、“对不同物种或基因组版本的支持”、“生信人才培养和队伍建设”等不同等方面的挑战。
为应对这些困难与挑战,该研究基于Shiny/R框架开发了SnpHub数据库模型。该数据库模型适用于大规模重测序数据“再分析”的数据库系统模型。利用该数据库模型,可在已有的VCF数据的基础上,在本地快速搭建一个高效率查询和快速轻量级分析的数据库。SnpHub的用户可以通过指定样本列表和特定的基因组区域在大量基因组变异数据中快速定位,并通过Shiny/R框架执行轻量级分析和可视化(图1)。用户只需打开浏览器访问数据集对应的网址,点击鼠标、输入一些基本信息就能得到分析与可视化结果,而繁杂的数据转换、处理任务将由SnpHub自动完成。

图1. SnpHub数据库模型的设计模式图
为了提升易用性,SnpHub在四个主要方面进行了设计和优化。(一)实现了基因型快速检索,极大的减少了磁盘的读写频率,提高了分析速度,使用户能在网页上即时得到分析结果。(二)考虑到样本在送测序、编号和展示分析时可能使用不同的名字,SnpHub采用了“三套样本名策略”,分别对应“源数据文件(VCF)”、供用户网页端查询的“编号名简短版本)”(和分析绘图输出“展示名(可读性好)”使用,使得数据管理分析更加轻松。(三)在一些分析需求中,需要对两组或多组样本进行比较展示(如不同地理来源、生态型,不同的抗逆性状等)。SnpHub支持多种方式样本分组,使得用户可以在群体水平进行比较分析。分组可由实例管理员预先定义,也可由用户临时定义。(四)为满足用户对分析结果和作图进行后续修改的需要,SnpHub支持将分析结果得到的表格下载为CSV格式(表格),图片保存为PNG格式(位图)或PDF格式(矢量图),以便进行个性化修改。
SnpHub提供的功能主要包括:查看当前数据集基本信息、变异数据表格查询(VarTable)(直接查询/下载原始变异数据)、变异数据的热图可视化(Heatmap)(查看指定区间的变异情况)、特定基因/区间的单倍型网络分析(HapNet)(研究指定区间的群体遗传构成)、样本列表或样本集合在指定区间的Phylogenetic Tree分析(PhyloTree)(研究指定区间的样本间关系)、变异信息的地图映射(HapMap)(不同基因型的地理分布信息)、将变异频率和功能在基因结构上显示(SnpFreq)(快速寻找重要突变位点,也可用于对比位点组间频率分化情况)、样本序列的替换生成等(SeqMaker)(图2)。在之前的推送中,我们已经对SnpHub的功能进行过整体性的概述(一千零一技 | Wheat-SnpHub-Portal数据库介绍及使用示例),并对其中的VarTable功能进行过详细介绍(一千零一技 | SnpHub:利用VarTable查询变异数据),今后我们还将继续推送每项功能的详细介绍、具体使用案例等内容。

图2. SnpHub数据库模型支持的查询、分析和可视化功能示意图。
SnpHub的定位是对于大型基因组变异数据的数据库的轻量级分析框架,为了满足查询和分析的即时性,更侧重对特定区域或基因的分析,而非全基因组尺度的分析(如全基因组关联分析等)。为了进一步说明SnpHub的适用倾向,本工作将SnpHub与其它三种相关的数据库进行了比较(表1)。总体上,SnpHub允许用户交互地探索包含大量基因组多样性的数据集,并且在执行轻量级重分析方面更为强大。

表1. SnpHub 与Gigwa v2, CanvasDB, JBrowse 功能比较。
包括普通小麦、粗山羊草、二粒小麦在内的小麦族中不同倍型物种的基因组和重测序数据已经陆续公布。利用这些已经公布的数据集(表2),我们构建7套SnpHub数据库实例,并搭建了“Wheat-SnpHub-Portal(小麦SnpHub门户)”网站,为广大小麦研究同行对这些数据的检索和利用提供便利。Wheat-SnpHub-Portal将在未来持续更新小麦及其祖先种的基因组变异数据集。(温馨提示:每个SnpHub实例需要10s-30s的加载时间,请耐心等待)。

表2. Wheat-SnpHub-Portal中所包含的数据集。
中国农业大学农学院小麦研究中心的王文熙(本科生)与王梓豪(博士生)为该论文的共同第一作者,中国农业大学农学院的郭伟龙副教授为该论文的通讯作者。论文其他主要参与者还包括中国农业大学农学院李欣桐(本科生)、倪中福教授、胡兆荣副教授、辛明明教授、彭慧茹教授、姚颖垠教授和孙其信教授。相关工作得到了国家自然科学基金(31701415)、国家重点研发计划(2018YFD0100803 and 2016YFD0100801)和中央高校基本科研业务费的支持。
感谢西北农林科技大学的姜雨教授团队和山东农业大学孔令让教授团队提供相关数据集的VCF文件。感谢谢小明(本科生)、陈永明、杨正钊、余阔海等同学提供技术支持。感谢中国农业大学农学院小麦研究中心的各位师生等初期用户对该数据库功能的反馈和建议。
一作心声:
小麦研究已经进入了后基因组时代,包括基因组学在内的各种组学数据已成为了这个时代的“基础设施”,如何利用和挖掘这些海量数据是一个绕不开的话题。最初想到做这样的数据库,是因为小麦研究中心各位同学对测序数据的利用中有极大的需求,但又存在诸多困难。在开发过程中,我们逐渐意识到,虽然每位研究者关注的问题不尽相同,但涉及的数据分析方法和策略存在一定的相似性;设计一套既能满足共性需求,同时又能通过调整参数、样本等进行个性化分析工具可能会研究工作和科研合作起到事半功倍的效果。后来,随着功能的增加与对类似软件文章的阅读,我们发现这样一款封装了多种较为成熟的可视化手段的数据库软件可以帮助到更多研究者,从而决定走向了投稿的漫漫长路。很高兴这个工具最终能够成功发表,能够让更多人看到、用到。也非常感谢“小麦研究联盟”公众号提供平台,让我们可以在文章正式发表前就对这项工作进行介绍。 和很多生物信息学软件/数据库的工作一样:“文章的正式发表也意味着相关工作的才刚正式开始”。我们非常欢迎专家、同行、朋友同行反馈! (联系方式: 王文熙,wangwenxi20@gmail.com)Citation:
Wenxi Wang, Zihao Wang, Xintong Li, Zhongfu Ni, Zhaorong Hu, Mingming Xin, Huiru Peng, Yingyin Yao, Qixin Sun, Weilong Guo. (2020) SnpHub: an easy-to-set-up web server framework for exploring large-scale genomic variation data in the post-genomic era with applications in wheat , GigaScience , 9(6):giaa060
(引用SnpHub的同时,也请引用对应的数据集的来源文章 )
)






















 1057
1057











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








