前言:
从NCBI上下载的sra格式的测序文件经过Sratoolkit软件转换后变成fastqc格式的文件,我们如何能知道下载的这个数据是否准确并值得信赖,这时候我们通过conda安装一个fastqc文件的质控软件来可视化的展示转化后的数据结果。至于为什么利用conda来安装而不是直接安装在服务器上,是因为conda能帮助我们解决一些软件过程中需要的依赖,而且避免因为一些软件需要特殊的环境(有的需要python3环境,有的需要python2等等)从而会导致软件无法正常运行,我们下面先来安装conda,再来安装质检软件,顺利安装conda结束后,质控软件只需要一行命令就可以解决了!那么我们下面正式开始
Conda的安装与使用:
安装环境:
Centos Linux release 7.8.2003 (Core)
Conda 4.8.5
conda和miniconda本质上是一个东西,miniconda是conda的精简版,需要什么就装什么,不需要的就不装,所以我个人使用的是miniconda。
miniconda官方网站:
https://docs.conda.io/en/latest/miniconda.html其他系统的可以自行下载选择自己的安装包:
进入到指定的软件安装目录:
因为miniconda本质上并不属于生信软件,所以我没有把他装在之前的生信软件目录(/home/biological/biosoftware)下(这也是一个很好的习惯,大家也可以借鉴这种有点强迫症的思想,这样可以很清楚的管理自己服务器下的文件),而是放在了根目录下:
cd ~下载安装包
wget -c https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh下载完成以后,给下载的文件赋予权限:
chmod 777 Miniconda3-latest-Linux-x86_64.sh然后运行:
bash Miniconda3-latest-Linux-x86_64.sh程序运行时,最简单的方式是一路“yes”下来,但是conda安装中,当你的服务器之前有安装过软件的话,conda可能会污染你原来的环境,所以在安装conda中,最好不要将其加入到环境变量里,碰到下面的选项时,注意要选择 “no”

因为我们没有选择将conda加入环境变量中,所以输入conda是会报找不到此命令的,而且也不能通过conda --version来验证是否正确安装,那么我们要如何启动conda呢?
因为我们软件安装在根目录(~)中,所以我们进入到安装目录里:
cd /root/miniconda3/再进入到bin/目录下找到一个名为activate的文件
我们首先给activate这个文件,修改权限,不然不能正常使用
chmod 777 activateconda启动:
利用
. ./activate的命令,启动conda
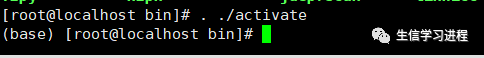
这时候,我们可以看到命令行前面,出现了(base),这就代表我们已经进入了conda的环境,这时候利用conda --version就能看到安装的conda 版本号
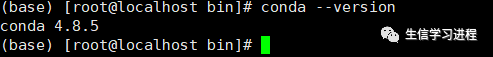
这时候conda就已经正确安装完成了
添加频道:
conda config --add channels biocondaconda config --add channels conda-forge conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free/conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main/conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forge/conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/bioconda/利用conda安装fastqc:
安装fastqc:
conda install fastqc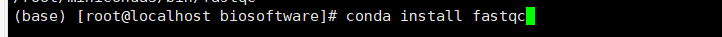
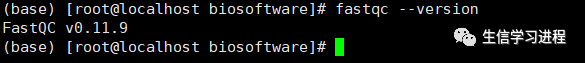
查看是否正确安装及安装的fastqc软件版本号:
我们可以利用fastqc -h查看官方的技术文档:
这里只简单列出几个常用的参数:
-o:FastQC生成的报告文件的储存路径 (./ 代表的是当前目录)
-t:需要用到的线程数(越多速度越快,但是要按照自己机器的配置来选)
进入到需要处理的数据所在目录:
/home/biological/biodata/test/RNAseq_out运行软件:
fastqc -o ./ -t 8 SRR12062121_1.fastq.gz SRR12062121_2.fastq.gz
进入到输出目录,查看运行结果:
tree(如果提示没有tree命令,则可以用yum install tree来下载这个查看目录文件的小工具,这个工具和fastqc软件无关,你可以用在任何一个目录下,只是为了方便管理目录文件的小工具)
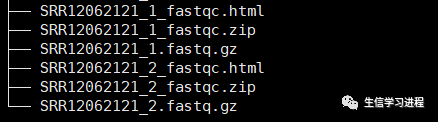
能看到每一个数据都得到了一个.html网页文件与一个压缩包文件。
这时候我们就可以打开.html文件,查看分析结果了:
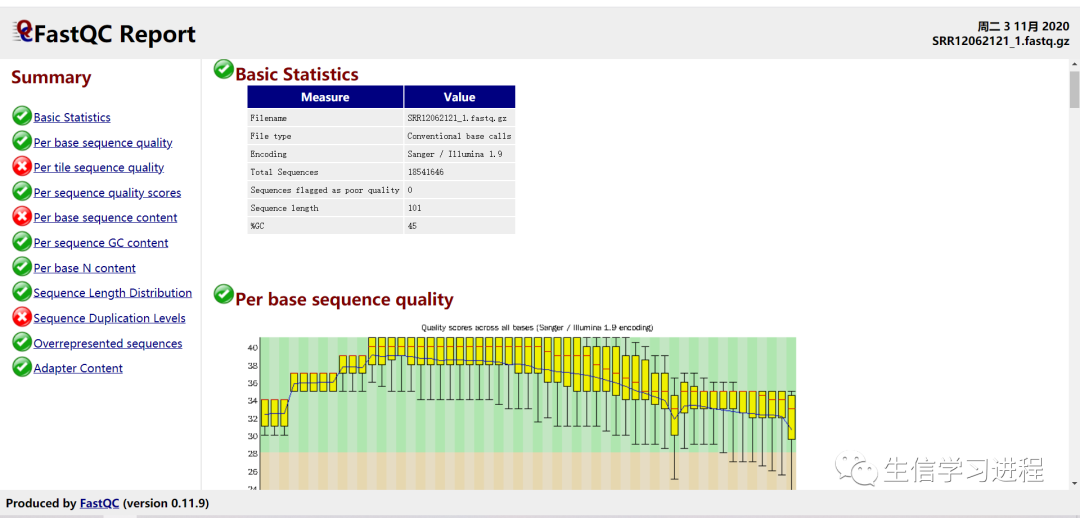
这里只对基本信息图表所代表的生物学意义用图示的方法讲解,其他的详细图表可自行查找相关资料进行学习,在这里因为篇幅关系就不再一一展开讲解了。

后面我们不需要fastqc这个软件,所以我们可以退出当前所在的conda环境。
conda关闭:
conda deactivate




















 2226
2226











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








