论文地址:https://papers.nips.cc/paper/6448-combining-fully-convolutional-and-recurrent-neural-networks-for-3d-biomedical-image-segmentation.pdf
论文摘要
3D图像的分割是生物医学图像分析中的基本问题。深度学习(DL)方法已经实现了最先进的分割性能。利用神经网络探查3D图片深度的信息,已知的DL分割方法:①3D卷积;②在与2D正交的平面上的2D卷积图像切片;③多向LSTM。以上方法对常见的3D生物医学图像中的高度各向异性尺寸不是很兼容。在本文中,我们基于组合提出了用于3D图像分割的新DL框架——使用完全卷积网络(FCN)负责提取片内信息,利用递归神经网络(RNN)提取片间信息。据我们所知,这是第一个用于3D图像的DL框架明确利用3D图像各向异性的分割。评估使用来自ISBI神经元结构分割挑战和内部的数据集,这个数据集用于3D真菌分割的图像堆栈。基于DL的3D分割方法相比较,我们的方法表现出了更为优异的结果。
现有方法的局限
已知的基于DL的3D分割方法主要存在三个问题。
- 首先,简单地连接 2D图片拼接成3D不能利用沿z方向的空间相关性。
- 第二, 3D卷积可能导致极高的计算成本(例如,高内存) 消费和长时间训练[ 10 ])。
- 第三,3D卷积和其他减少三维卷积的密集计算规避解决方案,(如三平面方案或Pyramid - LSTM),在各向异性3D图像上使用各向同性内核执行2D卷积可能是有问题的,特别是对于深度分辨率(z轴)明显较低的图像。例如,三平面方案和Pyramid-LSTM都在xz和yz上执行2D卷积。对于xz平面中的两个正交的线,一个沿z方向和一个 另一个沿着x方向,它们可以对应于两个非常不同尺度的结构,并且 因此可以对应于不同类型的对象 - 或者甚至可以不对应感兴趣的物体。 但是,具有各向同性内核的xz平面上的2D卷积是不能的区分这两行。 另一方面,如果以3D旋转,相同类型的3D对象可能具有 在xz或yz平面上有非常不同的外观。 这个事实使得这些2D提取的特征成为可能 xz或yz平面中的各向同性卷积具有较差的通用性(例如,可能导致过度拟合)。
研究方法
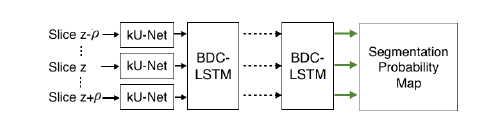
模型架构如上图所示。第一个部分为FCN部分,第二个部分为RNN部分。
设计的FCN组件
采用多个FCN(本文用U-Net),在不同的图像尺度上工作,构成新架构
kU-Net。参考人类专家标记的行为:首先缩小图像 找出目标对象的位置,然后放大以标记准确的边界 那些目标(其实也就是通过卷积提取特征,再反卷积输出)。 kU-Net的两个关键机制来模拟这种行为:
- 使用一系列子模块FCN来顺序提取不同比例的信息 (从最粗规模到最精细规模)。
- 子模块FCN提取的信息 负责较粗的规模将传播到随后的子模块FCN来协助 特征提取更精细。

文中展示了4个kU_Net的连接方式,实验表明A,D效果较好。且多U_Net性能优于单一U_Net。
设计RNN组件
所用的网络为BDC-LSTM:将CLSTM网络扩展到双向卷积。一个在z -方向,另一个在z +方向。避免了Pyramid-LSTM6个方向消耗内存的情况。该网络的详细结构为:
输入(64×126×126),p = 0.5的dropout层,两个 BDC-LSTM具有64个隐藏单元和5×5 kernel(64×118×118),2×2最大池(64×59×59), p = 0.5的dropout层,两个BDC-LSTM具有64个隐藏单元和5×5个kernel(64×51×51),2×2 反卷积(64×102×102),p = 0.5的dropout层,3×3卷积层,(64×100×100),1×1卷积层,(2×100×100)。 (注意:BDC-LSTM中的所有卷积使用与层中指示的相同的kernel大小。)因此, 为了预测100×100区域的概率图,我们需要以126×126的输入,且中心在相同的位置。 因为深BDC-LSTM沿z方向完全卷积,在评估阶段overlapping-tile策略 [16 ],。 假设整个切片的特征图的大小为64×W×H。 输入张量将填充 边框上的零调整为64×(W + 26)×(H +26)。 然后,序列为64×126×126 每次都会处理补丁。 缝合结果以形成3D分割。

训练策略
可以端到端训练,也可以分别训练kU-Net和BDC-LSTM。每次迭代中随机选择一个训练样例进行旋转、翻转、镜像进行数据增强。为避免梯度爆炸,每次迭代将梯度剪裁为【-5,5】;损失函数使用加权交叉熵损失。
结果

参考文献
[1] A. Cardona, S. Saalfeld, S. Preibisch, B. Schmid, A. Cheng, J. Pulokas, P. Tomancak, and V. Hartenstein.An integrated micro-and macroarchitectural analysis of the drosophila brain by computer-assisted serialsection electron microscopy. PLoS Biol, 8(10):e1000502, 2010.
[2] H. Chen, X. Qi, L. Yu, and P.-A. Heng. Dcan: Deep contour-aware networks for accurate gland segmentation.arXiv preprint arXiv:1604.02677, 2016.
[3] H. Chen, X. J. Qi, J. Z. Cheng, and P. A. Heng. Deep contextual networks for neuronal structuresegmentation. In AAAI Conference on Artificial Intelligence, 2016.
[4] D. Ciresan, A. Giusti, L. M. Gambardella, and J. Schmidhuber. Deep neural networks segment neuronalmembranes in electron microscopy images. In NIPS, pages 2843–2851, 2012.
[5] R. Collobert, K. Kavukcuoglu, and C. Farabet. Torch7: A Matlab-like environment for machine learning.In BigLearn, NIPS Workshop, 2011.
[6] Y. N. Dauphin, H. de Vries, J. Chung, and Y. Bengio. Rmsprop and equilibrated adaptive learning rates for
non-convex optimization. arXiv preprint arXiv:1502.04390, 2015.
[7] K. He, X. Zhang, S. Ren, and J. Sun. Delving deep into rectifiers: Surpassing human-level performance on imagenet classification. In CVPR, pages 1026–1034, 2015.
(参考初始化策略)
[8] S. Hochreiter and J. Schmidhuber. Long short-term memory. Neural Computation, 9(8):1735–1780, 1997.
[9] D. Kingma and J. Ba. Adam: A method for stochastic optimization. arXiv preprint arXiv:1412.6980, 2014.
[10] M. Lai. Deep learning for medical image segmentation. arXiv preprint arXiv:1505.02000, 2015.
[11] K. Lee, A. Zlateski, V. Ashwin, and H. S. Seung. Recursive training of 2D-3D convolutional networks for
neuronal boundary prediction. In NIPS, pages 3559–3567, 2015.
[12] N. Léonard, S.Waghmare, and Y.Wang. rnn: Recurrent library for Torch. arXiv preprint arXiv:1511.07889,2015.
[13] J. Long, E. Shelhamer, and T. Darrell. Fully convolutional networks for semantic segmentation. In CVPR,
pages 3431–3440, 2015.
[14] V. Patraucean, A. Handa, and R. Cipolla. Spatio-temporal video autoencoder with differentiable memory.
arXiv preprint arXiv:1511.06309, 2015.
[15] A. Prasoon, K. Petersen, C. Igel, F. Lauze, E. Dam, and M. Nielsen. Deep feature learning for knee
cartilage segmentation using a triplanar convolutional neural network. In MICCAI, pages 246–253, 2013.
[16] O. Ronneberger, P. Fischer, and T. Brox. U-Net: Convolutional networks for biomedical image segmentation.
(生物医学图像太大的解决方案)
In MICCAI, pages 234–241, 2015.
[17] X. Shi, Z. Chen, H. Wang, D.-Y. Yeung, W.-K. Wong, and W. chun Woo. Convolutional lstm network: A
machine learning approach for precipitation nowcasting. arXiv preprint arXiv:1506.04214, 2015.
[18] M. F. Stollenga, W. Byeon, M. Liwicki, and J. Schmidhuber. Parallel multi-dimensional LSTM, with
application to fast biomedical volumetric image segmentation. In NIPS, pages 2980–2988, 2015.
[19] C. Sun, M. Paluri, R. Collobert, R. Nevatia, and L. Bourdev. Pronet: Learning to propose object-specific
boxes for cascaded neural networks. arXiv preprint arXiv:1511.03776, 2015.
9
(解决图像多尺度问题)






















 1887
1887











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








