目录
事先准备
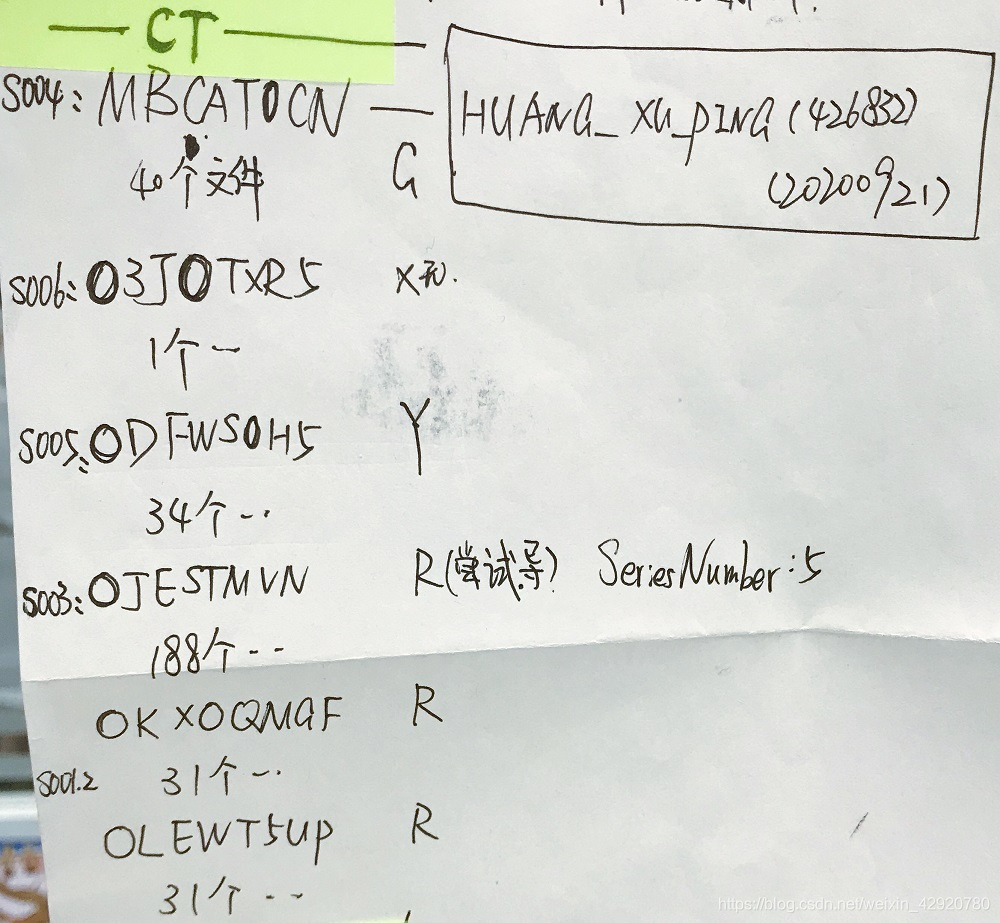
- 数据:植入电极前的MRI(20年4月)、植入电极后的CT(20年9月)

- 软件:ContactSegmentation_SYSU、3D Slicer
操作步骤
声明:一定要看“User Manuals for ContactSegmentation_SYSU"和fsppt
一、图像数据预准备

一开始所给数据没带.dcm后缀,想用3D Slicer export一遍(export的路径不能带中文),后来重新给的MRI就是dcm格式的,只需将文件夹重命名为“MR_T1”就好
1.1.1 import带目标dicom的文件夹
1.1.2 load目标series

1.1.3 在“data”Module下导出series
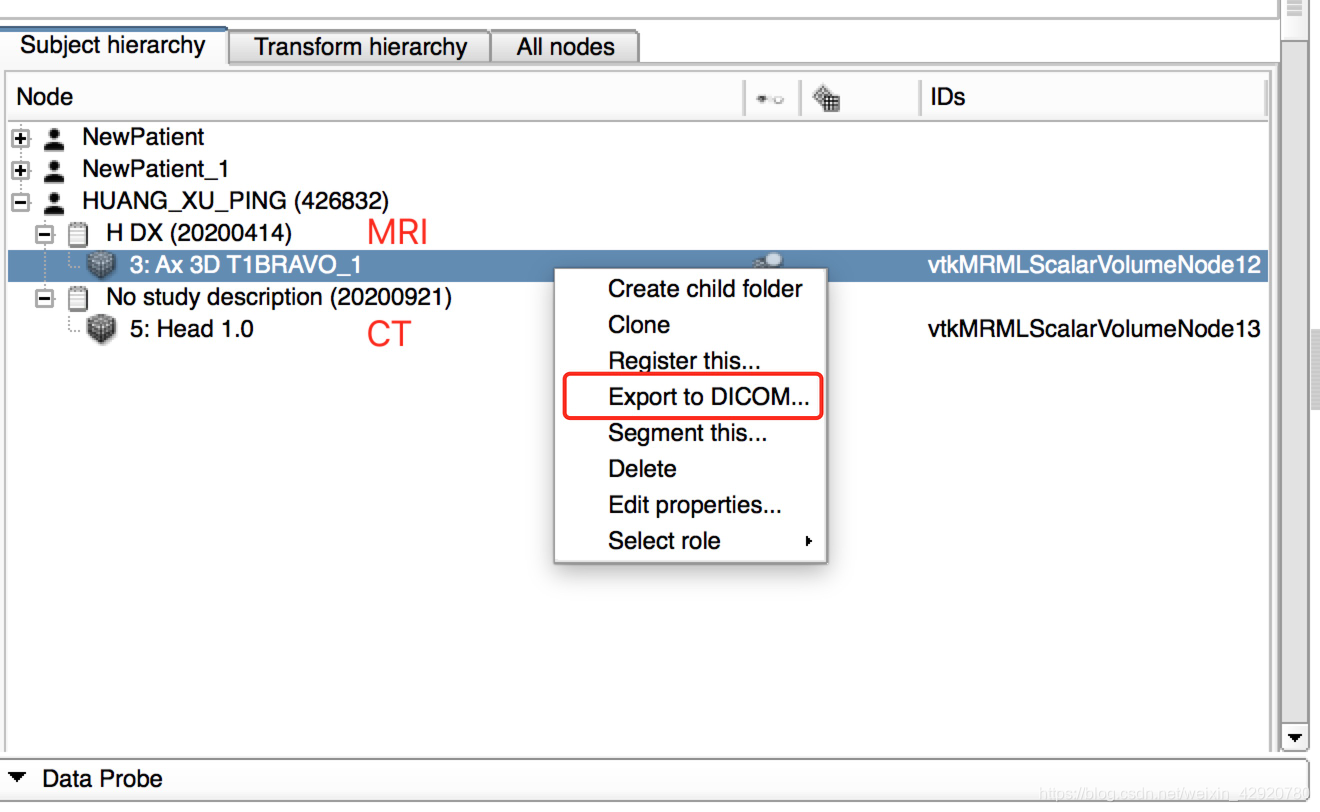

![]()
数据没带.dcm后缀,解决方法一:用3D Slicer export一遍,变成dicom(export的路径不能带中文),和1.1类似;方法二:将文件后缀加上.dcm,就可以变成dicom格式
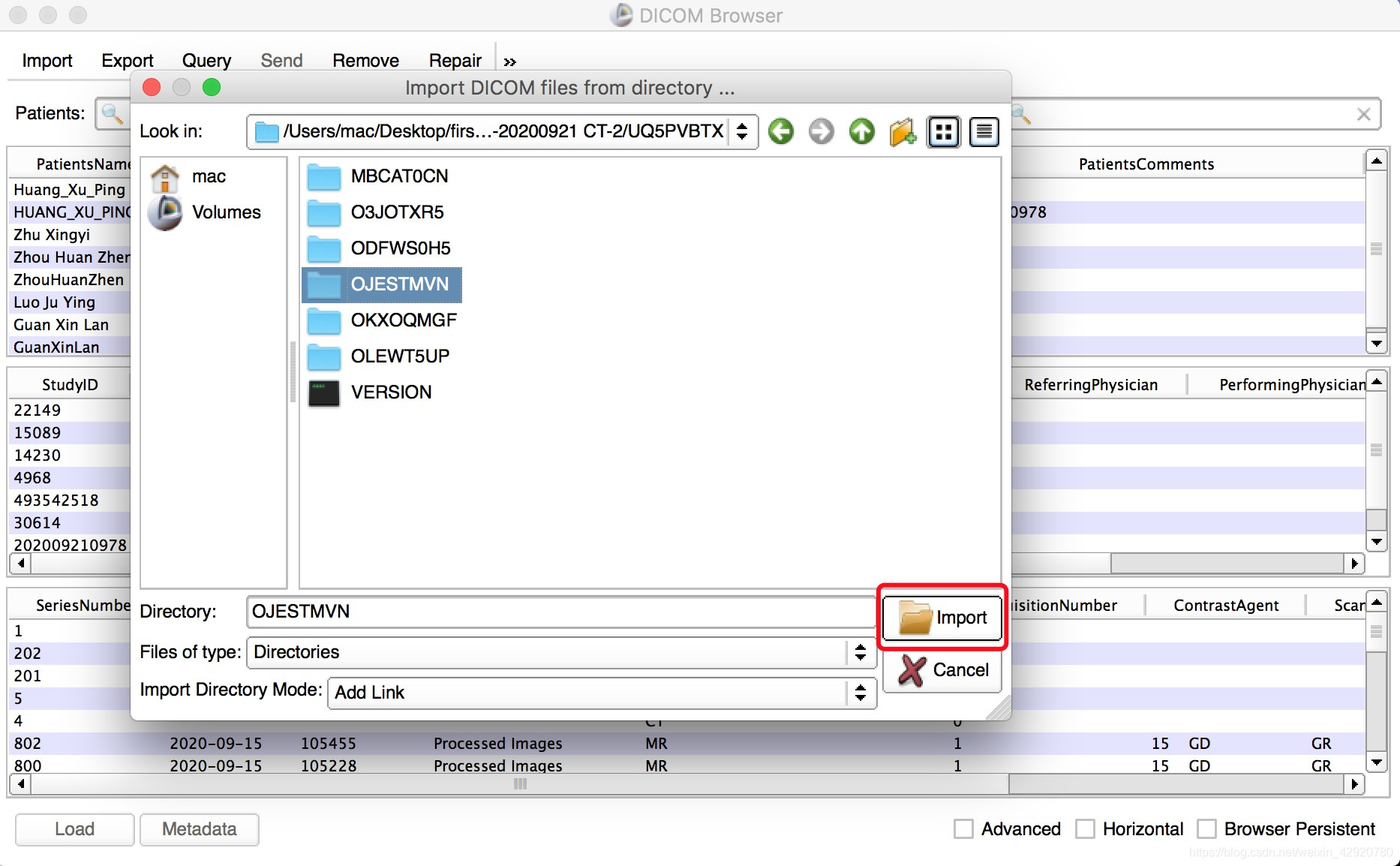
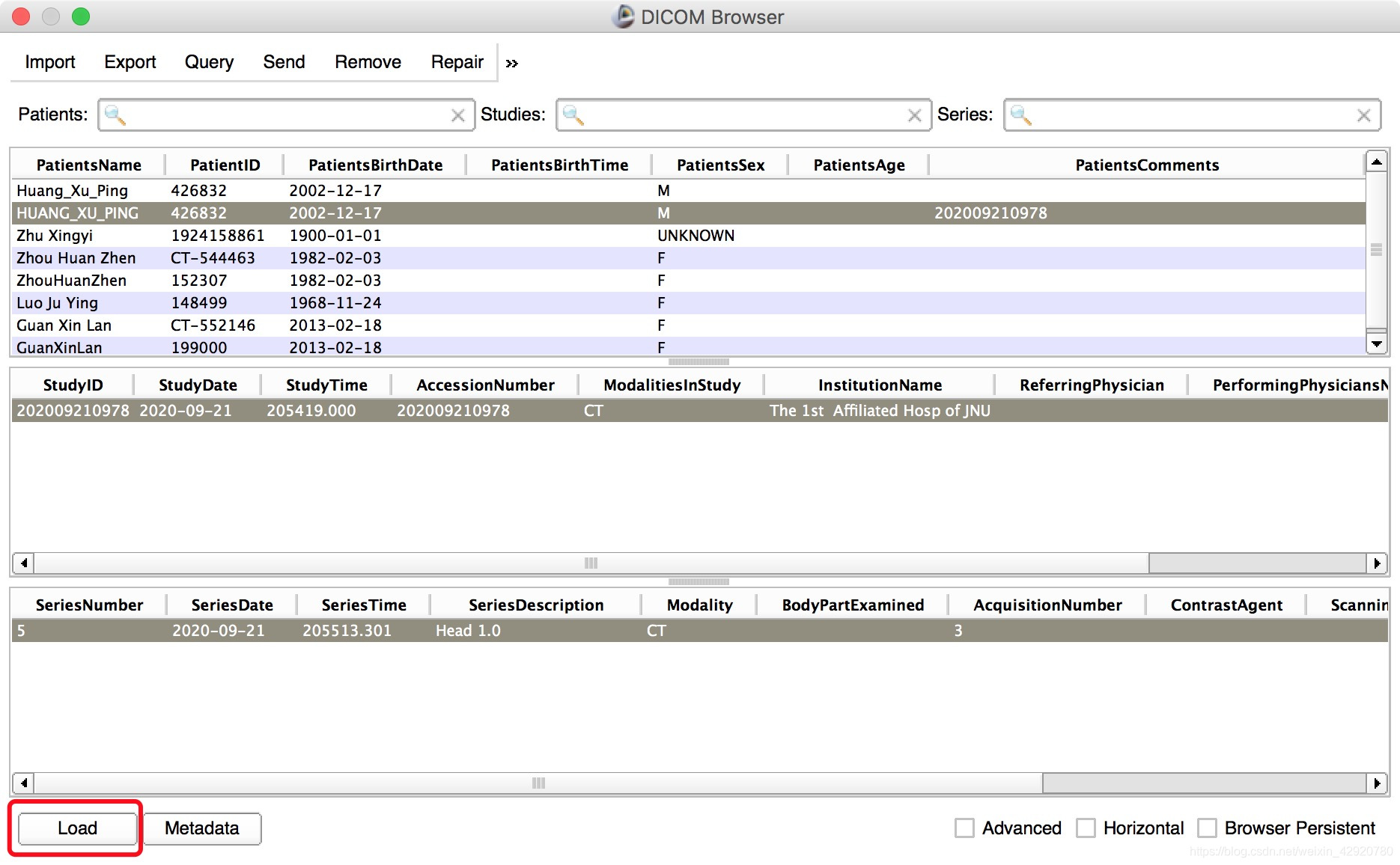

1.3.1 FreeSurfer入门(将.dcm→brainmask.mgz)
看PPT:freesurfer--第10次--用Freesurfer进行预处理1.0--page10
过程:
1、找到freesurfer的工作目录。
打开Terminal,输入命令:
cd $SUBJECTS_DIR用pwd检查当前工作目录;

2、手动找到mysubjectdata文件夹,并在里面新建文件夹“huang_xuping",然后将“MR_T1"文件夹放入“huang_xuping"
3、Terminal输入命令:
cd huang_xupingdcmunpack -src . -scanonly huang.logdcmunpack:意为“文件打包”,表示对MR_T1的dicom序列进行打包整理,生成一个文本文件,筛选出符合freesurfer输入要求的dicom文件。
-src:是“source”是所写,表示需要访问的源文件,后面接“.”或者完整路径。
. :表示指定当前dicom文件所在路径。
-scanonly scan.log:表示浏览并生成文本文件scan.log,“scan.log”是文件名称,可以自行更改。
上述命令用于筛选出符合freesurfer输入要求的dicom文件,将用时2-5分钟,耐心等待,不能关闭终端窗口。

4、进入MR_T1文件夹,再接着进入ScalarVolume_17文件夹:
cd MR_T1
cd ScalarVolume_17再输入命令:
recon-all -all -i IMG0001.dcm -s surf_huang
第二个‘-all’表示进行全部的重建过程; ‘IMG0001.dcm’表示符合要求的dcm文件之一;‘surf_huang’表示重建结果的文件夹名称
等待8-30个小时即可得到重建结果,结果在“surf_huang”中

结果在surf_huang文件夹中会生成八个子文件夹:label、mri、scripts、stats、surf、tmp、touch、trash ,如下图所示:

各个文件夹的包含关系如下:

1.3.2 将得到的brainmask.mgz export 成‘FsBrainmask’
brainmask.mgz位于mri文件夹:
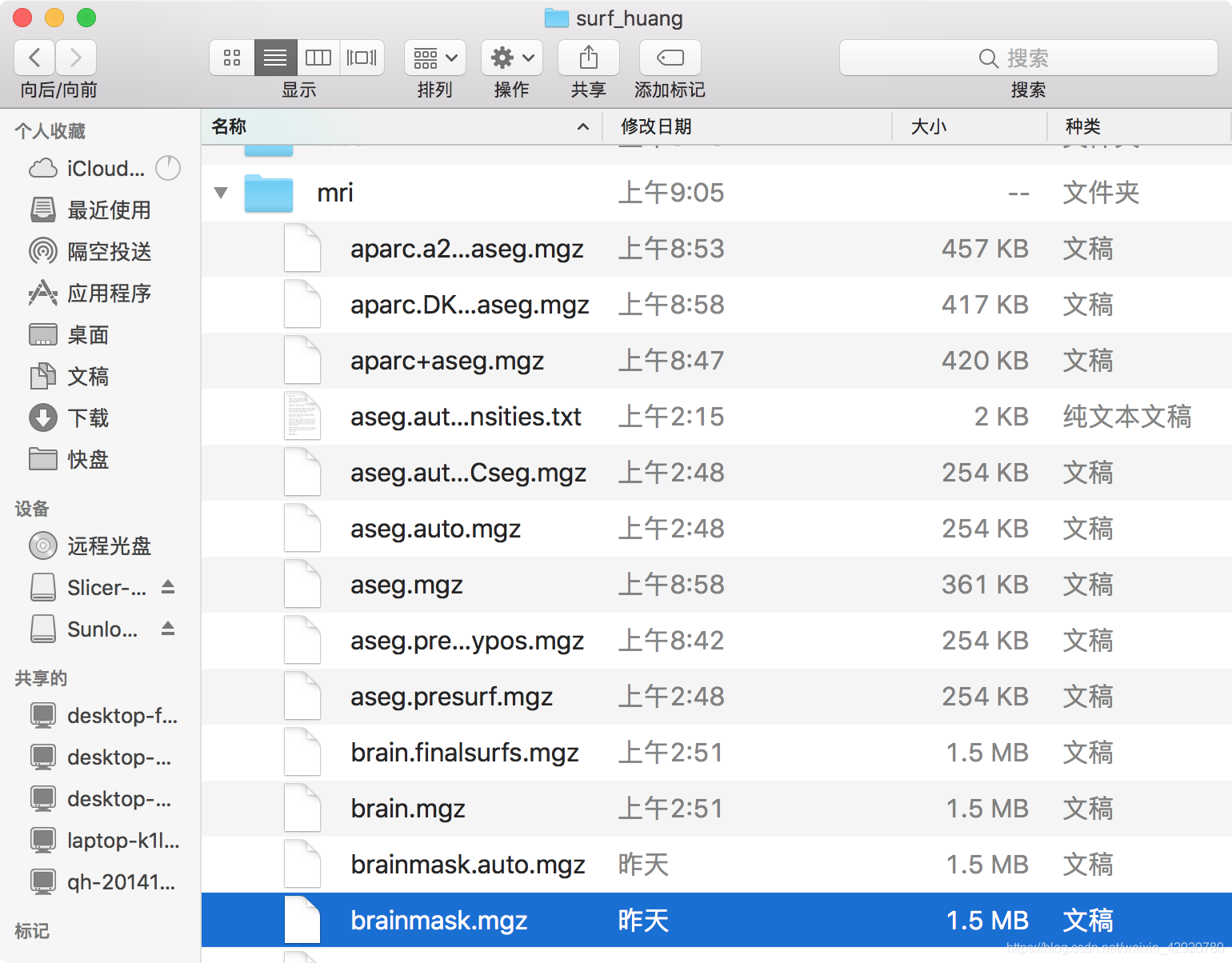
打开3D Slicer,从“data” load brainmask.mgz


再export成dicom格式:

二、数据导入及预准备
- 所用的软件为ContactSegmentation_SYSU
- Mac所用数据存放位置 MR_T1:FirstLYL→Presurgery-T1202004A→MR_T1 CT:FirstLYL→CT FsBrainmask:FirstLYL→FsBrainmask
2.1 数据导入与预处理
2.1.1 选择dicom类型,导入MR_T1(MR_T1里面应该直接是dicom文件!!!不能有ScalarVolume!!!否则无法配准!!!其他文件夹的导入同理)

2.1.2 先将导入的dicom进行最近邻插值,对三维体数据进行重建。再进行RAS坐标变换

2.1.3 对CT和brainmask.mgz分别重复2.1.1-2.1.2
2.1.4 选择nii类型,导入MNI标准脑文件avg152T1.nii,重复2.1.2

2.2 图像配准
2.2.1 将CT配准到MRI上
Fixed Volume: MR_T1
Moving Volume: CT
InitialRadius: 0.005
MaxInterations: 1000

2.2.2 将FsBrainmask(大脑掩膜)配准到regCT(已配准的CT)上
Fixed Volume: regCT
Moving Volume: FsBrainmask
InitialRadius: 0.005
MaxInterations: 1000

2.2.3 将MRI配准到标准脑上
Fixed Volume: avg152T1
Moving Volume: MR_T1
InitialRadius: 0.0001
MaxInterations: 3000
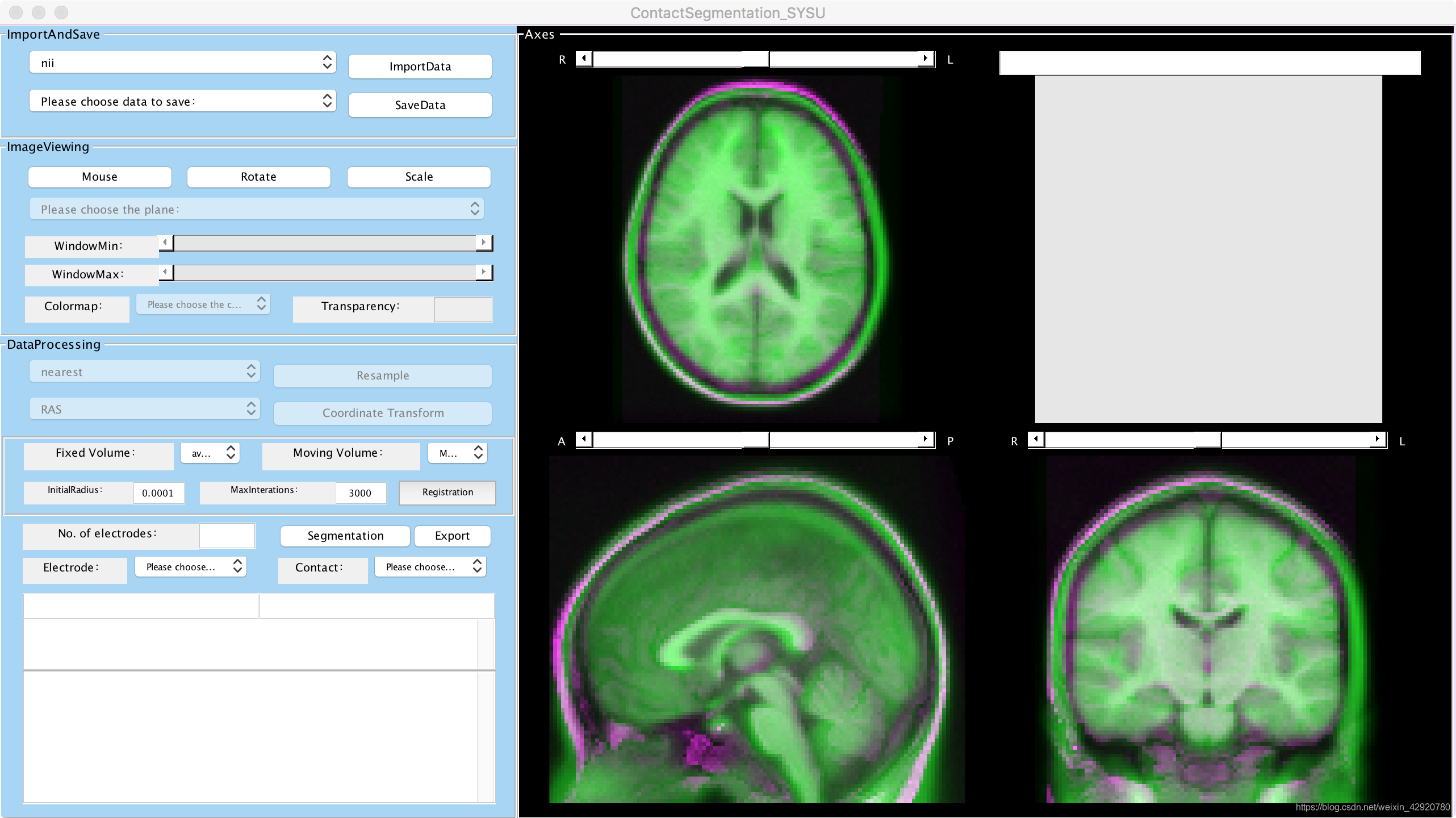
三、分割电极
输入待分割的电极数,并点“segmentation”,等半天就会出电极分割的结果了,还有分割过程的数据

补充知识
括号内的是3D Slicer界面的各平面简称
1.冠状面(R)Coronal section,又称额状面。从左右方向,沿人体的长轴将人体纵切为前、后两部分的切面。这种提法只是为了在临床中将器官位置描述的更具体
2.矢状面(Y)Sagittal section,把人体分成左右两面的解剖面。
3.水平位(G)Transverse section,又称横断位,即左右、前后构成的面为水平位


























 3万+
3万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








