序
非负矩阵分解(NMF)是一种无监督学习算法,其目的在于提取有用的特征。它的工作原理类似于PCA,也可以用于降维。与PCA相同,我们试图将每个数据点写成一些分量的加权求和。但在PCA中,我们想要的是正负分量,并且能够解释尽可能多的数据方差;而在NMF中,我们希望分量和系数均为负,也就是说,我们希望分量和系数都大于或等于0。因此,NMF只能应用于每个特征都是非负的数据,因为非负分量的非负求和不可能变为负值。
将数据分解成非负加权求和的这个过程,对由多个独立源相加(或叠加)创建而成的数据特别有用,比如多人说话的音轨或包含很多乐器的音乐。在这种情况下,NMF可以识别出组合成数据的原始分量。总的来说,与PCA相比,NMF得到的分量更容易解释,因为负的分量和系数可能会导致难以解释的抵消效应。
PCA原理传送门:无监督学习与主成分分析(PCA)
接下来,我们将NMF应用于人脸识别。
NMF实际应用
1.数据源
数据是之前我们已经处理好的人脸图像数据,一共有15个人物,每个人有10张头像。想了解具体处理过程的可以去看一下主成分分析(PCA)应用——特征提取_人脸识别(上)。
提数代码如下:
import os
all_folds = os.listdir(r'C:\Users\Administrator\Desktop\源数据-分析\lfw_funneled')###https://www.kaggle.com/atulanandjha/lfwpeople?select=pairs.txt
all_folds = [x for x in all_folds if '.' not in x]
import pandas as pd
numbers_img=pd.DataFrame(columns=["文件名称","图片数量"])####统计各个文件夹里面的图片数量
for i in range(len(all_folds)):
path = 'C:\\Users\\Administrator\\Desktop\\源数据-分析\\lfw_funneled\\'+all_folds[i]
all_files = os.listdir(path)
numbers_img.loc[i]=[all_folds[i],len(all_files)]
img_10=numbers_img[numbers_img["图片数量"]==10].reset_index()#####为了降低数据偏斜,选取图片数量为10的文件(否则,特征提取会被图片数量过多的数据影响)
from PIL import Image
import numpy as np
image_arr_list=[]###存放灰度值numpy数组
flat_arr_list=[]###存放灰度值一维数组
target_list=[]###存放目标值
for m in range(len(img_10["文件名称"])):
file_address='C:\\Users\\Administrator\\Desktop\\源数据-分析\\lfw_funneled\\'+img_10["文件名称"][m]+"\\"####指定特定的文件地址
image_name=os.listdir(file_address)###获得指定文件夹下的左右文件名称
for n in image_name:
image=Image.open(file_address+n)
image=image.convert('L')###RGB(红绿蓝)像素值转换成灰度值
image_arr=np.array(image,"f")###灰度值转化成numpy数组(二维)
flat_arr=image_arr.ravel()###将数组扁平化处理,返回的是一个一维数组的非副本视图,就是将几行的数据强行拉成一行
image_arr_list.append(image_arr)
flat_arr_list.append(flat_arr)
target_list.append(m)###这里的m设定是数字,如果是文本的话后面的算法会报错
faces_dict={"images":np.array(image_arr_list),"data":np.array(flat_arr_list),"target":np.array(target_list)}
2.建模
提取完数据集之后,我们划分数据集为训练集和测试集,并用核向量算法SVM来进行建模和评估。
SVM算法讲解传送门:支持向量机(SVM)算法之补充说明
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
from sklearn.svm import SVC
train=faces_dict["data"]/255
X_train,X_test,y_train,y_test=train_test_split(train,faces_dict["target"],random_state=0)###划分训练集和测试集
clf = SVC(kernel="linear",random_state=0)
clf.fit(X_train, y_train)#训练
y_predict = clf.predict(X_test)#预测
print(accuracy_score(y_test, y_predict))#评分
结果如下:
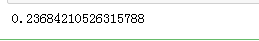
这样,我们就得到了一个精度为23.6%的模型。
3.NMF处理
NMF的主要参数是我们想要提取的分量个数——n_components。通常来说,这个数字要小于输入特征的个数(否则的话,将每个像素作为单独的分量就可以对数据进行解释)。代码如下:
from sklearn.decomposition import NMF
nmf=NMF(n_components=20,random_state=0,max_iter=10000).fit(X_train)###增加最大迭代次数,不然会预警
X_train_nmf=nmf.transform(X_train)
X_test_nmf=nmf.transform(X_test)
clf=SVC(kernel="linear",random_state=0)
clf.fit(X_train_nmf,y_train)#训练
clf_predict=clf.predict(X_test_nmf)
print(accuracy_score(y_test,clf_predict))
需要注意的是,如果特征值过多的话,NMF默认的迭代次数便会限制模型的精度,并且预警。所以我们还需要设立下NMF的最大迭代次数。最后得到的结果如下:
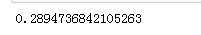
通常来说,提取的特征越多,即n_components越大,模型的精度就越高,但是模型的训练时间也就越长。这里我就不再继续尝试了,感兴趣的朋友们可以试试改变n_components的值来提高模型精度。
与PCA的比较
PCA对于数据特征的处理是找到特征重建的最佳方向。而NMF通常并不用于对数据进行重建或者编码。而是寻找用于数据中的有趣的模式。正如我们之前提到的一样,NMF最适合于具有叠加结构的数据,包括音频,基因表达和文本数据。接下来我们通过一段模拟信号来与PCA比较一下。
假设有一段信号,它是由三个不同的信号源组成的,代码如下:
import mglearn
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif'] = ['SimHei']###防止中文显示不出来
plt.rcParams['axes.unicode_minus'] = False###防止坐标轴符号显示不出来
S=mglearn.datasets.make_signals()
plt.figure(figsize=(10,1))
plt.plot(S,"_")
plt.xlabel("Time")
plt.ylabel("Signal")
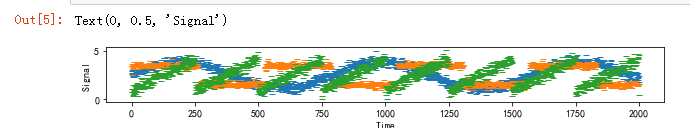
不幸的是,我们无法观测到原始信号,只能观测到三个信号的叠加混合。而我们的目的便是将混合信号分解成原信号。假设我们有100台测量装置,每个测量装置都为我们提供了一系列测量结果。所以接下来我们将数据混合成100维的状态:
A=np.random.RandomState(0).uniform(size=(100,3))###假设有100台装置测量混合信号
X=np.dot(S,A.T)###将数据混合成100维的状态
接下来,我们分别用NMF和PCA来还原这三个信号:
from sklearn.decomposition import PCA
pca=PCA(n_components=3)
H=pca.fit_transform(X)
nmf=NMF(n_components=3,max_iter=10000,random_state=0)
S_=nmf.fit_transform(X)
最后,我们将结果画出来:
models=[X,S,S_,H]
names=["观测信号","真实信号","非负矩阵(NMF)还原信号","主成分分析(PCA)还原信号"]
fig,axes=plt.subplots(4,figsize=(10,10),gridspec_kw={"hspace":1},subplot_kw={"xticks":(),"yticks":()})
plt.figure(figsize=(6,1))
for model,name,ax in zip(models,names,axes):
ax.set_title(name)
ax.plot(model[:,:3],"_")

可以看到,NMF在发现原始信号源是得到了不错的成果,而PCA的表现却很差,仅使用第一个成分来解释数据中的大部分变化。
这里需要注意的是,NMF生成的分量是没有顺序的。在上面的例子中,NMF分量的顺序与原始信号完全相同(可以从三条线的颜色看出来),但这纯属偶然。
总结
除了PCA和NMF之外,还有许多算法可用于将每个数据点分解为一系列固定分量的加权求和。通常描述对分量和系数的约定会涉及到概率论。如果朋友们对这种类型的模式感兴趣,可以去看下scikit-learn中关于独立成分分析(ICA),因子分析(FA)和稀疏编码(字典学习)等。
个人博客:https://www.yyb705.com/
欢迎大家来我的个人博客逛一逛,里面不仅有技术文,也有系列书籍的内化笔记。
有很多地方做的不是很好,欢迎网友来提出建议,也希望可以遇到些朋友来一起交流讨论。






















 2588
2588











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








