在发表科研论文的过程中,科研绘图是必不可少的,一张好看的图形会是文章很大的加分项。
为了便于使用,本系列文章介绍的所有绘图都已收录到了 sciRplot 项目中,获取方式:
本期绘图预览:

1. 导入包
我们首先导入本期绘图用到的 R 包:
library(ggplot2)2. 准备数据
接下来我们导入绘图用到的数据,在 sciRplot 中给大家提供了测试数据:
data <- sciRplot_data3. 准备配色
颜色的选择往往是一件让人特别纠结的事情,这里我们直接使用 sciRcolor 来设置配色:
colors <- sciRcolor::pal_scircolor(37)[1:5]sciRcolor 是为了 R 语言科研绘图开发的配色工具,包含了 100 种常用配色,详细信息见:
4. 绘制图形
接下来我们通过下面的代码来绘制图形:
p <-ggplot(data, aes(x="", y=Value, fill=Type)) +geom_bar(stat="identity", width=1, color="white", position = position_stack(reverse =T)) +geom_text(aes(x=1.1, label=Value), color="white", size=10, position = position_stack(reverse =T, vjust=0.5)) +coord_polar("y", start=0) +theme_void(base_size = 25) +theme(legend.position = "top") +scale_fill_manual(values = colors)p
5. 保存图形
最后我们保存绘制的图形:
ggsave("save/pie-base.png", p, width = 8, height = 6, dpi = 300)sciRplot 介绍
为了解决 R 语言中科研绘图的问题,我推出了 sciRplot 项目。sciRplot 项目包含了以下内容:
① 100 种绘图代码,按照图形类型进行分类,包含 60 种基础绘图和 40 种进阶绘图

② 配备一站式 html文档,包含测试数据,代码一键复制,交互式阅读提高用户体验
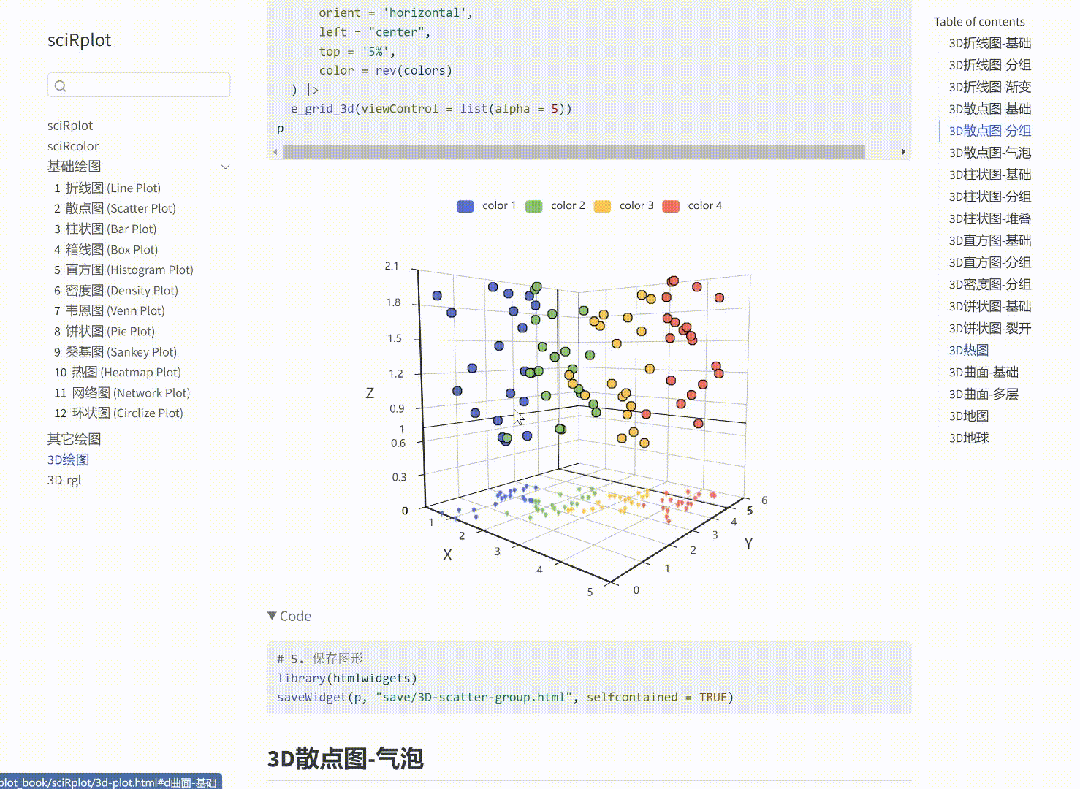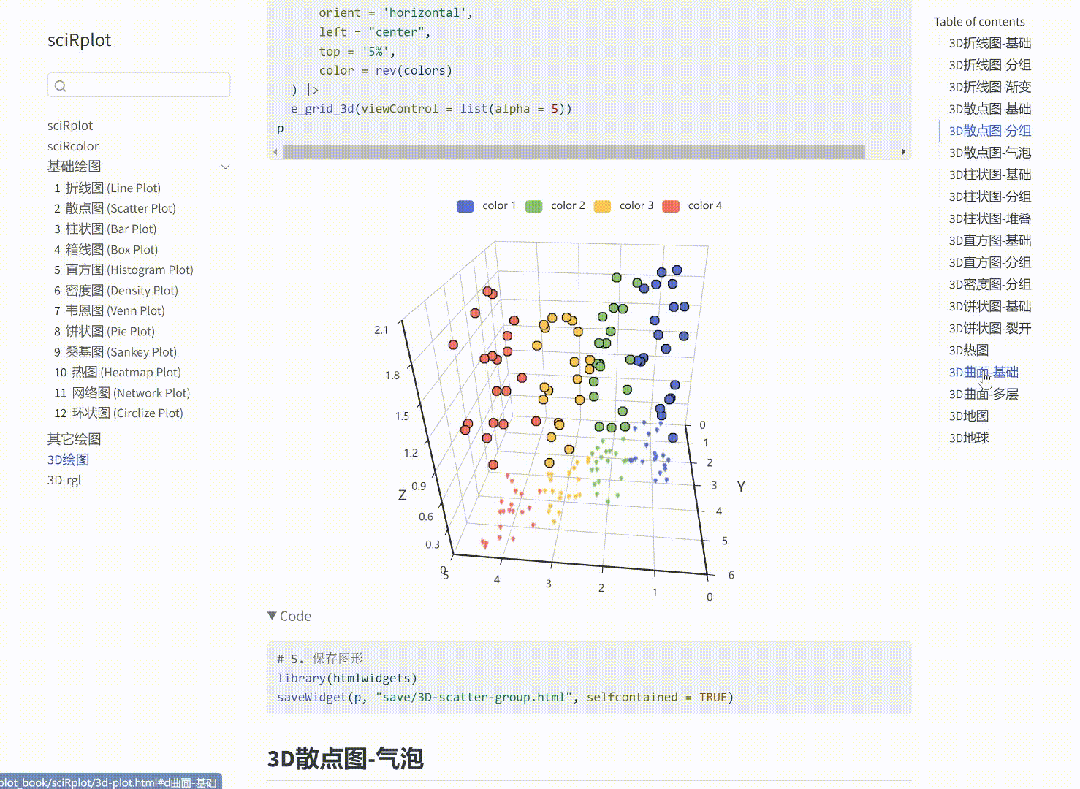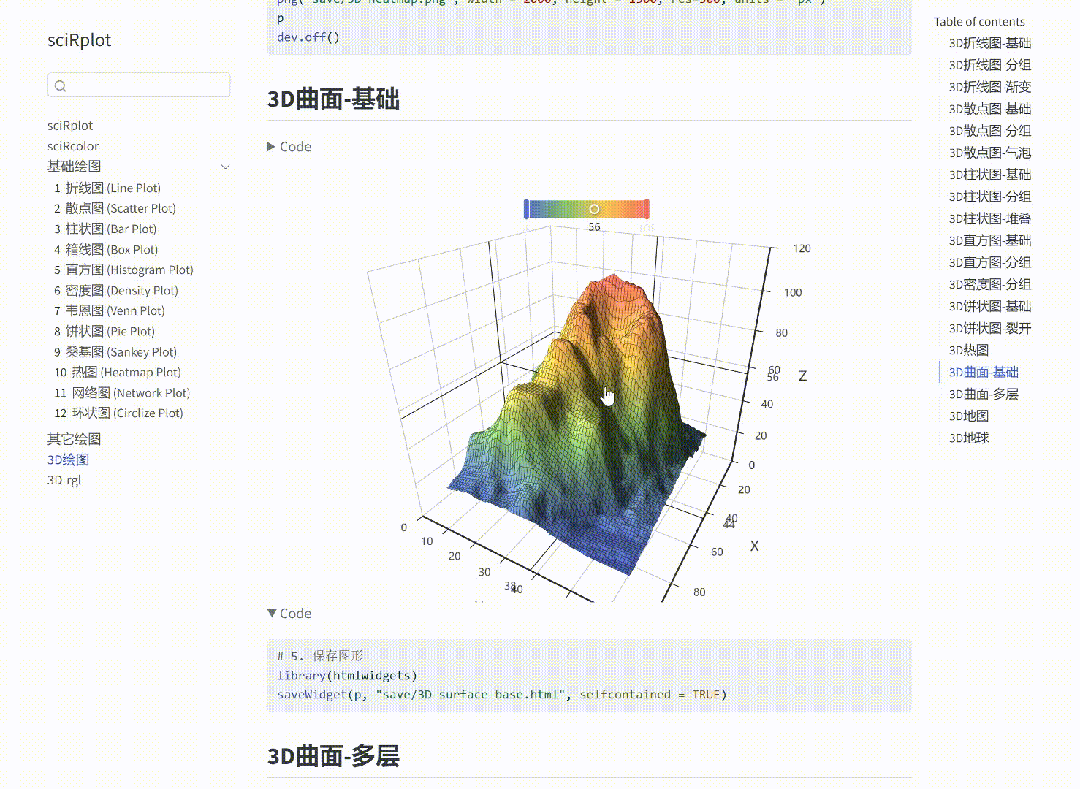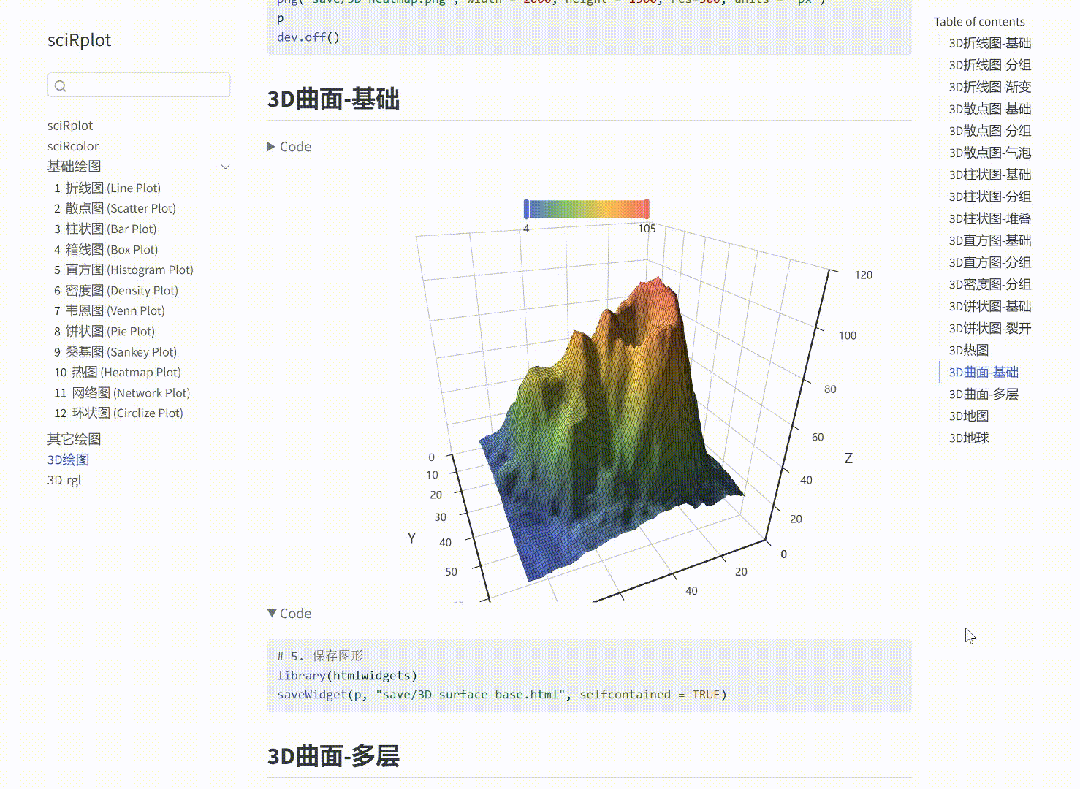
























 1474
1474

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










