复现内容
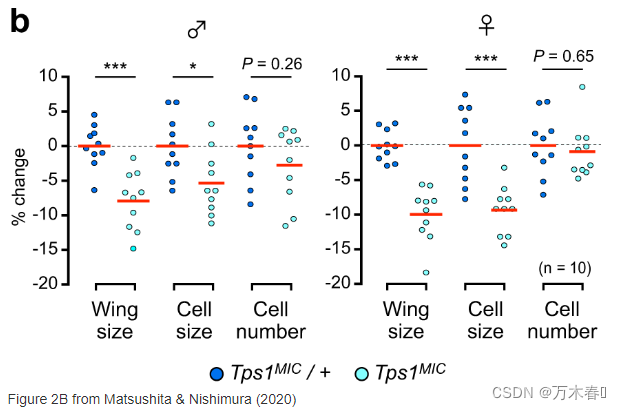
下图,来自2020年发表在Communications Biology杂志上的文章:Trehalose metabolism confers developmental robustness and stability in Drosophila by regulating glucose homeostasis,这里复现文章中图二的B图。
准备
PS:需要的工具需要自行安装
library(dplyr)
library(ggplot2)
library(ggprism)
library(ggbeeswarm)
library(rstatix)
data("wings")
# 在分析或者可视化之前,一定要了解数据内容和结构
head(wings)
# A tibble: 6 x 4
sex genotype measure percent.change
<fct> <fct> <fct> <dbl>
1 male Tps1MIC/+ wing.size -2.45
可以看到数据已经是“长格式”(不了解的可以搜索:R 数据长宽转换),进行简单的修改之后就可以直接画图:
# 首字母改成大写,把“.”换成空格
wings$measure <- wings$measure %>%
gsub("\\.", " ", .) %>%
tools::toTitleCase() %>%
factor(., levels = c("Wing Size", "Cell Size", "Cell Number"))
可视化
粗画
p <- ggplot(wings, aes(x = measure, y = percent.change))
p <- p + ggbeeswarm::geom_beeswarm(
aes(fill = genotype),
dodge.width = 0.9,
shape = 21,
cex = 3.5
)
p

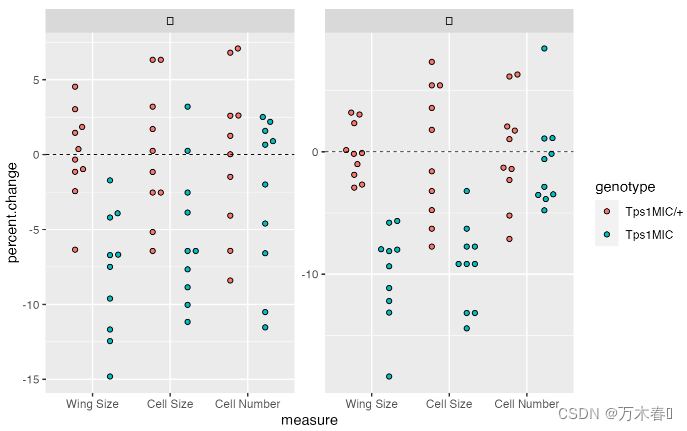
分面
按照性别分面:
p <- p + facet_wrap(
~ sex,
scales = "free",
labeller = labeller(sex = c(male = "\u2642", female = "\u2640"))
)
p

添加辅助线
使用geom_hline(),添加虚线:y = 0:
p <- p + geom_hline(yintercept = 0, linetype = 2, size = 0.3)
p
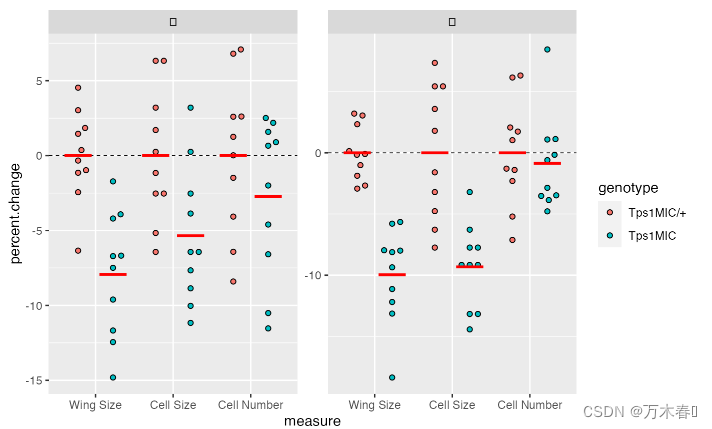
添加均值
使用 geom_crossbar(), 给每组数据添加均值:
p <- p + stat_summary(
geom = "crossbar",
aes(fill = genotype),
fun = mean,
position = position_dodge(0.9),
colour = "red",
size = 0.4, width = 0.7,
show.legend = FALSE
)
p
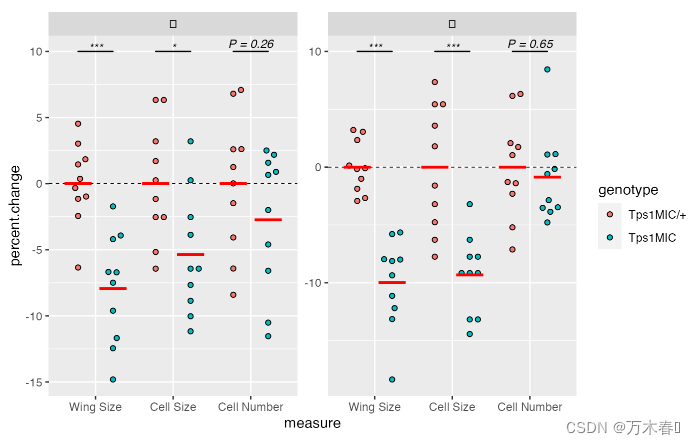
添加显著性标记
统计分析并BH矫正
wings_pvals <- wings %>%
group_by(sex, measure) %>%
rstatix::t_test(
percent.change ~ genotype,
p.adjust.method = "BH",
var.equal = TRUE,
ref.group = "Tps1MIC/+"
) %>%
rstatix::add_x_position(x = "measure", dodge = 0.9) %>% # dodge must match points
mutate(label = c("***", "*", "P = 0.26", "***", "***", "P = 0.65"))
wings_pvals
# A tibble: 6 x 14
sex measure .y. group1 group2 n1 n2 statistic df p x
<fct> <fct> <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl>
1 male Wing Si… perce… Tps1M… Tps1M… 10 10 4.85 18 1.29e-4 1
使用add_pvalue添加显著性标记:
p <- p + add_pvalue(
wings_pvals, y = 10, xmin = "xmin", xmax = "xmax", tip.length = 0,
fontface = "italic", lineend = "round", bracket.size = 0.5
)
p
微调
主题
使用theme_prism主题:
p <- p + theme_prism(
base_fontface = "plain",
base_line_size = 0.7,
base_family = "Arial"
)
p
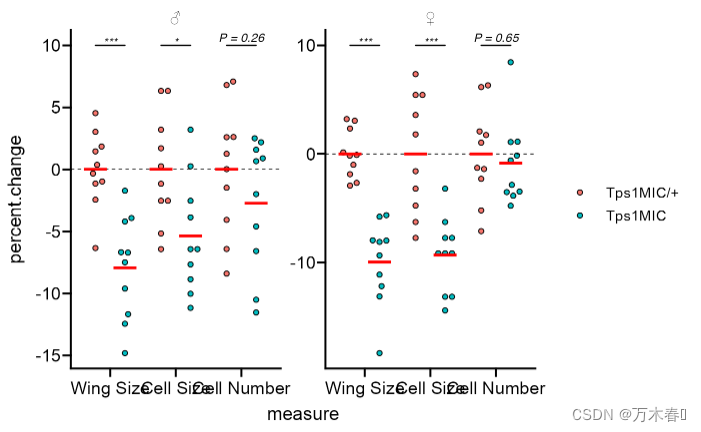
坐标轴形式
p <- p + scale_x_discrete(
guide = guide_prism_bracket(width = 0.15),
labels = scales::wrap_format(5)
) + scale_y_continuous(
limits = c(-20, 12),
expand = c(0, 0),
breaks = seq(-20, 10, 5),
guide = "prism_offset"
) + labs(y = "% change")
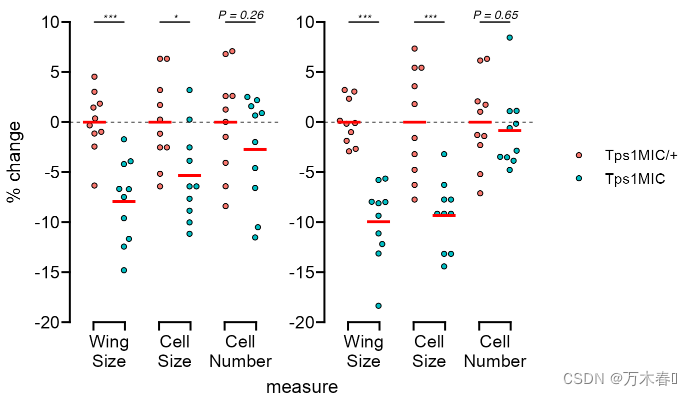
图例样式和位置
先搞定上标
p <- p + scale_fill_manual(
values = c("#026FEE", "#87FFFF"),
labels = c(expression("Tps"*1^italic("MIC")~"/ +"),
expression("Tps"*1^italic("MIC")))
)
调整图例位置
p <- p + theme(
legend.position = "bottom",
axis.title.x = element_blank(),
strip.text = element_text(size = 14),
legend.spacing.x = unit(0, "pt"),
legend.text = element_text(margin = margin(r = 20))
)
p
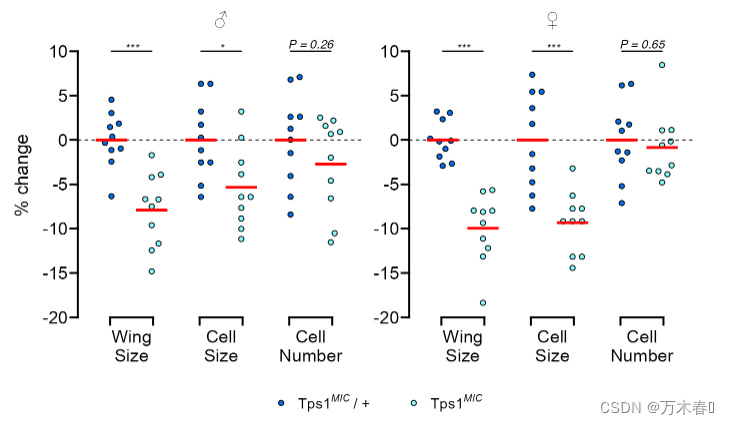
右下角添加 n=10
p <- p + geom_text(
data = data.frame(
sex = factor("female", levels = c("male", "female")),
measure = "Cell Number",
percent.change = -18.5,
lab = "(n = 10)"
),
aes(label = lab)
)
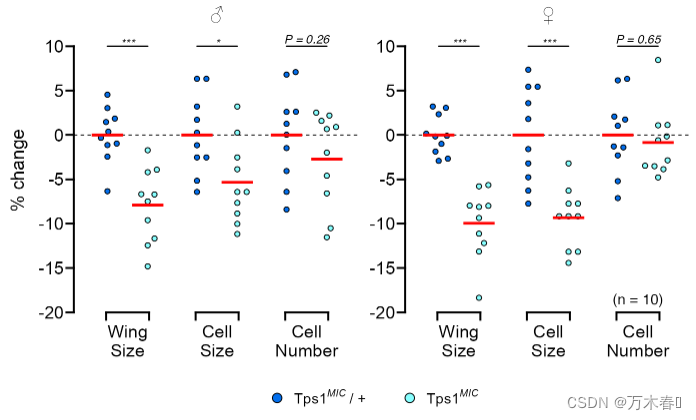
最后调整一下图例大小
p <- p + guides(fill = guide_legend(override.aes = list(size = 3)))
p





















 5万+
5万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








