聚类分析
层次聚类:对于小样本来说很实用(如150个观测值或更少)
-
每一个观测值自成一类
-
这些类每次两两合并
-
直到所有的类被聚成一类为止
划分聚类:能处理更大的数据量,但是需要事先确定聚类的个数
-
首先指定类的个数K
-
然后观测值被随机分成K类
-
再重新形成聚合的类

层次聚类方法可以用hclust()函数来实现,格式是hclust(d,method=)
-
其中d是通过dist()函数产生的距离矩阵
-
并且方法包括 "single"、"complete"、"average"、"centroid"和"ward"

摘自《R语言实践》
代码如下
setwd("D:/数据汇总/初步分析/聚类分析")
library(gclus)
library(vegan)
library(cluster)
#获得二元差异矩阵的函数,对于火和数据类型的聚类分析用cluster包中的daisy()函数
grpdist <- function(X)
{
require(cluster)
gr <- as.data.frame(as.factor(X))
distgr <- daisy(gr, "gower")
distgr
}
mydata<-read.csv("表型聚类数据.csv")
data.norm<-decostand(mydata[,4:12],"normalize")
data.ch <- vegdist(data.norm, "euc")
dev.new(
title = "Compare clustering methods",
width = 12,
height = 8,
noRStudioGD = TRUE
)
par(mfrow = c(2, 2))
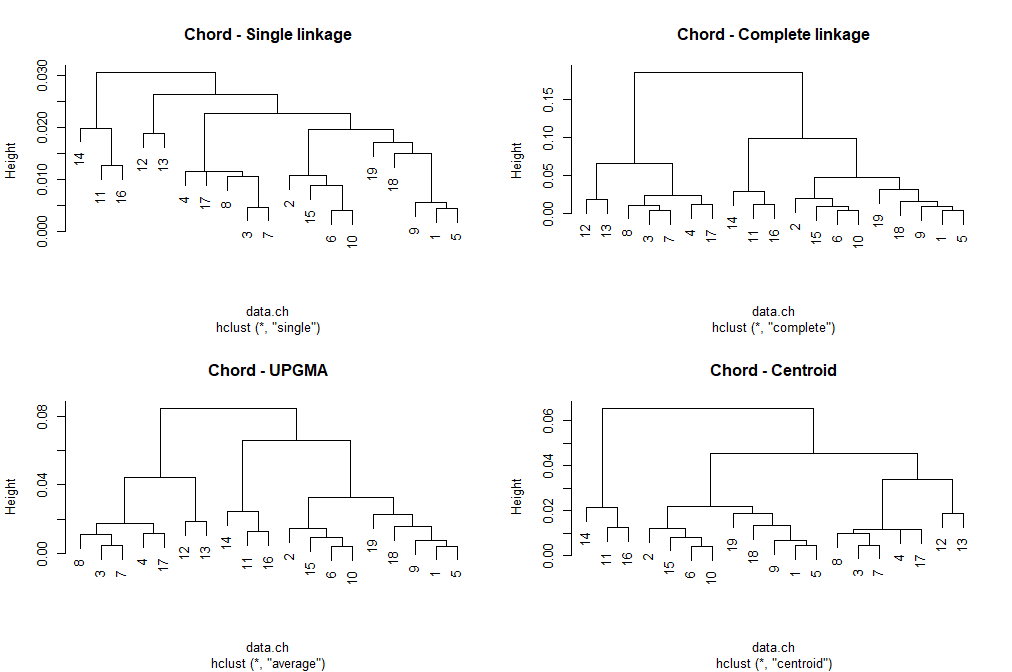
data.ch.single <- hclust(data.ch, method = "single")
plot(data.ch.single,
labels = rownames(mydata),
main = "Chord - Single linkage")
data.ch.complete <- hclust(data.ch, method = "complete")
plot(data.ch.complete,
labels = rownames(mydata),
main = "Chord - Complete linkage")
data.ch.UPGMA <- hclust(data.ch, method = "average")
plot(data.ch.UPGMA,
labels = rownames(mydata),
main = "Chord - UPGMA")
data.ch.centroid <- hclust(data.ch, method = "centroid")
plot(data.ch.centroid,
labels = rownames(mydata),
main = "Chord - Centroid")


























 1603
1603











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










