目录
1. 前言
热图比较有意思,最常用的科研可视化做图,观察显著、直接、简单粗暴。这是Toussaint Loua在1873年就曾使用过热图来绘制对巴黎各区的社会学统计。

2. 基本图形
以下均使用mtcars数据集作图:
rm(list = ls())
df <- scale(mtcars)
dat <- mtcars
2.1 经典简单热图
library(circlize)
mycols <- colorRamp2(breaks = c(-2, 0, 2),
colors = c("lightblue", "white", "red"))
Heatmap(df, name = "dat", col = mycols)
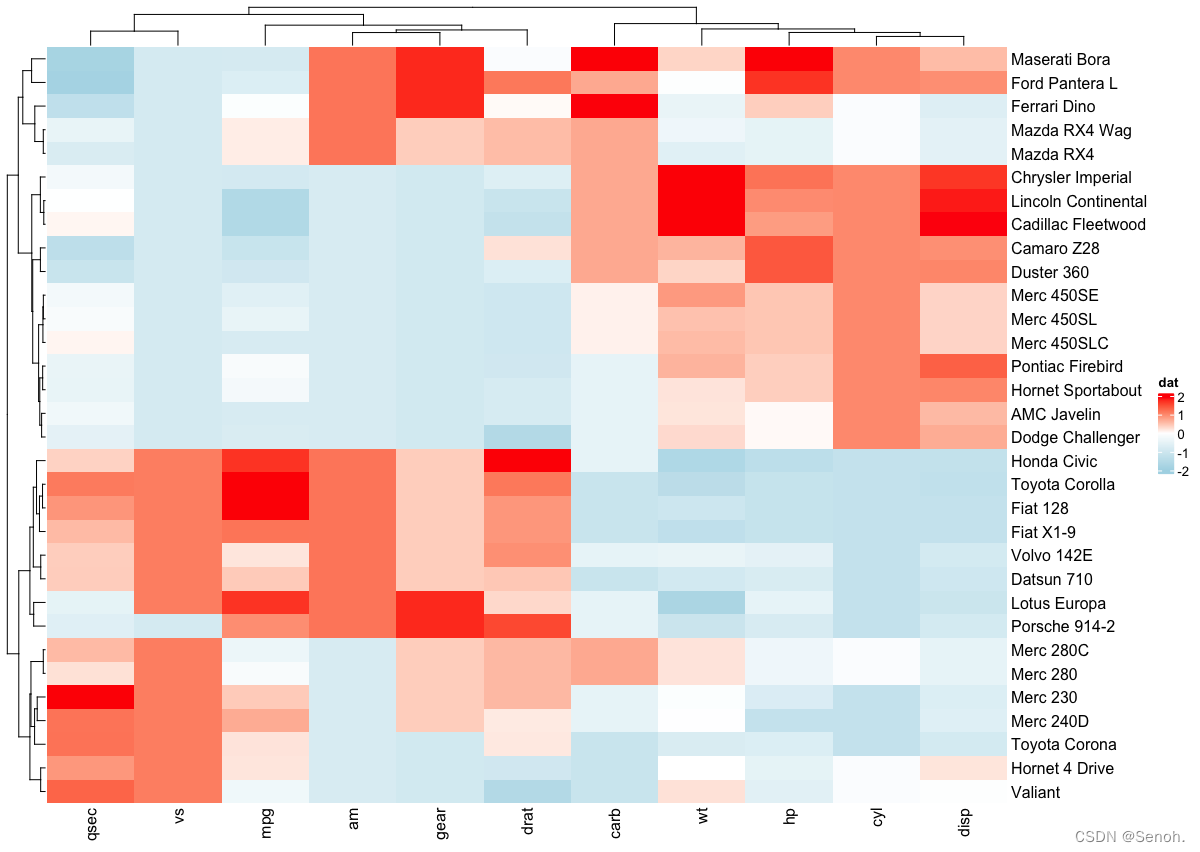
留意Heatmap和heatmap两个包不同!前者是ComplexHeatmap的,后者是base的
若无ComplexHeatmap包需要下载:
library(devtools)
install.packages('riskRegression')
#install_github("tagteam/riskRegression")
插播:M1芯片有可能会出现以下报错
ERROR: compilation failed for package ‘riskRegression’
* removing ‘/Library/Frameworks/R.framework/Versions/4.2-arm64/Resources/library/riskRegression’
* restoring previous ‘/Library/Frameworks/R.framework/Versions/4.2-arm64/Resources/library/riskRegression’
Error in for (i in seq_along(specs)) { :
Shadow graphics device error: r error 4 (lazy-load database '/Library/Frameworks/R.framework/Versions/4.2-arm64/Resources/library/gtable/R/gtable.rdb' is corrupt)
Error in for (i in seq_along(specs)) { :
Shadow graphics device error: r error 4 (lazy-load database '/Library/Frameworks/R.framework/Versions/4.2-arm64/Resources/library/gtable/R/gtable.rdb' is corrupt)
Warning messages:
1: In doTryCatch(return(expr), name, parentenv, handler) :
"name" is not a graphical parameter
2: In doTryCatch(return(expr), name, parentenv, handler) :
"split" is not a graphical parameter
3: In doTryCatch(return(expr), name, parentenv, handler) :
"name" is not a graphical parameter
4: In doTryCatch(return(expr), name, parentenv, handler) :
"split" is not a graphical parameter
5: In doTryCatch(return(expr), name, parentenv, handler) :
"name" is not a graphical parameter
6: In doTryCatch(return(expr), name, parentenv, handler) :
"split" is not a graphical parameter
7: In doTryCatch(return(expr), name, parentenv, handler) :
supplied color is neither numeric nor character
8: In i.p(...) :
installation of package ‘/var/folders/_6/nkcw7lgd5xb1kklnd4lb9q500000gn/T//Rtmpyx8rR7/file4c62493edad0/riskRegression_2023.03.18.tar.gz’ had non-zero exit status
这个因为依赖包riskRegression的最新版本2023.03.22目前好像仅intel芯片没问题,m芯片的下载2022.11.28版本即可,library检查一下
install.packages('riskRegression')
There is a binary version available but the source version is later:
binary source needs_compilation
riskRegression 2022.11.28 2023.03.22 TRUE
Do you want to install from sources the package which needs compilation? (Yes/no/cancel)
这里记得选no
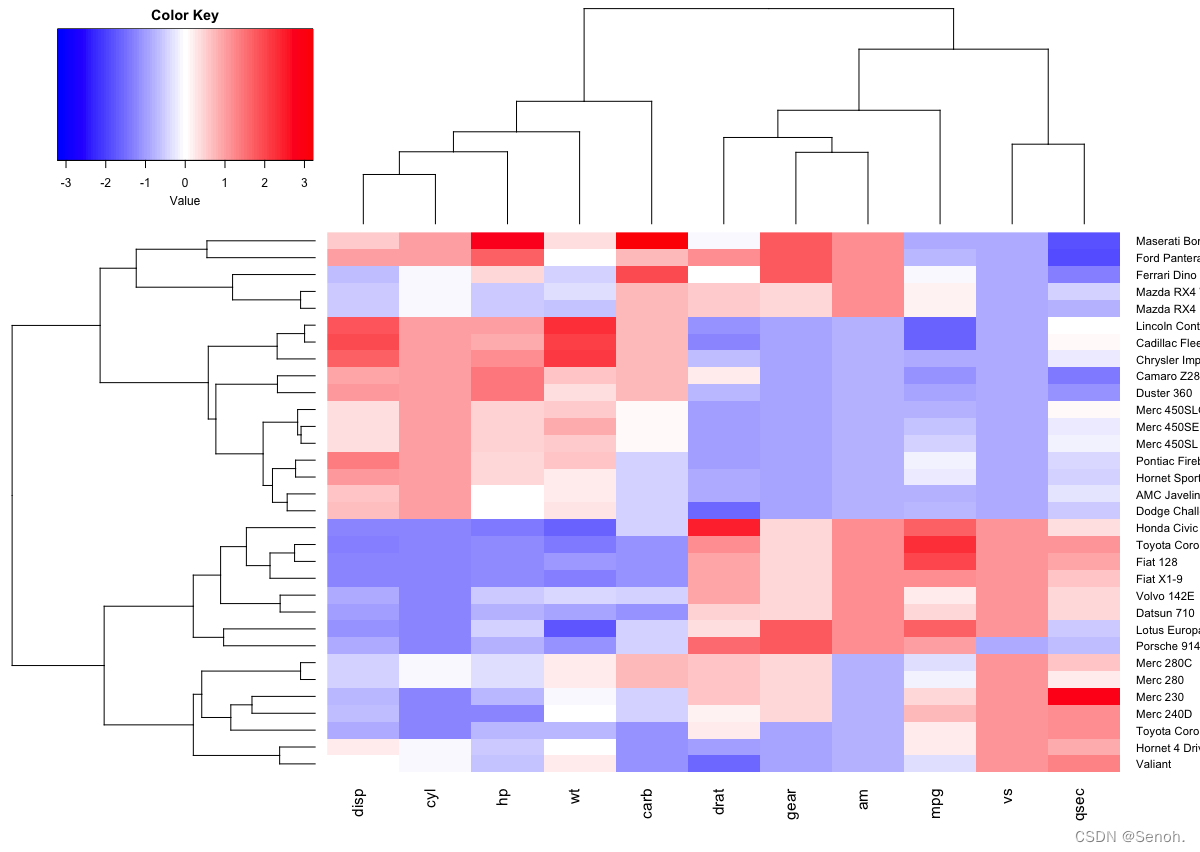
2.2 用heatmap2.0画热图
library("gplots")
heatmap.2(df, scale = "none", col = bluered(100),
trace = "none", density.info = "none")
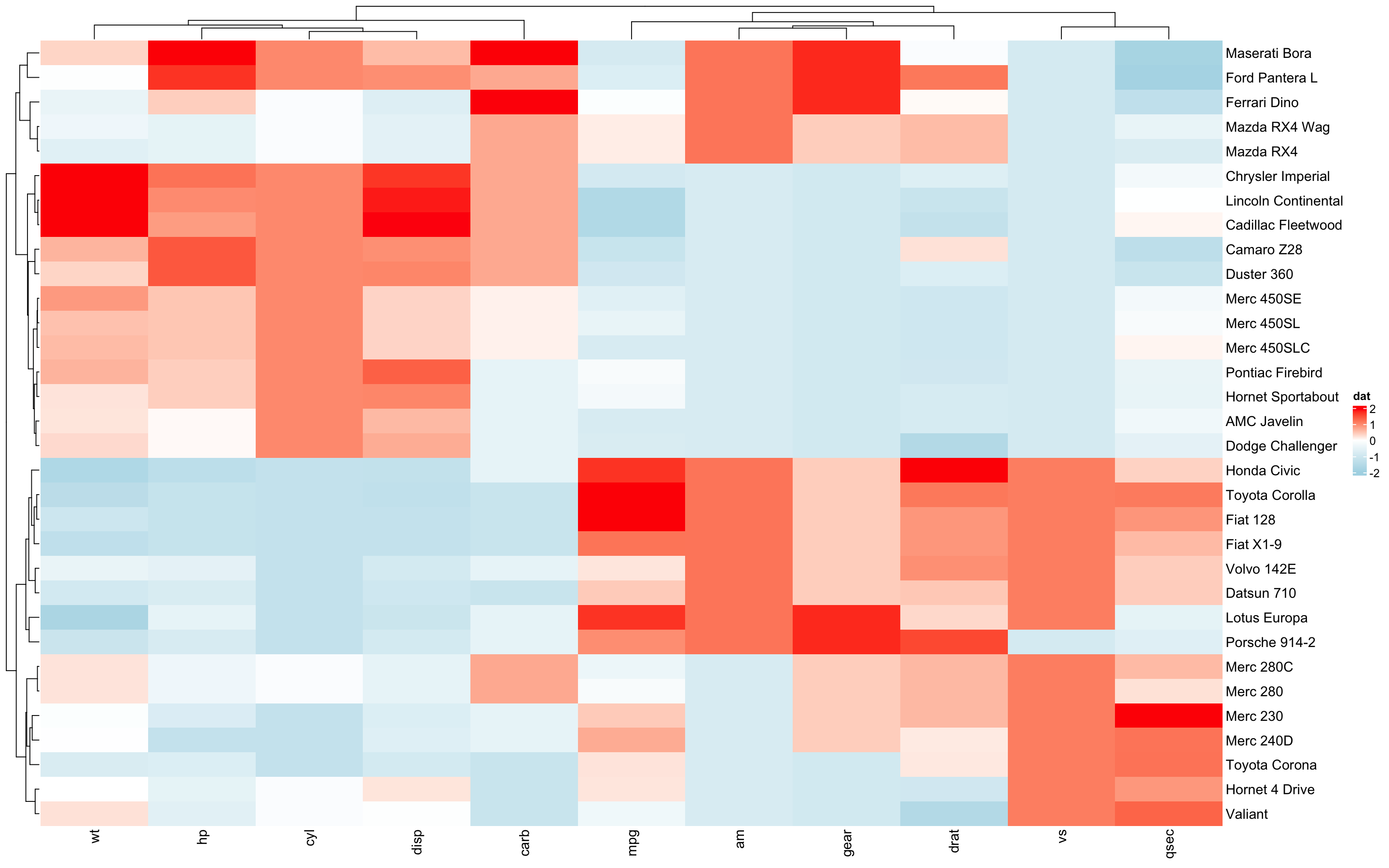
2.3 拆分热图
Heatmap(df, name = "dat", split = dat$cyl,
row_names_gp = gpar(fontsize = 7))
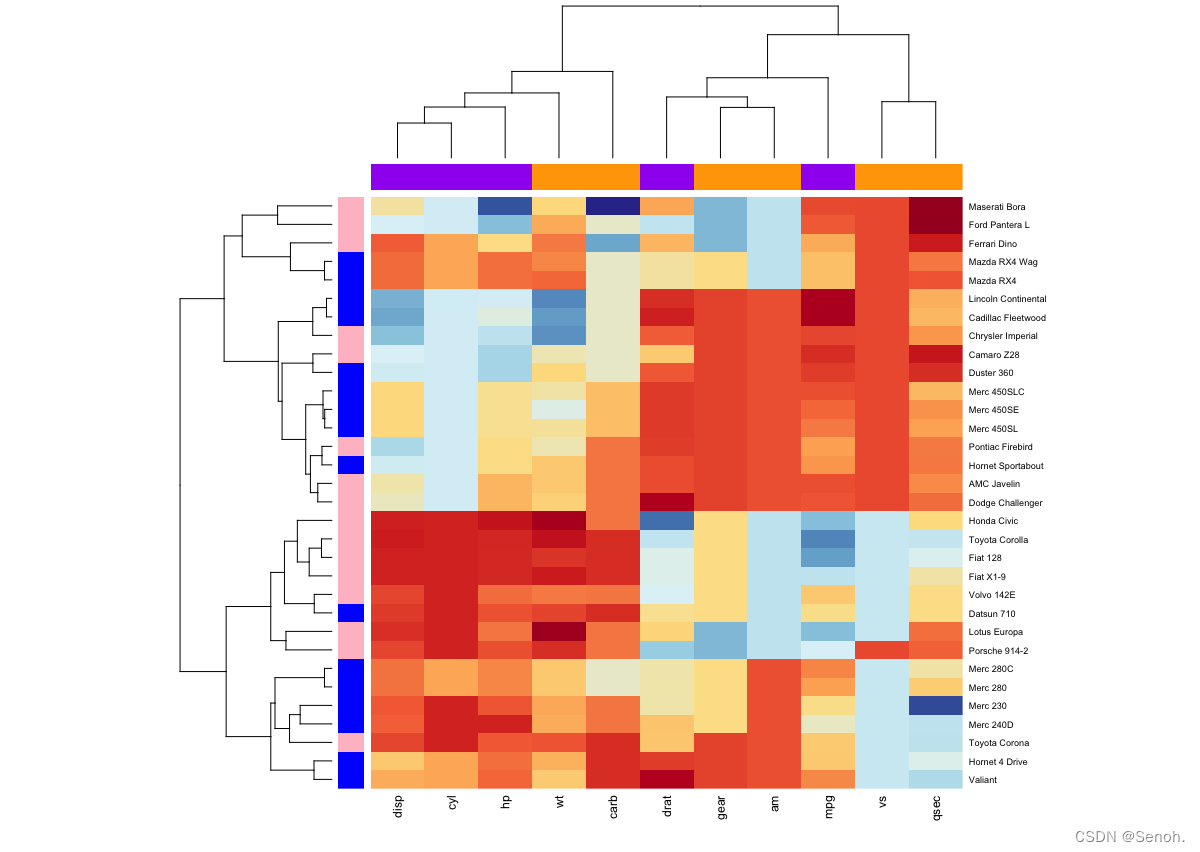
2.4 分类热图
library("RColorBrewer")
col <- colorRampPalette(brewer.pal(10, "RdYlBu"))(256)
heatmap(df, scale = "none", col = col,
RowSideColors = rep(c("blue", "pink"), each = 16),
ColSideColors = c(rep("purple", 5), rep("orange", 6)))
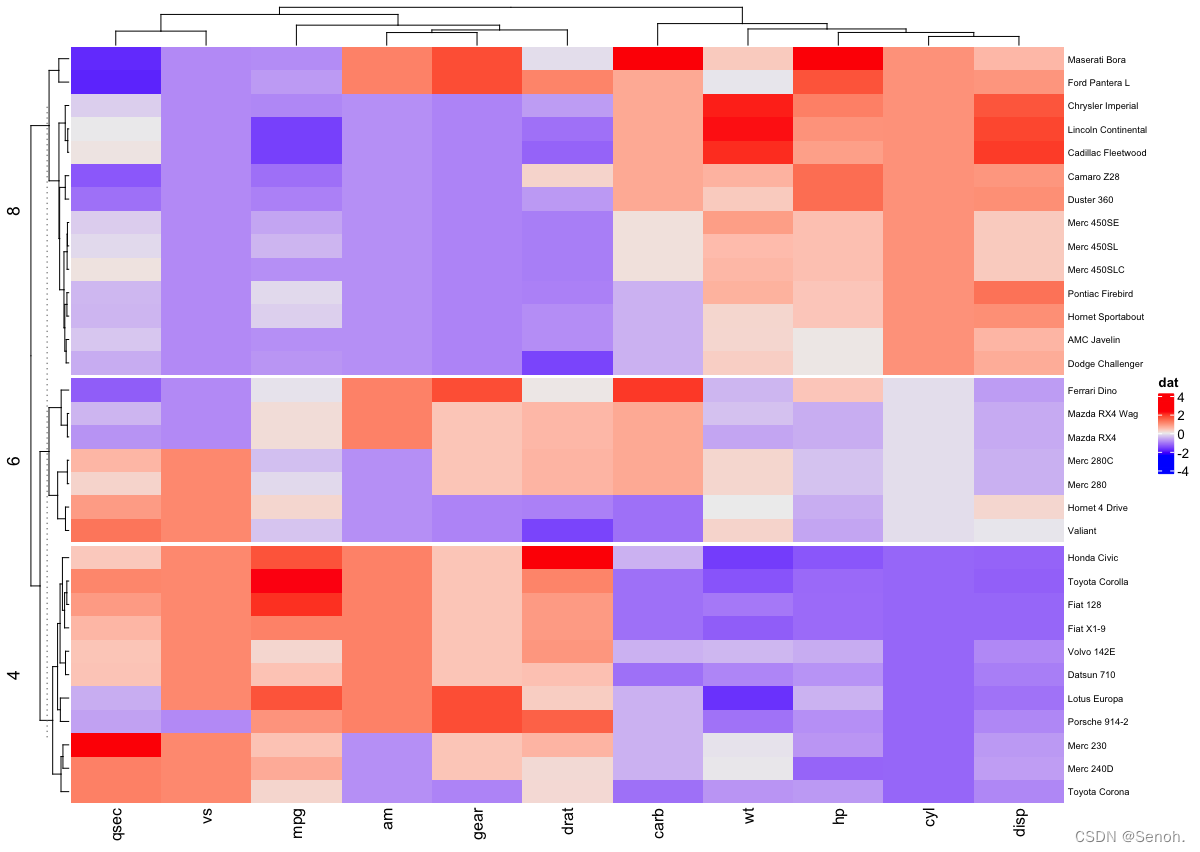
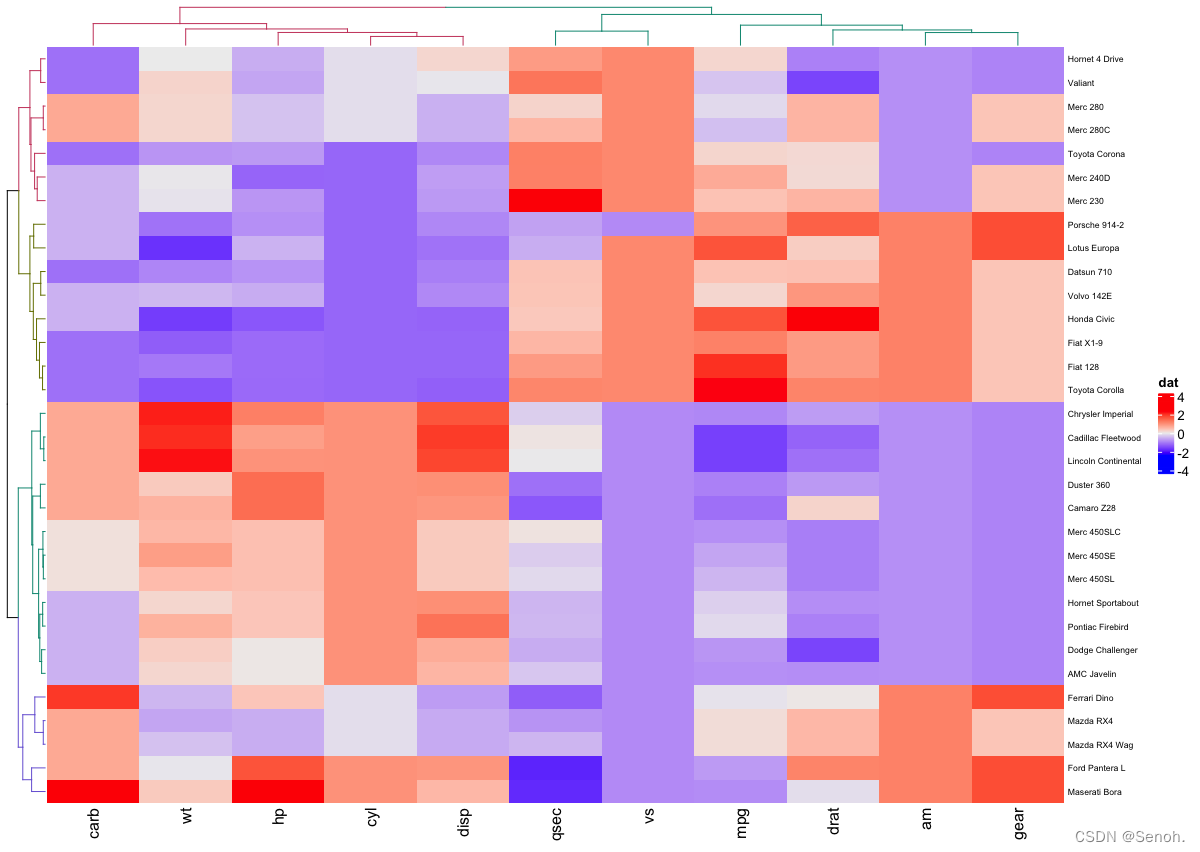
2.5 聚类树上色
#install.packages("dendextend")
library(dendextend)
row_dend = hclust(dist(df)) # 行聚类
col_dend = hclust(dist(t(df))) # 列聚类
Heatmap(df, name = "dat",
row_names_gp = gpar(fontsize = 6.5),
cluster_rows = color_branches(row_dend, k = 4),
cluster_columns = color_branches(col_dend, k = 2))
3. 进阶画图
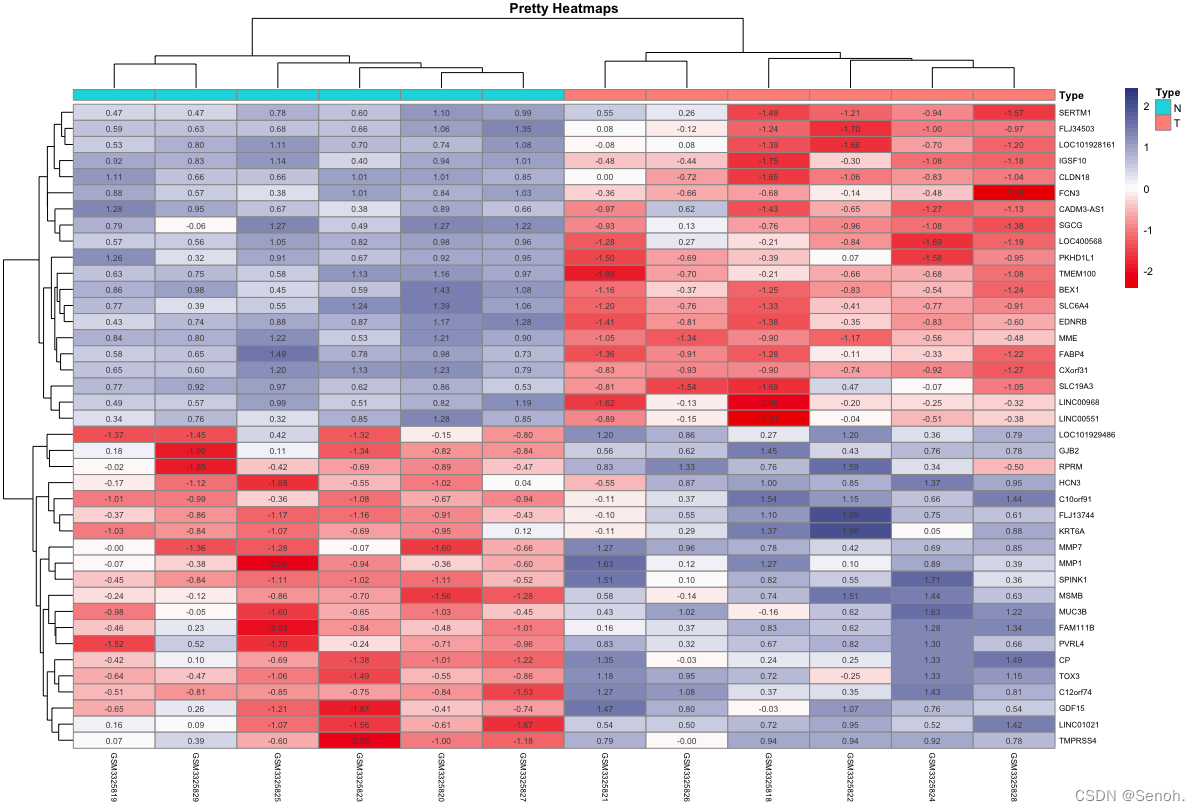
3.1 简单分组热图
这是热图最常见的形式,常用于样本正常组和疾病组之间的谱系表达差异
library(ComplexHeatmap)
library(pheatmap)
norcol="#EE0000FF" #疾病组
concol="#3B4992FF" #正常组
#读取文件
rt=read.table(inputFile,header=T,sep="\t",check.names=F,row.names=1)
group=read.table(groupFile,header=T,sep="\t",row.names=1,check.names=F)
rt <- as.matrix(rt)
pdf(file=outFile,width=8,height=7)
pheatmap(rt,
annotation=group,
main = "The Pretty Heatmaps by myself",#标题
cluster_cols = T,
color = colorRampPalette(c(norcol, "white", concol))(50),
show_colnames = T,
scale="row", #矫正
#border_color ="NA",
display_numbers = TRUE, #单元格数值
#cellwidth = 15, cellheight = 12,#设置单元格大小
fontsize = 8,
fontsize_row=6,
fontsize_col=6)
dev.off()
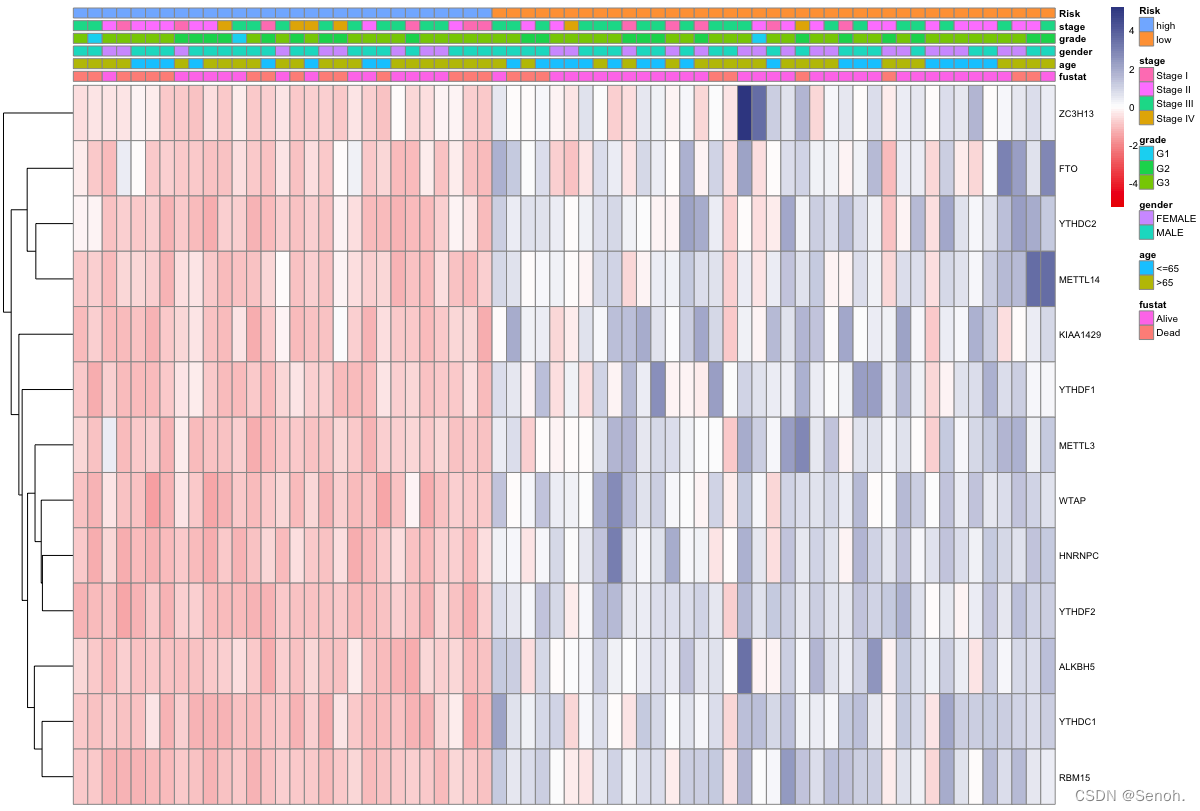
3.2 多性状分组热图
相比于常规转录组分析,多形状更倾向于描述性统计可视化谱系,可以附上各个需要展示的临床形状,结果简单直观
library(pheatmap)
var="Risk" #按照风险排序
norcol="#EE0000FF" #疾病组
concol="#3B4992FF" #正常组
#读取文件
rt=read.table(inputFile, sep="\t", header=T, row.names=1, check.names=F)
Type=read.table(groupFile, sep="\t", header=T, row.names=1, check.names=F)
#样品取交集
sameSample=intersect(colnames(rt),row.names(Type))
rt=rt[,sameSample]
Type=Type[sameSample,]
Type=Type[order(Type[,var]),] #按临床性状排序
rt=rt[,row.names(Type)]
#绘制热图
pdf(outFile,height=5,width=8)
pheatmap(rt, annotation=Type,
color = colorRampPalette(c(norcol, "white", concol))(90),
cluster_cols =F, #是否聚类
scale="row", #基因矫正
show_colnames=F,
fontsize=7.5,
fontsize_row=7,
fontsize_col=5)
dev.off()
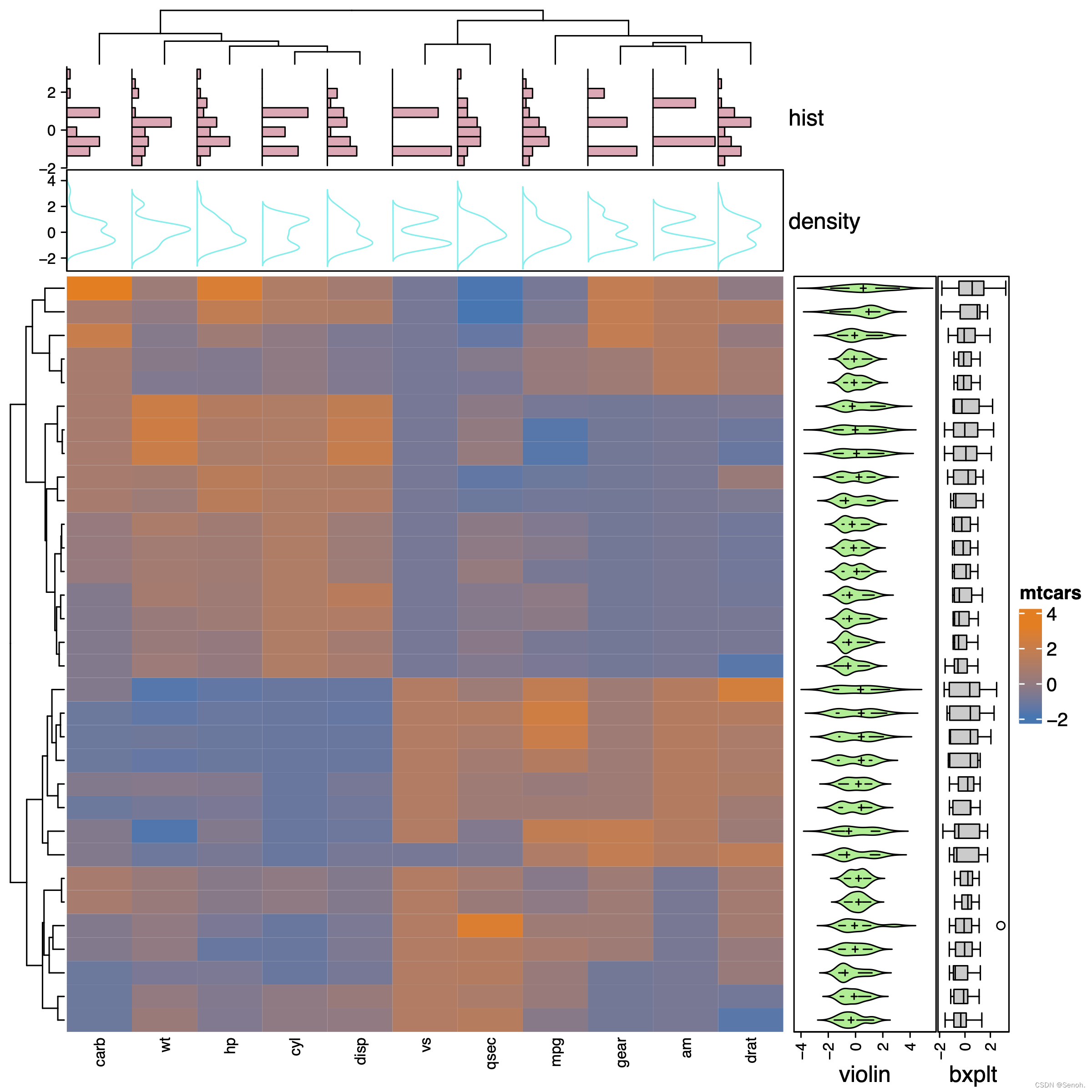
3.3 组合型热图
和上图目的类似,不过这里是依据每个样本或者每个基因进行小提琴图、条形图等可视化
找不到合适的数据,这里用的还是mtcars的数据
library(ComplexHeatmap)
#读取文件
dat=read.table(inputFile,header=T,sep="\t",check.names=F,row.names=1)
dat <- as.matrix(dat)
# 列的标注做图
.hist = anno_histogram(dat, gp = gpar(fill = "pink2"))
.density = anno_density(dat, type = "line", gp = gpar(col = "cyan2"))
ha_mix_top = HeatmapAnnotation(
hist = .hist, density = .density,
height = unit(3.8, "cm")
)
# 行的标注做图
.violin = anno_density(dat, type = "violin",
gp = gpar(fill = "lightgreen"), which = "row")
.boxplot = anno_boxplot(dat, which = "row")
ha_mix_right = HeatmapAnnotation(violin = .violin, bxplt = .boxplot,
which = "row", width = unit(4, "cm"))
# 将标注结合到热图上
pdf(outFile,height=8,width=8)
Heatmap(dat, name = "mtcars", col = c(ggsci::pal_d3()(2)),
column_names_gp = gpar(fontsize = 8),
top_annotation = ha_mix_top) + ha_mix_right
dev.off()
4. 讨论
热图的最大特点就是通过两种或以上的颜色直观反映部分数据的差异分布,以上大部分数据采用初始包的mtcars数据集,需要示例数据和完整代码的关注公众号「生信初学者」回复【pheatmap】领取。






















 257
257











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










