一、主成分分析的主要步骤
主成分分析(PCA)是一种无监督的线性变换技术,广泛地应用于不同的领域,特别是特征提取和降维。他的应用领域包括股票市场交易的探索性数据分析和去噪,以及生物信息学的基因组数据和基因表达水平分析。
PCA帮助我们根据特征之间的相关性来识别数据中的模型。PCA旨在寻找高维数据中存在的最大方差的方向,并将数据投影到维度小于或等于原始数据的新子空间。假设新特征轴彼此正交,该空间的正交轴(主成分)可以解释为方差最大的方向,如下图所示。其中x1、x2为原始特征轴,而PC1和PC2为主成分方向。
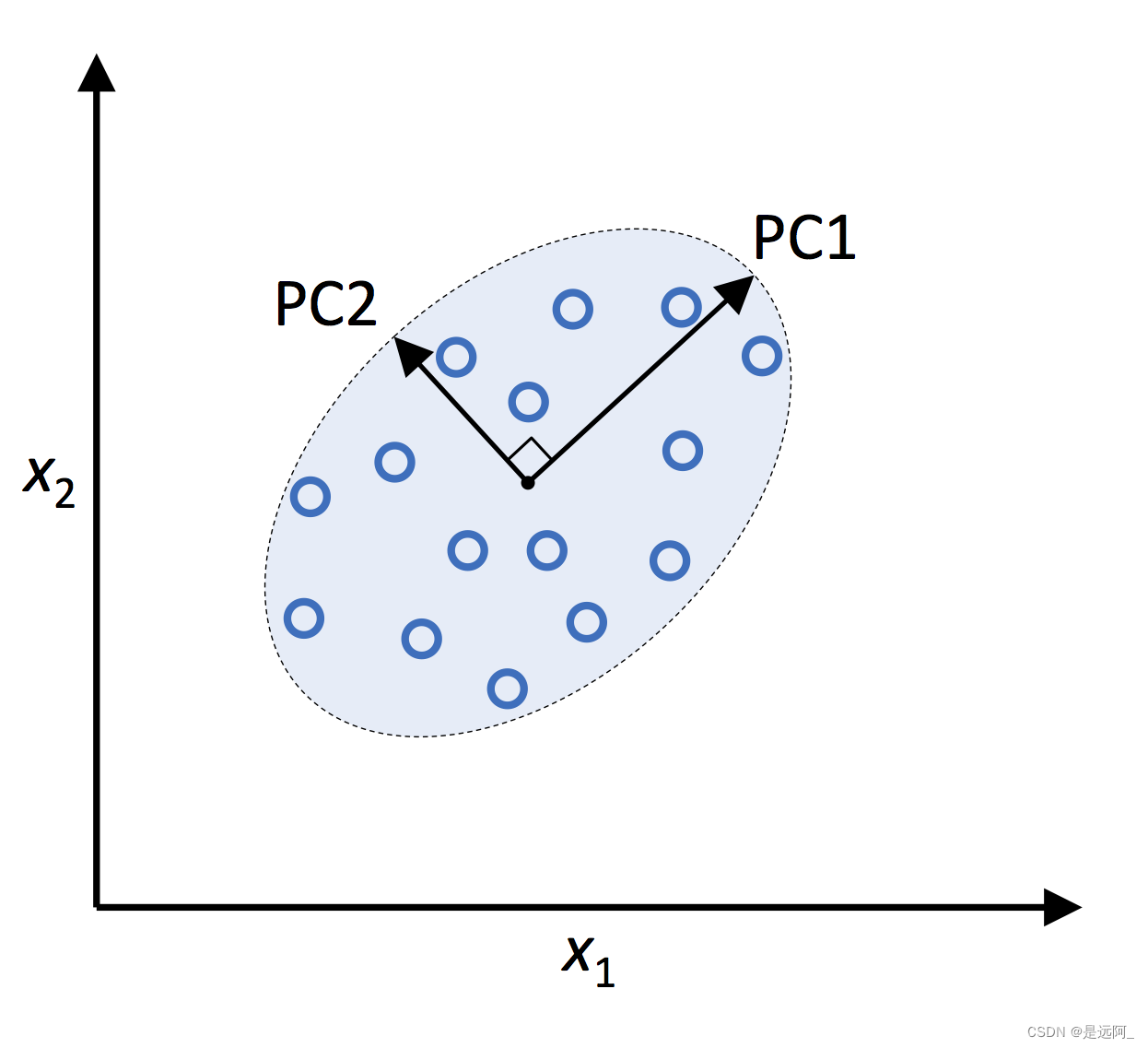
如果用PCA降维,我们可以构建dk维的变换矩阵W,它能把训练样本的特征向量x映射到新的k维特征子空间,该空间的维数比原来的d维特征空间要少。
例如,假设我们有一个特征向量x:
接着通过一个变换矩阵 进行变换:
结果以向量方式表达如下:
在原高维数据转换到k维新子空间(通常k≤d)的结果中,第一主成分的方差最大。另外,PCA 的方向对数据尺度非常敏感,需要在进行PCA之前对特征进行标准化。
假设我们只取一个主成分,则,该主成分为
,
因为为正定阵,所以有分解
,其中
为单位对称矩阵,
中的特征值降序排列。于是又有:
取为
的最大特征根
对应的特征向量
,
,则有:
同理,若取两个主成分分析,有,第二个主成分
此时,是最大的,且
基于此可构造:
PCA步骤简单概括如下:
1) 标准化d维数据集。
2) 构建协方差矩阵。
3) 将协方差矩阵分解为特征向量和特征值。
4) 以降序对特征值排序,从而对相应的特征向量排序。
5) 选择对应k个最大特征值的k个特征向量,其中k为新特征子空间的维数(k≤d)。
6) 由前k个特征向量构造投影矩阵W。
7) 用投影矩阵W变换d维输入数据集X以获得新的k维特征子空间。
二、提取主成分
以下讨论 PCA 的前四个步骤:
1) 标准化数据集。
2) 构建协方差矩阵。
3) 获取协方差矩阵的特征值和特征向量。
4) 以降序対特征值排序,从而对特征向量排序。
首先,加载葡萄酒数据集。
import pandas as pd
df_wine = pd.read_csv('https://archive.ics.uci.edu/ml/'
'machine-learning-databases/wine/wine.data',
header=None)
df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue',
'OD280/OD315 of diluted wines', 'Proline']
df_wine.head()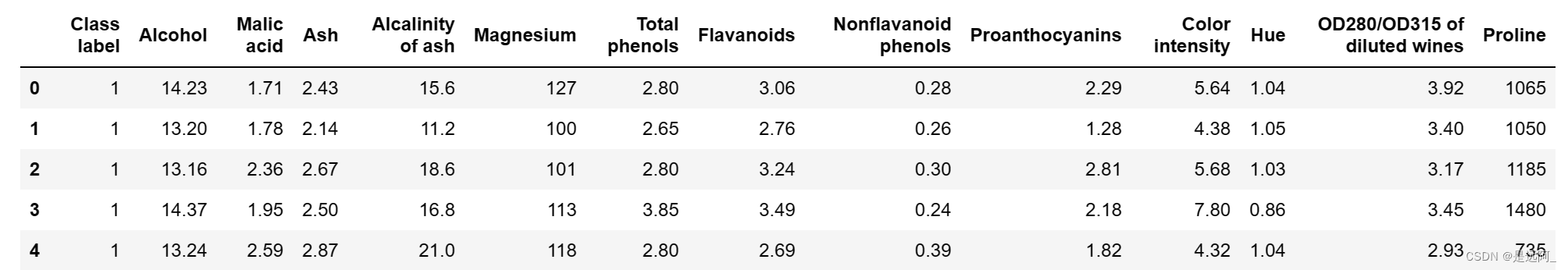
按照7:3的比例划分训练集、测试集,标准化为单位方差。
from sklearn.model_selection import train_test_split
X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
X_train, X_test, y_train, y_test = \
train_test_split(X, y, test_size=0.3,
stratify=y,
random_state=0)from sklearn.preprocessing import StandardScaler
sc = StandardScaler() #标准化
X_train_std = sc.fit_transform(X_train)
X_test_std = sc.transform(X_test)然后,构造协方差矩阵。
其中,特征和
之间的整体协方差计算公式如下:
特征向量v满足以下条件:
是特征值。
调用Numpy的linalg.eig函数来获得葡萄酒数据集协方差矩阵的特征向量和特征值。
import numpy as np
cov_mat = np.cov(X_train_std.T) #计算标准化训练集的协方差矩阵
eigen_vals, eigen_vecs = np.linalg.eig(cov_mat) #用np.linalg.eig完成特征分解
print('\nEigenvalues \n%s' % eigen_vals) #eigen_vals特征值
产生13个特征向量eigen_vals,及对应的特征向量储存在1313维矩阵eigen_vecs的列中。
三、总方差和解释方差
因为我们要降低维度,所以要找出前k个最重要的特征向量。在这之前,先把特征值的方差解释比计算出来。特征值的方差解释比就是
与特征值总和之比:
方差解释比 =
调用Numpy的cumsum函数,可以计算出解释方差和,然后使用Matplotlib的step函数绘图。
tot = sum(eigen_vals) #计算特征值的总和
var_exp = [(i / tot) for i in sorted(eigen_vals, reverse=True)] #计算每个特征值所占的方差解释比例
cum_var_exp = np.cumsum(var_exp) #对方差解释比例进行累积求和import matplotlib.pyplot as plt
plt.bar(range(1, 14), var_exp, alpha=0.5, align='center',
label='Individual explained variance')
plt.step(range(1, 14), cum_var_exp, where='mid',
label='Cumulative explained variance')
plt.ylabel('Explained variance ratio')
plt.xlabel('Principal component index')
plt.legend(loc='best')
plt.tight_layout()
plt.show()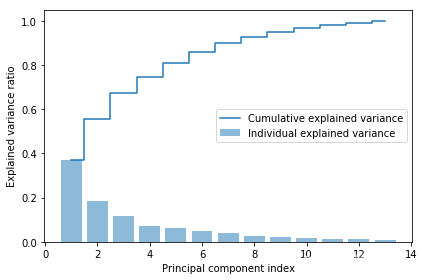
结果表明,第一主成分本身占方差的40%左右,前两个主成分合起来可解释数据集中几乎60%的方差。
四、特征变换
在把协方差矩阵分解为特征向量之后,接着完成最后的三个步骤,将葡萄酒数据集变换到新的主成分轴。
其余的步骤如下:
5)选择与前k个最大特征值对应的k个特征向量,其中k为新特征子空间的维数(k≤d)。
6)用前k个特征向量构建投影矩阵W。
7)用投影矩阵W变换d维输入数据集X以获得新的k维特征子空间。
把特征向量按特征值降序排列,从所选的特征向量构建投影矩阵,用投影矩阵把数据变换到低维子空间。
把特征向量按特征值降序排列:
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i])
for i in range(len(eigen_vals))] #将每个特征值的绝对值与对应的特征向量组成一个元组。
eigen_pairs.sort(key=lambda k: k[0], reverse=True)
#通过lambda k: k[0]指定按照元组中的第一个元素进行排序,reverse=True表示降序排列。收集对应前两个最大特征值的特征向量,从数据集中捕获大约60%的方差。这里只选择两个特征向量来进行说明。
w = np.hstack((eigen_pairs[0][1][:, np.newaxis],
eigen_pairs[1][1][:, np.newaxis]))
#eigen_pairs[0]表示第一个特征值-特征向量对,eigen_pairs[0][1]表示该特征值对应的特征向量。通过[:, np.newaxis]将其转换为列向量。
#使用np.hstack()函数将这两个列向量水平拼接起来,形成投影矩阵W。
print('Matrix W:\n', w)
#每一列都是一个主成分
依据前两个特征向量创建一个132维的投影矩阵W。
用投影矩阵将x(表示13维的行向量)变换到PCA子空间(主成分1和2),从而获得x’,即由两个新特征组成的二维实例向量:
X_train_std[0].dot(w) #X_train_std的第一条样本数据,使用投影矩阵W进行线性变换。通过计算矩阵点积,将整个训练集变换成两个主成分:
X_train_pca = X_train_std.dot(w)
colors = ['r', 'b', 'g']
markers = ['s', 'x', 'o']
for l, c, m in zip(np.unique(y_train), colors, markers):
plt.scatter(X_train_pca[y_train == l, 0], #属于标签为l的样本在第一和第二个主成分上的投影。
X_train_pca[y_train == l, 1],
c=c, label=l, marker=m)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
可视化后,由散点图可以看出与第二主成分(y轴)相比,数据更多地沿着x轴(第一主成分)传播,与前面方差解释比的结论一致。
五、使用sklearn进行实现
计算13个解释方差比。
from sklearn.decomposition import PCA
pca = PCA()
X_train_pca = pca.fit_transform(X_train_std)
pca.explained_variance_ratio_ #每个方差比例
解释方差比可视化。
plt.bar(range(1, 14), pca.explained_variance_ratio_, alpha=0.5, align='center')
plt.step(range(1, 14), np.cumsum(pca.explained_variance_ratio_), where='mid')
plt.ylabel('Explained variance ratio')
plt.xlabel('Principal components')
plt.show()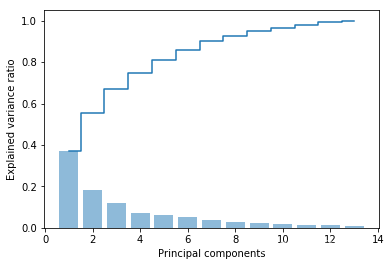
定义降维后维度并标准化。
pca = PCA(n_components=2) #降维后的特征数为2
X_train_pca = pca.fit_transform(X_train_std)
X_test_pca = pca.transform(X_test_std)plt.scatter(X_train_pca[:, 0], X_train_pca[:, 1])
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.show()PCA 是scikit-learn 的一个转换器类,我们首先用训练数据来拟合模型,然后用相同模型参数转换训练数据和测试数据。将PCA 类应用到葡萄酒训练数据集上,通过逻辑回归对转换后的样本进行分类,调用 plot_decision_regions函数实现决策区域的可视化。
from matplotlib.colors import ListedColormap
def plot_decision_regions(X, y, classifier, resolution=0.02):
markers = ('s', 'x', 'o', '^', 'v')
colors = ('red', 'blue', 'lightgreen', 'gray', 'cyan')
cmap = ListedColormap(colors[:len(np.unique(y))]) #colors[:len(np.unique(y))] 的意思是根据类别数量选择对应数量的颜色。
x1_min, x1_max = X[:, 0].min() - 1, X[:, 0].max() + 1 #X[:, 0] 表示提取特征向量 X 的所有行(样本),并只保留第一个维度的数值。
x2_min, x2_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution), #x1_min 到 x1_max 步长为 resolution 的一维数组
np.arange(x2_min, x2_max, resolution))
#np.meshgrid 函数生成一个二维的网格坐标矩阵。
Z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T) #将 xx1 和 xx2 的二维网格坐标矩阵展平为一维
#利用分类器的 predict 方法对展平后的特征向量进行预测,得到对应的类别标签。对于每个网格点,都得到了一个预测值。
Z = Z.reshape(xx1.shape)
plt.contourf(xx1, xx2, Z, alpha=0.4, cmap=cmap)
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
for idx, cl in enumerate(np.unique(y)): #表示对于每个唯一值 cl,使用变量 idx 来追踪它在数组中的索引
plt.scatter(x=X[y == cl, 0],
y=X[y == cl, 1],
alpha=0.6,
color=cmap(idx),
edgecolor='black',
marker=markers[idx],
label=cl)from sklearn.linear_model import LogisticRegression
pca = PCA(n_components=2)
X_train_pca = pca.fit_transform(X_train_std)
X_test_pca = pca.transform(X_test_std)
lr = LogisticRegression(multi_class='ovr', random_state=1, solver='lbfgs')
lr = lr.fit(X_train_pca, y_train)plot_decision_regions(X_train_pca, y_train, classifier=lr)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
训练数据的决策区域减少为两个主成分轴。
在测试数据集上同样绘制决策区域,可以得到样本的分类效果较好。
plot_decision_regions(X_test_pca, y_test, classifier=lr)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
将参数 n_components 设置为 None来初始化 PCA类,这样就可以保留所有的主成分,系统将返回排过序的所有主成分而不是进行降维。然后通过调用 explained_variance_ratio_属性访问解释方差比。
pca = PCA(n_components=None)
X_train_pca = pca.fit_transform(X_train_std)
pca.explained_variance_ratio_






















 3930
3930











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








