Ui和Server
ui就是shinyApp的外表,可以通过控件让用户自定义输入一些值,经server的R代码处理分析后的结果也会返回到ui
-
ui的种常见布局:fluidpage()
navbarPage() -
ui决定了页面布局,还是放
控件的地方,控件可以让用户自定义输入的值,比如滑块:
官方控件网址:https://shiny.rstudio.com/gallery/widget-gallery.html
扩展控件:首先要安装包install.packages("shinyWidgets")
扩展控件网址:http://shinyapps.dreamrs.fr/shinyWidgets/
server部分是shinyApp大脑,运行R代码的地方,包括数据的整理,计算以及可视化,最后的结果要通过output来传递给ui才能呈现到app的页面上
首先写一个简单的ShinyApp
ui部分:通过sliderInput()创建滑块,sliderInput()中,"data"是滑块定义的变量,会传入到server中;"exp"是server运行的结果,被传入到ui进行展示;在ui部分的值要用引号包起来 server部分:output$exp是R代码运行的结果;input$data是ui传入的变量;在server部分传入传出的值要跟$
renderPlot:在server部分来进行绘图,server中用render* plotOutput:接受server画出的图,并在页面进行展示, ui中用*Output
renderText 和 textOutput用来展示一小段文本 renderUI和uiOutput,会根据控件进行变化,具有更大的灵活性,构建复杂的UI元素,许多图,文字,标题
##ui部分
fluidPage(
# Copy the line below to make a slider range
sliderInput("data",
label = h3("次方"),
min = 2,
max = 10,
value = c(4)),
shiny::plotOutput( "exp")
)
##server部分
##server中function,共有三项参数:input, output, session
#input:从ui中传入的参数
#output:R的代码运行结束后的结果,会传入ui中进行展示
#session:最后不要省略,决定依赖关系,省略有时代码会报错
function(input, output, session) {
##renderPlot:用来渲染绘图代码,其他的渲染就找render*
output$exp <- shiny::renderPlot({
curve(x^input$data, from = -5, to = 5)
})
}
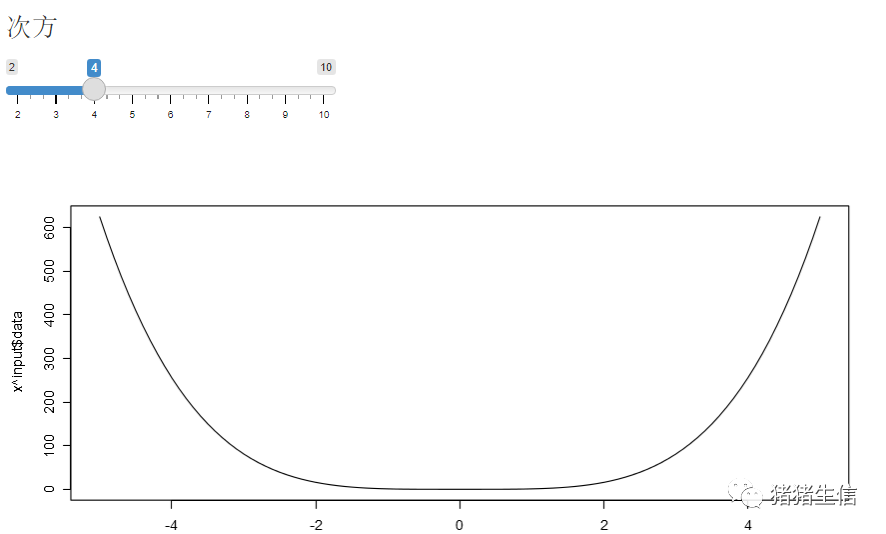
image-20210503162142045
具有相互依赖控件的ShinyApp

image-20210503170710978
ui <-navbarPage("相互依赖的控件",
tabPanel(
"第一页",
shinyWidgets::colorPickr(
inputId = "mycolor_1",
label = "选择一种颜色 :"),
plotOutput(
outputId = "hplot_1",
width = "100%",
height = "400px"
)
),
tabPanel(
"第二页",
shinyWidgets::colorPickr(
inputId = "mycolor_2",
label = "选择一种颜色 :"),
plotOutput(
outputId = "hplot_2",
width = "100%",
height = "400px"
)
)
)
function(input, output, session) {
library(ggplot2)
set.seed(1234)
wdata = data.frame(
sex = factor(rep(c("F", "M"), each=200)),
weight = c(rnorm(200, 55), rnorm(200, 58)))
##如果第一个图颜色改变,第二个图也更改为图一的颜色
shiny::observeEvent(input$mycolor_1,{
shinyWidgets::updateColorPickr(session,
inputId = "mycolor_2",
value = input$mycolor_1)
})
##如果第二个图颜色改变,第一个图也更改为图二的颜色
shiny::observeEvent(input$mycolor_2,{
shinyWidgets::updateColorPickr(session,
inputId = "mycolor_1",
value = input$mycolor_2)
})
server <- output$hplot_1 <- renderPlot({
ggplot(wdata, aes(x=weight)) +
geom_histogram(binwidth=1,colour = input$mycolor_1,fill = input$mycolor_1, alpha=0.5)
})
# Basic histogram
output$hplot_2 <- renderPlot({
ggplot(wdata, aes(x=weight)) +
geom_histogram(binwidth=1,colour = input$mycolor_2,fill = input$mycolor_2, alpha=0.5)
})
# Basic histogram
}
##运行ShinyApp
shinyApp(ui = ui, server = server)
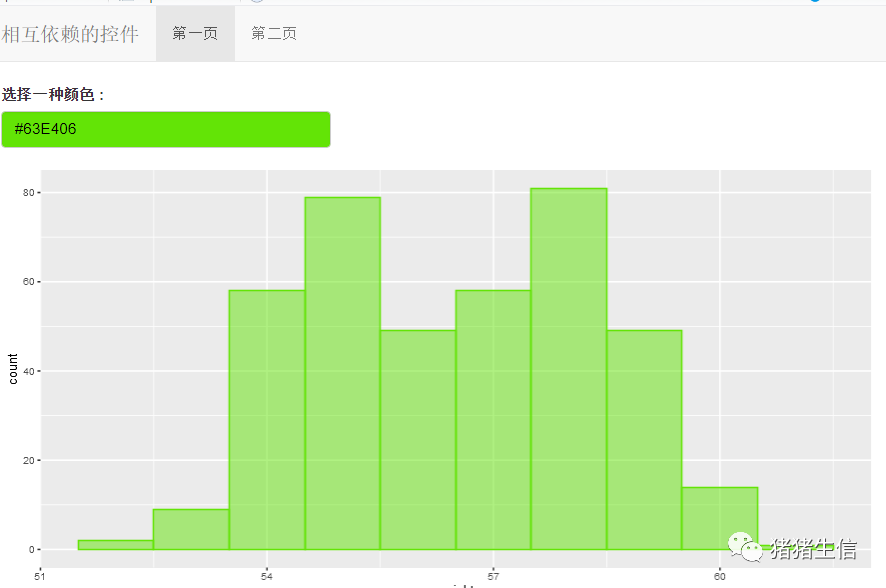
image-20210503170735837
制作热门基因词云shinyApp
思路:先敲出热门基因的词云图的代码,在嵌入到shiny代码中
R词云绘图代码:
下载NCBI的entrez ID号对应着基因发表过文章的pubmedID号:下载链接:ftp://ftp.ncbi.nlm.nih.gov//gene/DATA/gene2pubmed.gz
##直接打开网页下载或用bash命令下载都行
#发现有wget命令下载挺快的,下面是win10下wsl2的下载方式
alt+shift+T 打开win的终端
> bash
> wget -c ftp://ftp.ncbi.nlm.nih.gov//gene/DATA/gene2pubmed.gz #不到半分钟就好了
下载完的文件用notepad++打开,把第一行的#号删除保存在读取到R中
#The 5 main steps to create word clouds in R
#Step 1: Create a text file
#将上面下载好的文件读取到R中
gene2pub <- read.table("1.词云作业/gene2pubmed",header = T,sep = "\t")
head(gene2pub)
#Step 2 : Install and load the required packages
# Install
install.packages("tm") # for text mining
install.packages("SnowballC") # for text stemming
install.packages("wordcloud") # word-cloud generator
install.packages("RColorBrewer") # color palettes
# Load
library("tm")
library("SnowballC")
library("wordcloud")
library("RColorBrewer")
#Step 3 : Text mining
## 选取猪的基因 human:9606 pig:9823
## https://www.uniprot.org/taxonomy/9823
gene2pub <- gene2pub[gene2pub$tax_id==9823,]
head(gene2pub)
#Step 4 : Build a term-document matrix
tb <- as.data.frame(as.data.frame(table(gene2pub$GeneID)))
head(tb)
tb=tb[order(tb$Freq,decreasing = T),]
tb=head(tb,100)
colnames(tb)[1]='gene_id'
set.seed(1234)
head(tb)
#Step 5 : Generate the Word cloud
wordcloud(words = tb$gene_id, freq = tb$Freq, min.freq = 1,
max.words=200, random.order=FALSE, rot.per=0.35,
colors=brewer.pal(8, "Dark2"))
第一次结果:根据基因热门程度,以不同的大小显示出来:
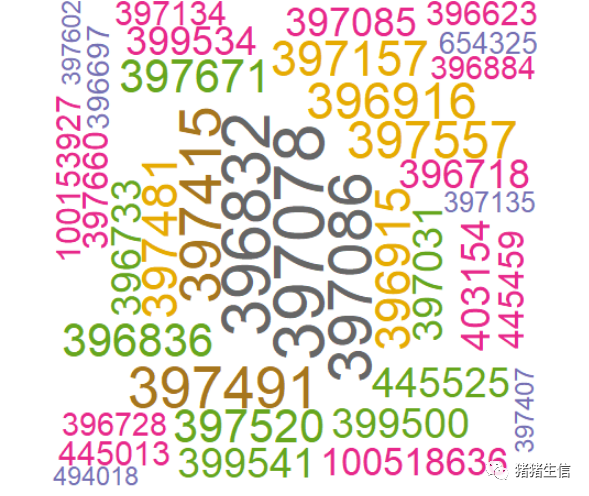
进行第二次绘图完善:因为上面的图还是entrez ID,所以要转换成gene名来进行展示
#Step 6 :
library(org.Ss.eg.db) ## 加载猪的数据库
ids=toTable(org.Ss.egSYMBOL)
head(ids)
tbs=merge(ids,tb,by='gene_id')
wordcloud(words = tbs$symbol, freq = tbs$Freq, min.freq = 1,
max.words=200, random.order=FALSE, rot.per=0.35,
colors=brewer.pal(8, "Dark2"))

image-20210503190809138
将绘图代码填入到shiny代码中:
gene2pub <- read.table("1.词云作业/gene2pubmed",header = T,sep = "\t")
library(shiny)
ui <- fluidPage(
# Copy the line below to make a slider range
sliderInput("num",
label = h3("选择热门基因的数量"),
min = 1,
max = 200,
value = c(100)),
# Copy the line below to make a set of radio buttons
radioButtons("radio", label = h3("please choose taxid"),
choices = list("human" = 9606, "pig" = 9823),
selected = 1),
shiny::plotOutput( "cloud")
)
server <- function(input, output, session) {
library(org.Hs.eg.db)
library(org.Ss.eg.db)
library("tm")
library("SnowballC")
library("wordcloud")
library("RColorBrewer")
#gene2pub <- read.table("gene2pubmed",header = T,sep = "\t")
#load("1.词云作业/gene2pub.rdata")
ids_hunman=toTable(org.Hs.egSYMBOL)
ids_pig=toTable(org.Ss.egSYMBOL)
#head(ids)
output$cloud <- shiny::renderPlot({
if (input$radio==9606){
a<- gene2pub[gene2pub$tax_id==9606,]
tb <- as.data.frame(as.data.frame(table(a$GeneID)))
# head(tb)
tb=tb[order(tb$Freq,decreasing = T),]
tb=head(tb,200)
colnames(tb)[1]='gene_id'
set.seed(1234)
tbs=merge(ids_hunman,tb,by='gene_id')
}else{
a<- gene2pub[gene2pub$tax_id==9823,]
tb <- as.data.frame(as.data.frame(table(a$GeneID)))
# head(tb)
tb=tb[order(tb$Freq,decreasing = T),]
tb=head(tb,200)
colnames(tb)[1]='gene_id'
set.seed(1234)
tbs=merge(ids_pig,tb,by='gene_id')
}
wordcloud(words = tbs$symbol, freq = tbs$Freq, min.freq = 1,
max.words=input$num, random.order=FALSE, rot.per=0.35,
colors=brewer.pal(8, "Dark2"))
})
}
##运行
shinyApp(ui, server)
结果:
可以选择人和猪这两个物种的热门基因进行查看 选择human参数:
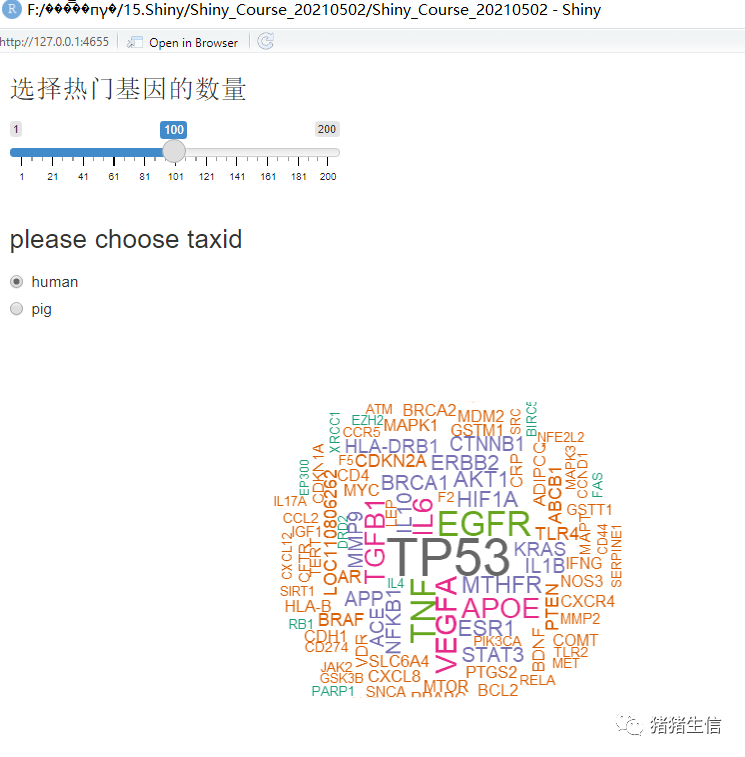
选择pig参数:

【参考资料】
https://mp.weixin.qq.com/s/VCRukFCQaagTF6GKu2DLgA






















 170
170











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










