铜绿假单胞菌是一种重要的人类细菌病原体,可导致多药耐药感染,尤其在免疫缺陷或患有炎症性肺部疾病(如囊性纤维化,CF)的人群中。然而,其如何进化为适应性强、全球传播的病原体尚不清楚。本研究结合群体基因组探索、转录组和表型研究,深入探究了铜绿假单胞菌的致病进化过程。

论文链接:
https://www.science.org/doi/10.1126/science.adi0908
本研究通过对铜绿假单胞菌的研究,揭示了其从环境微生物进化为主要人类病原体的关键步骤,包括水平基因转移产生流行克隆、克隆对宿主的不同偏好、多轮宿主特异性适应以及最终导致不同患者群体间传播受限,强调了全球监测和预防交叉感染的重要性。文章中基因组和转录组数据处理过程中用到的FastTree、Kallisto、DESeq2等工具都可以在Galaxy生信云平台(网站:usegalaxy.cn)上使用分析,无需写代码,无需安装软件配置环境。下面我们一起来学习这篇文章的研究思路和测序数据分析方法。

研究结果
系统发育分析揭示全球流行克隆
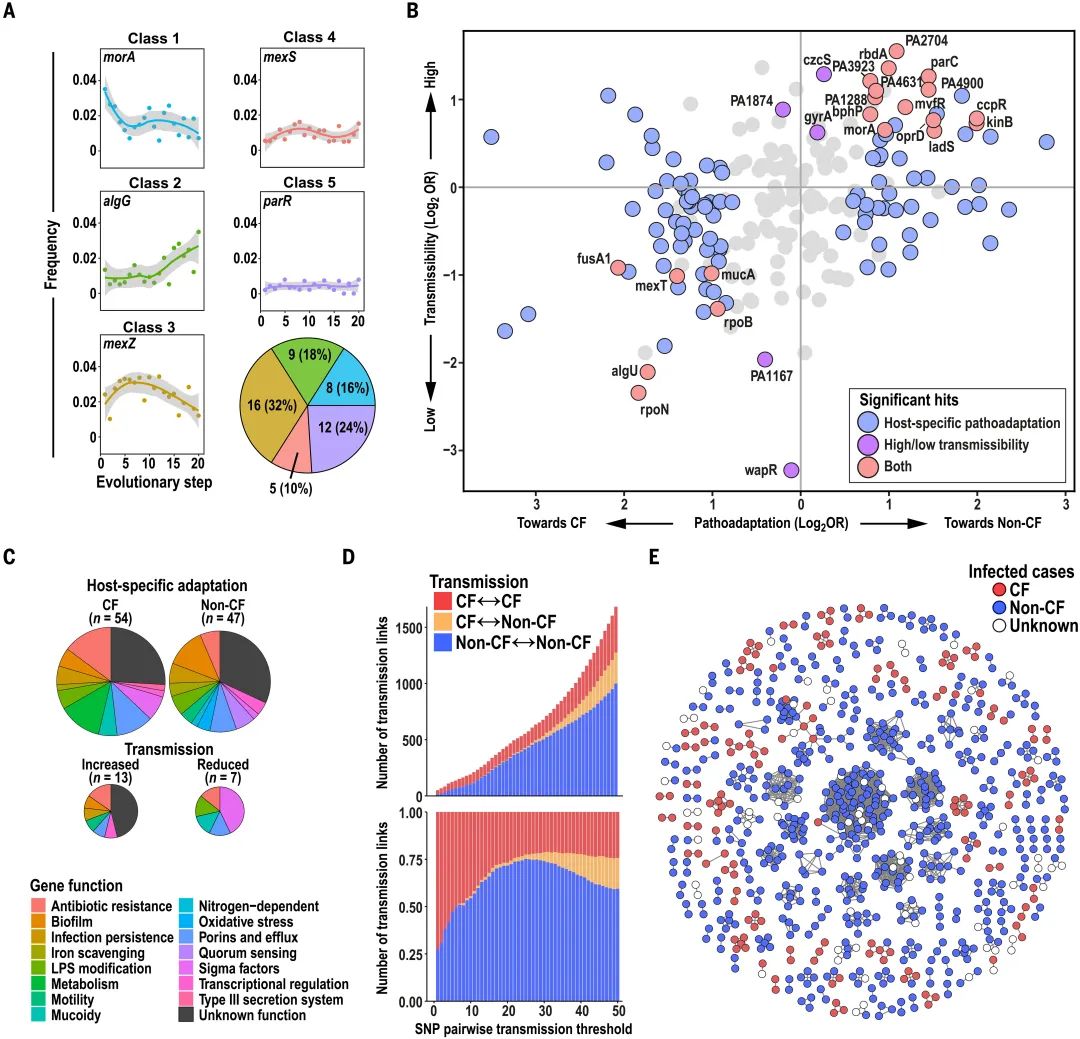
分析9829个来自全球的铜绿假单胞菌分离株,包括9573个来自2765名患者的临床样本,基于单核苷酸多态性(SNP)距离分组,确定了21个主要克隆,即“流行”克隆 。这些流行克隆在感染人类中更易被检测到,导致全球51%的临床感染,广泛分布在系统发育树中且全球传播。
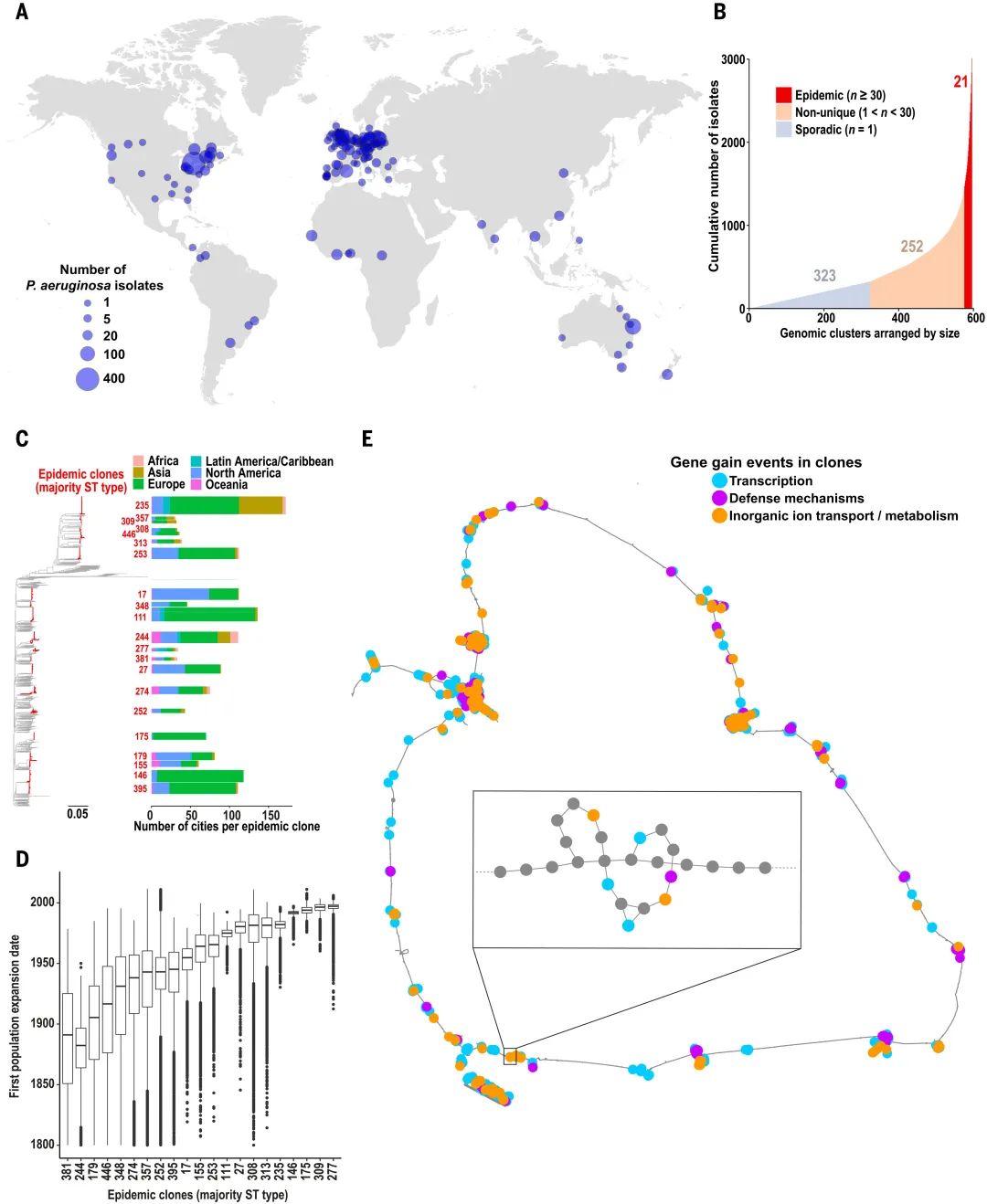
推断流行克隆的种群扩张和地理分布随时间变化
样本跨越100多年,通过贝叶斯时间重建估计流行克隆在17世纪末至20世纪末非同步出现,1850年至2000年间经历种群扩张 。对于地理聚集的克隆,用贝叶斯系统地理方法推断其祖先位置,发现流行克隆可能起源于世界各地。
水平基因转移可能驱动流行克隆的出现
通过泛基因组图分析,比较流行克隆和散发性分离株的附属基因组,发现流行克隆在转录调控、无机离子运输等相关基因显著富集,而细菌防御和分泌相关基因显著减少,表明细菌生理的根本变化可能驱动了流行克隆的发展。
流行克隆的内在宿主偏好不同
研究发现不同流行克隆对CF和非CF个体感染偏好不同 。泛基因组分析未发现宿主亲和力与特定功能基因获取有关,转录组分析则表明临床分离株基于流行克隆的宿主偏好在转录空间聚集,确定了624个与CF感染亲和力正相关和514个负相关的基因。
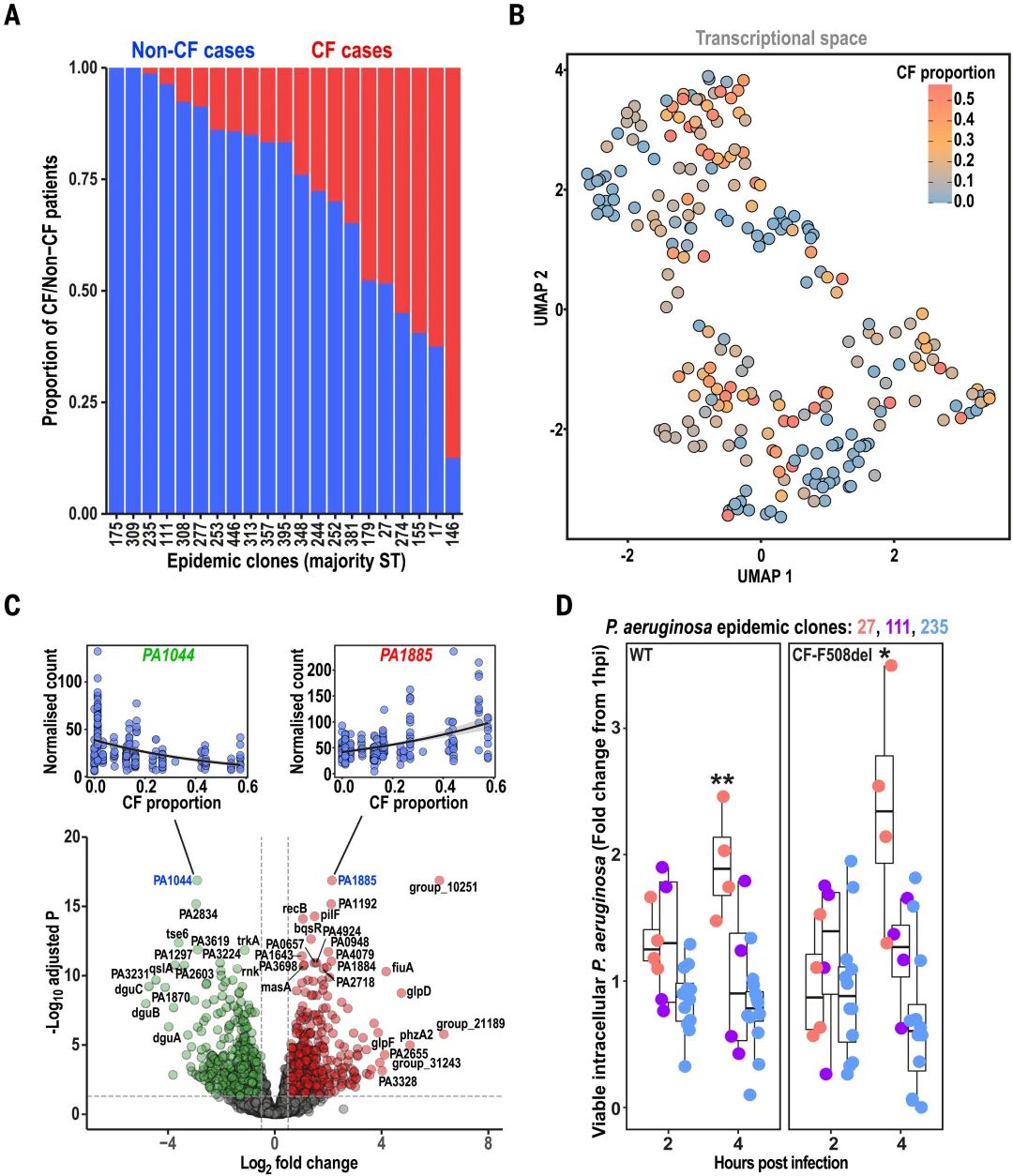
高CF亲和力克隆在巨噬细胞内的存活率增加
对49个代表性分离株多维表型特征分析发现,高CF亲和力克隆(如ST27)的分离株在野生型和CF巨噬细胞系中的细胞内存活和复制能力显著高于低、中CF亲和力克隆,表明增强的宿主先天免疫逃避可能解释其感染CF患者的成功。
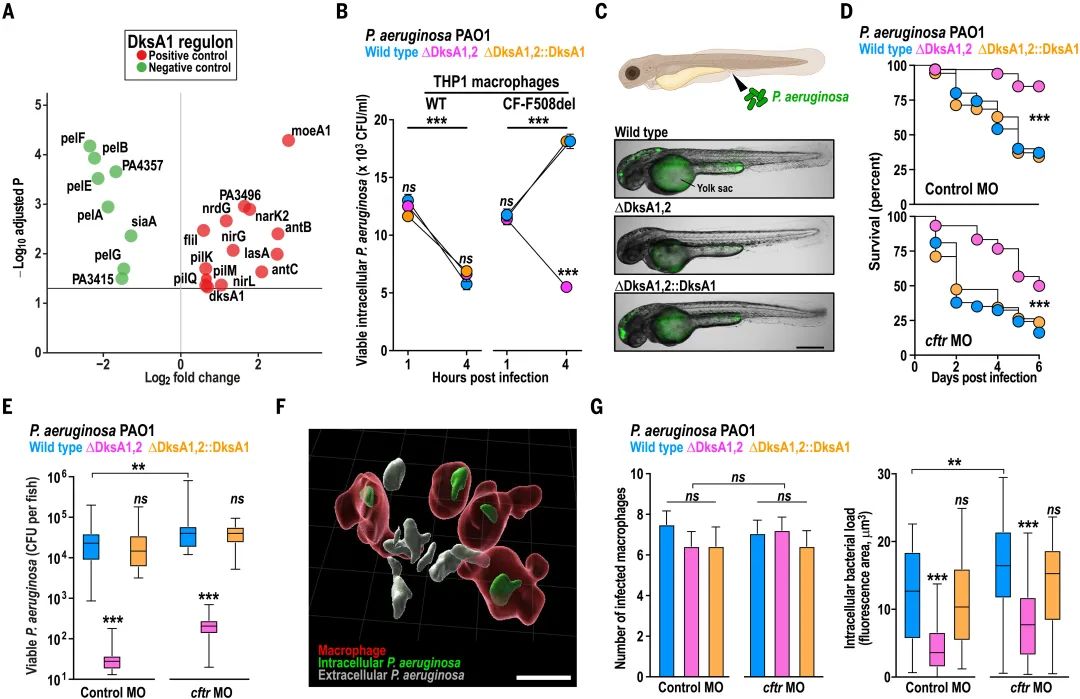
流行菌株的宿主偏好由DksA1表达介导
研究发现严格反应调节剂DksA1的表达及其调控子的激活与CF亲和力相关 。在巨噬细胞和斑马鱼感染模型中,缺失DksA1会影响细菌在CF巨噬细胞内的存活和在体内的感染能力,表明DksA1表达差异可能解释流行克隆在巨噬细胞内存活能力和宿主偏好的不同。
铜绿假单胞菌的趋同宿主特异性适应
分析单个克隆的近期突变历史,发现224个“致病适应性”基因,其突变多为非同义且有害,功能注释表明它们在致病过程中起重要作用 。CF分离株与其他分离株聚类不同且积累更多突变,表明CF肺是具有不同选择压力的独特生态位。
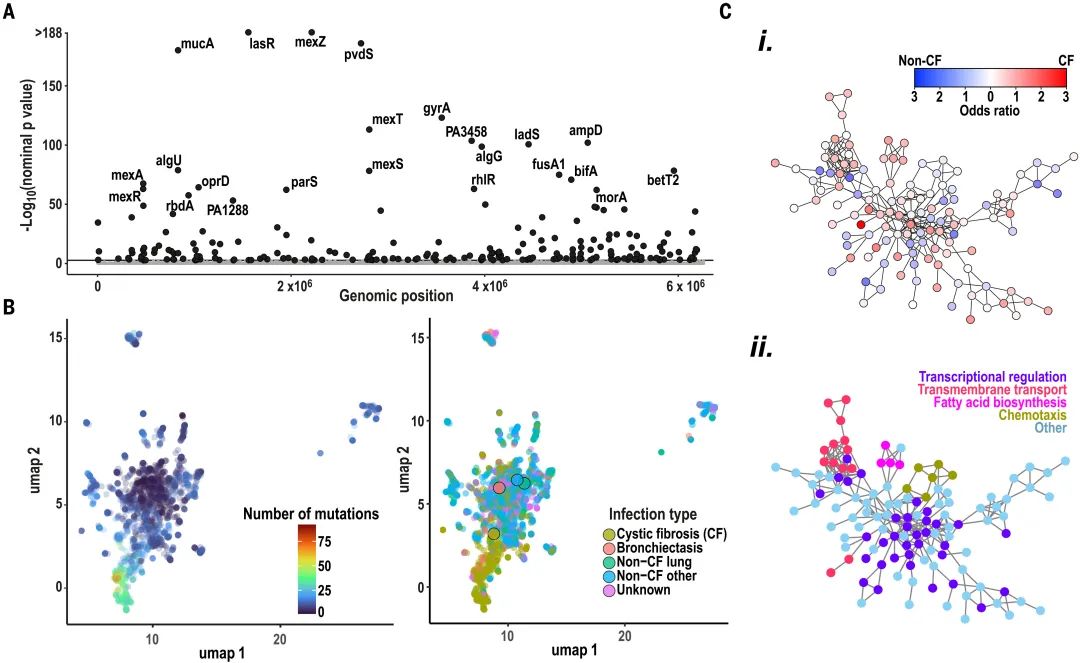
不同的进化轨迹导致宿主特化
通过重建每个致病适应性突变的获取顺序,发现CF分离株进化轨迹更长 。分析致病适应性基因对宿主特异性适应和细菌传播性的影响,发现一些基因与传播性或宿主特异性适应改变有关,许多基因同时参与这两个过程。此外,发现宿主特异性致病适应限制了流行铜绿假单胞菌在不同宿主间的传播。
研究方法
1. 基因组数据集与克隆划分
• 收集全球32个研究的9829株分离株(含9573株临床样本),新测序样本提取DNA后在Illumina平台测序 。
• 通过比对参考基因组、严格质量控制和筛选,使用进行多位点测序分型,FastTree推断系统发育树,基于SNP距离(UPGMA聚类)、多基因序列分型(MLST)和阈值分配克隆,用Gubbins去除重组。
参考文章(点击下面蓝色字体跳转):
2. 分子测年与系统地理学
• BEAST 2.6.6进行贝叶斯时间重建
• TempEst评估时序信号,SPREAD分析洲际传播路径
• 估算克隆扩张时间(1850-2000年)与地理起源
3. 泛基因组分析
• 从短读数据组装基因组,用Panaroo聚类基因序列并推断图形泛基因组,进行简约祖先特征状态重建
• 比较流行克隆与散发株的基因获得/缺失
• EggNog-mapper进行功能注释
4. 表型与机制研究
• THP-1巨噬细胞感染实验(WT与CF-F508del细胞系)
• 斑马鱼感染模型(系统性感染与活体显微成像)
• 转录组分析:获取临床菌株基因表达数据,用Kallisto伪比对产生丰度估计,用DESeq2归一化并建模,用Wald检验评估系数,用k-均值聚类和置换检验评估转录多样性分布和聚类。
参考文章(点击下面蓝色字体跳转):
5. 适应性进化分析
• 用Treetime重建每个克隆中每个核苷酸位置的祖先特征状态 ,识别单核苷酸变化并注释变异效应,用Poisson检验评估每个基因的突变负担
• STRING数据框蛋白互作网络分析
• 用Fisher精确检验比较基因功能注释(Gene Ontology与实验验证)
推荐阅读
中国银河生信云平台(UseGalaxy.cn)以“让生信分析更简单”为使命。平台致力于为科研工作者、医疗机构和生物产业技术人员提供全栈式生物信息学分析解决方案。
优先技术响应、定制化工具部署、阶梯式能力培养,请加入「Galaxy生信星球」。咨询微信:usegalaxy 或 galaxy-help

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








