点击蓝字·关注我们
编辑:王凌琴 校对:林丽敏、朱琪
论文ID

原名:A functional selection reveals previously undetected anti-phage defence systems in the E. coli pangenome
译名:功能选择揭示大肠杆菌泛基因组中未被发现的抗噬菌体防御系统
通讯作者:Michael T. Laub
作者单位:Department of Biology, Massachusetts Institute of Technology, Cambridge
期刊:Nature Microbiology
发表时间:2022

研究背景
噬菌体是一种非常多样和普遍存在的病毒,对细菌构成几乎持续的威胁。噬菌体是地球上最丰富的生物实体,在某些环境中驱动着所有细菌中约20%的更替。细菌和它们的病毒捕食者被锁定在一场永久的共同进化战中,导致噬菌体操纵和利用宿主的复杂机制的出现,以及一套同样多样的细菌免疫机制,统称为抗噬菌体防御系统。近年来,基于防御岛基因组共定位的计算方法鉴定了许多抗噬菌体系统,但此类筛选并不能穷举。因此,识别额外的抗噬菌体防御系统有望为病毒和宿主之间古老的共同进化冲突提供新见解。本文采用功能性宏基因组学方法绘制大肠杆菌泛基因组防御系统的范围。揭示了大肠杆菌未表征噬菌体防御系统,还提供了在几乎任何细菌基因组或宏基因组样本适应系统识别噬菌体防御机制的筛选方法。在71株不同的E.coli揭示了21个此前未被表征的防御系统,同时阐述了完整的原噬菌体和可移动遗传元件是大肠杆菌防御系统的主要储存库和分配器。本文揭示了大肠杆菌抗病毒免疫的扩展前景,并为其他物种防御系统的定位提供新方法。
结果与讨论
01
新型抗噬菌体防御系统的鉴定
本文从ECOR标本中心收集了一组不同的野生分离菌株和19株临床同源菌株(共71株),71株菌株共编码21129个特异性基因簇,其中超过10000个基因组仅存在于一个或两个菌株中。本文基于gDNA构建了一个覆盖率为100倍的F黏粒文库,每个F黏粒在E. coli K12的衍生物EPI300中包含一个约40 kb的基因组片段,使用大插入的F黏粒最小化库大小,以包含潜在的大型防御系统。不同于Abi防御机制,本文使用 “tab”方法(T4流产)的选择策略,即将携带gDNA文库的细胞与噬菌体在结构化培养基(软琼脂)中以不同浓度的噬菌体混合。在中等浓度下,文库中的单个克隆在遇到噬菌体颗粒前可生长并形成微菌落,从而使种群的其余部分得以生存。因此,该筛选方法允许识别Abi和常规防御系统。
利用该策略,使用T4、λVir和T7攻击含F黏粒文库的细胞(图1)。从中分离出大约90个存活的克隆,然后对每个克隆的载体插入末端进行测序,以确定每个片段的基因组区域和起源菌株。对每个阳性克隆测量了斑块效率(EOP)(图2),EOP分析表明即使在噬菌体滴度大于109p.f.u. mL-1时,也显示出完全的保护作用。同时其他阳性克隆产生保护作用常伴随高比率的逃逸斑块,测序显示高频逃逸斑块克隆编码RM系统(图2)。基于此,257个初始克隆中的117个和9个被淘汰,推测是由于细胞表面特性的变化影响了吸附或RM介导的防御。考虑到剩余的131个克隆冗余,假设有43个克隆可以编码新的防御系统。作者将每个噬菌体对应的三组剩余的F黏粒集合,随机生成6-12 kb片段,并将其亚克隆到低拷贝质粒载体中,建立三个高覆盖率子库。随后选择该库细胞对各自的噬菌体进行侵染,并用纳米孔长读测序法对阳性克隆质粒进行测序。结果表明从选择中存活下来的子库克隆每个均包含最初选择的防御系统,两侧是来自最初F黏粒插入的随机长度的相邻DNA。因此,当reads映射回给定F黏粒中的基因组片段时,覆盖最大值通常描述了每个候选防御系统的边界(图2)。这与以前表征的系统相似,包括III型和IV型RM系统和一个古老的核酸内切酶家族,或解释噬菌体抗性的非防御基因。排除上述情况,筛出21个独特的候选防御系统,其中的10、6和5个系统分别来自T4、λvir和T7的选择。

图1. 鉴定噬菌体防御系统的选择策略

图2. 噬菌体防御系统的鉴定
02
候选防御系统的验证
为验证新防御系统,本文在野生型MG1655原生启动子控制下,将每个候选开放阅读框(ORF)或操纵子克隆到其低拷贝载体中,并用10个不同的噬菌体组成的群落对每个系统进行侵染(图 3a)。结果证实每个候选系统基本上降低了最初用于选择系统的噬菌体的EOP,并伴随其他系统EOP的降低。尽管大多数防御系统是相对特异性的,仅针对少数噬菌体提供保护,但部分系统提供了相对广泛的保护,如PD-λ-5。大多数系统对最初用于选择它的噬菌体保护作用最强。进一步对每个系统通过Abi或直接免疫进行分类,测试了21个系统在低多重感染(MOI)为0.05和5时感染的每个防御系统的菌株生长。其中9个为直接免疫,在两个MOIs下产生类似的保护作用;其余12个则采用Abi机制,在低MOI有较强的保护作用(图3)。验证了该筛选策略检测Abi防御的能力,并强调了Abi系统对大肠杆菌免疫有很强的贡献。
与算法筛选相比,该筛选策略的优势在于含有防御系统起源菌株,通过删除PD-T4-2和PD-T4-9的起源菌株ECOR65和ECOR22的候选系统,测试其在原生环境中是否对噬菌体有原生保护作用。发现T4菌株可以吸附但不感染,删除每一个系统显著提高T4的攻击效率,表明这些系统在其原生环境中和引入E.coli K12时都能提供防御(图3)。此外,作者还验证出ECOR13和E.coli C中的四个候选防御系统,测试表明,这两个菌株天生对这三种噬菌体敏感,所有的四个系统都提供了保护,表明所识别的系统的功能并不严格依赖于应变背景。
在鉴定的21个系统中,32个蛋白中有26个在GenBank中被注释为“假定蛋白”或含有功能未知的结构域,且与任何表征的抗噬菌体防御系统没有主序列同源性。为更灵敏地表征每个蛋白质,本文使用HHpred检测与PFAM结构域的远程相似性,但未显示出其与已知系统的同源性,然而检测出与防御系统的某些基序或结构域的潜在相似性(图3a)。远程同源性检测揭示了几个与已知防御系统不具备特征性的有趣特征,包括:(1)核糖体依赖性核糖核酸酶(RELE)结合噬菌体鞘样结构域的相似性;(2)编码外毒素A样结构域,一种未知蛋白和SecB样伴侣蛋白的三基因操纵子;(3)具有中心螺旋结构域的假定膜锚定蛋白(DUF4041)和一个C端DNA结合/裂解结构域;(4)融合到DNA融合/裂解结构域的β-螺旋;(5)P4噬菌体β样蛋白;(6)脂蛋白;(7)与功能未知的C端结构域融合的锌指状结构域;(8)CoiA结构域和(9)DUF6575。
与核酸酶和螺旋酶等结构域相比,部分防御系统较少与防御系统相关的结构域表现出相似性。这些较少出现的结构域包括一种肽酶,一种真核类Ser/Thr激酶,NAD+结合的Sir2同源物和一个GIY-YIG核酸酶。揭示了参与细菌防御有关的蛋白多样性,并突出了细菌抗病毒免疫的广阔而独特的前景。
11个防御系统具有提示DNA结合或切割活性的成分。值得注意的是,11个中有7个提供直接免疫,表面以潜在新方式的非自身核酸靶向活性。在21个系统中,有6个系统与HEPN基序或结构域有较远的相似性,且该结构域存在于与CRISPR-CAS和毒素-抗毒素系统相关的核糖核酸酶中,即它们是防御系统共同的、多功能的组成部分。进一步对预测结构域中的关键保守残基进行突变,发现它们对噬菌体保护至关重要,这表明远程结构域预测在系统功能中具有特征(图3d)。进一步,通过21个系统的发育分布评估系统的保守性(图3e)。发现每个系统的同源物在其他γ-变形菌门中均被发现,其中α-和β-变形菌门中分别有16和18个同源物,超一半的还存在于厚壁菌门、放线菌门、拟杆菌门和螺旋体门,这表明许多系统代表了新的、广泛分布的抗噬菌体防御系统。

图3. 21个未被表征的防御系统位点的综述和注释
03
可移动元件在大肠杆菌的防御系统中占主导地位
此前对防御岛的富集蛋白研究证实了38种新的防御系统。而鉴定的21个系统中均不是其同源物,只有一个组分(PD-T7-2)与先前验证的多组分系统的蛋白相似(32%同源性)。基于给定的算法还鉴定了7472个富含防御岛的蛋白家族,在鉴定的32个蛋白中,只有14个在有限的蛋白区域内与这些蛋白具有同源性,且同源性小于35%。表明基于本文的实验选择可能会发现不同类型的防御系统。
进一步,作者通过分析21个系统的原生基因组背景,发现有12个位于完整的原噬菌体中。其中7个系统位于P2类原噬菌体中,4个位于P2基因组的相同位置,直接位于编码P2复制内切酶和门脉蛋白的基因之间。最近,该位置发现编码了一系列此前未被表征的防御系统,在P2类噬菌体中观察到第二个防御富集位点,该位点包含了本文发现的三个系统,以及此前在P2参考基因组中鉴定的防御基因fun(图 4a)。值得注意的是,本文的71个大肠杆菌菌株中有47株共编码63个P2门脉蛋白,其中110个独特的蛋白存在于邻近热点。研究表明,P2原噬菌体不仅编码丰富多样的抗噬菌体蛋白,同时其防御热点构成大肠杆菌免疫图谱的相当大部分(图4b)。
此外还发现5个防御系统与其他类型的原噬菌体或其残体相关,包括P4卫星原噬菌体或相关整合酶、Mu样噬菌体和λ类噬菌体。同时在一个整合共轭元件(ICE)中观察到一个防御富集位点,包含已识别的两个系统PD-T4-4和PD-T7-2(图4a)。PD-T7-4及其同源物通常与整合酶基因重叠,而PD-T4-5则位于质粒上。
研究支持以前的发现,即除了防御岛,原噬菌体和其他MGE也是一个丰富的防御系统库。然而,这些类别并不相互排斥,因为部分防御岛可能由MGEs携带或衍生而来。为记录在此鉴定系统的不同基因组背景,收集了21个系统中每个中844603个公开可用的细菌基因组中的所有同源物,检测到两种不同的模式,对于在本文菌株中发现的除原噬菌体之外的系统,同源物也与原噬菌体不相关(图4c),且通常靠近编码防御相关结构域的其他基因。因此,这些系统确实出现在防御岛中,尽管此前未发现其在防御岛富集。对于在大肠杆菌原噬菌体中识别的系统,其部分同源物也在原噬菌体中发现(图4d)。
值得注意的是,在防御岛中发现了鉴定系统的同源物(图4e),表明它们不需要原噬菌体环境来发挥作用。且原噬菌体中常见的系统同源物邻近防御相关基因的数量与非同源系统的数量呈负相关(图4f,g)。突出了原噬菌体或在给定热点内可携带多少防御系统受包装的DNA的大小限制。最后发现,对比于算法预测系统的防御岛和原噬菌体,发现实验选择的系统与已知防御基因的关联频率平均较低,但与原噬菌体基因的关联频率较高,表明防御岛富集方法在鉴定原噬菌体防御系统上灵敏度不高。
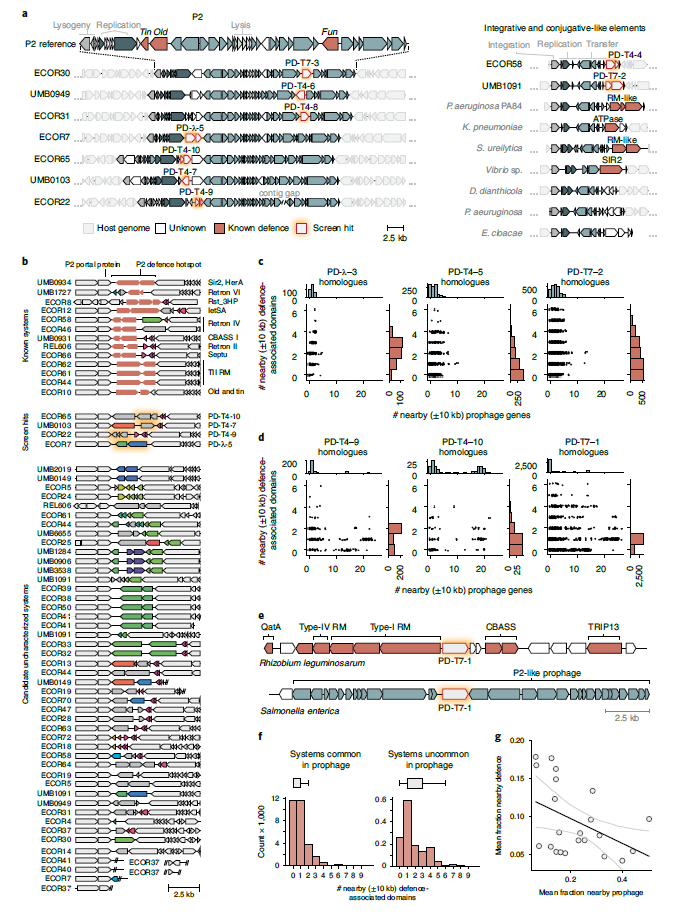
图4. 原噬菌体和MGE是防御系统的主要来源
04
未被表征的毒素-抗毒素系统
毒素-抗毒素(TA)系统通常由一种蛋白质毒素组成,可抑制细胞生长,但可被同源共表达抗毒素中和。TA系统在细菌基因组和MGEs中非常普遍,但其功能仍未可知。文中选择产生的四种不同体系,依据本质可识别为TA类,编码的基因产物与毒蛋白序列相似,具有多组分特征,并提供Abi防御功能(PD-T4-5、PD-T4-7、PD-T4-9和PD-λ-2),第五个(PD-T4-10)促进Abi防御,有两个重叠的ORF。以上所述系统以前均未被注释为TA系统,通过诱导启动子中表达每个组分进一步验证三个具有多组件的系统:PD-T4-10、PD-λ-2和PD-T4-9(图5a).
对于PD-T4-10的两个组分,PD-T4-10B具有毒性,当PD-T4-10A共表达时,毒性被完全中和(图5b)。因此作为抗毒素的PDT4-10A不能被删除,而毒素PD-T4-10B的删除则消除了对T4感染的抗性(图5c)。
对于PD-λ-2的三个组分。PD-λ-2A类似抑制翻译的HigB毒素,PD-λ-2B具有融合到C-末端肽酶结构域的HigA抗毒素样结构域(Xrefamilyhelix-turn-helix),PD-λ-2C与P4噬菌体抗毒素有关。过度表达的PD-λ-2B具有毒性,但可与PD-λ-2A共表达缓解(图5d)。且PD-λ-2C不是中和毒素所必需的,但为防御λVir所必需的(图5e)。
对于PD-T4-9,包含第三个类似SecB的分子伴侣,称为毒素-抗毒素-伴侣(TAC)系统并命名为CmdTAC。TAC系统的抗毒素C端的非结构化扩展称为伴侣成瘾结构域(ChAD),在缺少同源SecB类分子伴侣的情况下,ChAD使抗毒素易于聚集和蛋白水解,从而释放毒素。诱导CmdT的表达是有毒的,与推测的抗毒素CmdA共表达仅能略提高存活率。然而,与假定的抗毒素和伴侣成分共表达则可完全恢复活性(图5f)。表明CmdTAC是一种新的抗噬菌体的TAC系统。
进一步在CmdTAC+细胞的T4感染过程中过量产生伴侣蛋白CmdC。发现CmdC的过量供应使CmdTAC破坏其对噬菌体的保护,表明CmdC的破坏或CmdC从复合物中螯合可激活TA系统以应对噬菌体感染(图5g)。该伴侣通常促进CmdA对CmdT的中和作用,但感染后,可能会被噬菌体产物耗尽或螯合,导致毒素释放和流产感染。提供额外的CmdC可防止分子伴侣的损失或完全螯合,从而防止CmdA释放CmdT。但分子伴侣-噬菌体相互作用的机理需进一步研究。
进一步克隆E. coli C496_10的MQSRAC系统测试其他TAC系统是否能保护E. coli MG1655抵抗噬菌体感染。尽管MQSRAC在毒素和抗毒素序列上与CmdTAC完全无关,但它是一个典型的TAC系统,且已在分枝杆菌中表征,包括一个与CmdC同源的SecB样伴侣。该系统对T2提供了强有力的保护(图5h)。与CdmTAC相似,MqsR的毒性只能通过表达MqsA和MqsC(图5i)来解除。因此表明TAC系统可能是一种广泛而多样的新型噬菌体防御系统。
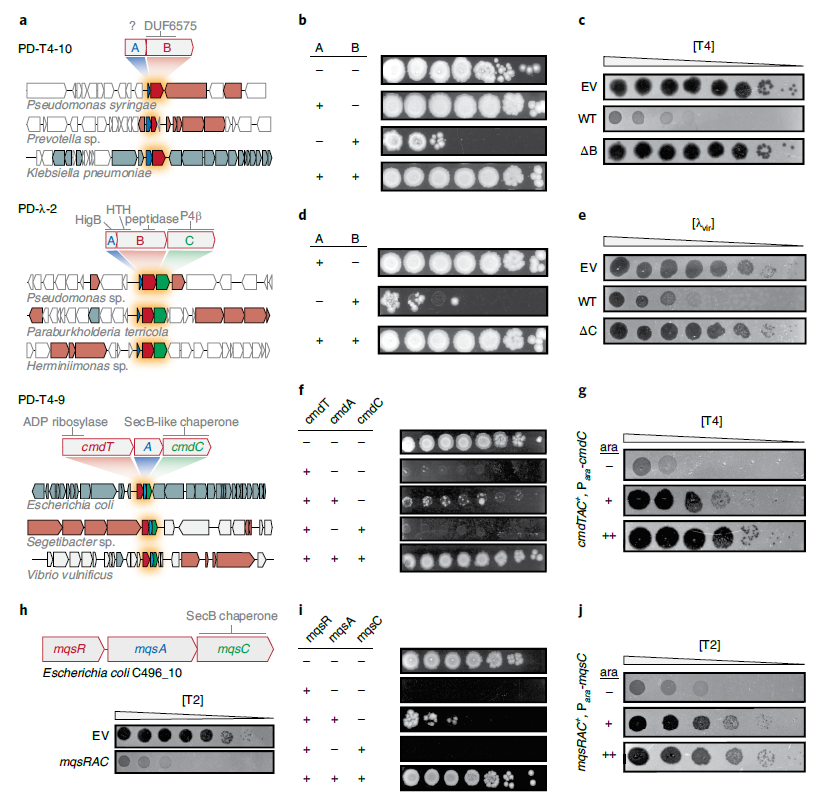
图5. 未被表征的毒素-抗毒素衍生的防御系统
结论
在大肠杆菌的泛基因组中分布着大量不同的、以前未知的噬菌体防御基因。像许多细菌一样,大肠杆菌的不同菌株的“附属”基因组有巨大的变异性。尽管在接近已知系统的基础上寻找新的防御系统的努力已被证明富有成效,但至今许多噬菌体保护系统仍未确定。本文开发的方法可以通过多种方式进行有力的扩展。首先,来自其他来源的基因组DNA,包括宏基因组DNA,皆可用作输入材料。其次,本文测试的噬菌体组仅限于三个,可进一步扩展,特别是考虑到噬菌体的多样性。最后,仅需小的修饰,任何可转化的细菌都可用作宿主菌株。细菌免疫系统的进一步鉴定和表征有望为细菌与其病毒捕食者之间的古老军备竞赛提供新的线索,并且还可能具有实际应用,为精确的分子工具提供基础,并有助于为将来开发噬菌体作为治疗剂的努力提供信息。

点击阅读原文查看更多
*:本文如有不足,请批评指正
EMBLab
EMBLab
环境微生物组与生物技术实验室
环境微生物组学研究环境中全部微生物及其遗传信息,其方法学基础与理论拓展应用是国际学术前沿和热点。西湖大学环境微生物组与生物技术实验室开展环境工程学与微生物学交叉学科研究,研究兴趣包括:1)环境工程与合成微生物组学;2)微生物组的群落构建理论与功能原理;3)抗生素耐药性产生与传播机制及风险监控;4)持久性有机污染物(塑料与药物)降解转化及健康效应;5)废水处理中磷资源利用与回收。

EMBLab成员
实验室目前招聘:
环境合成生物学、合成微生物组学及相关方向【副研究员】或【助理研究员】 1名,【博士后】 1 名。微生物组学、异源物质降解、废水磷资源回收方向【科研助理】2名。
欢迎大家与我们联系。
联系邮箱:
jufeng@westlake.edu.cn
xuyisong@westlake.edu.cn

鞠峰
EMBLab负责人
鞠峰,西湖大学研究员、博士生导师,环境微生物组与生物技术实验室(EMBLab)负责人、浙江省海岸带环境与资源研究重点实验室副主任。现任西湖大学工学院特聘研究员(环境学)、西湖实验室PI,兼聘生命科学学院(生物学),开展环境微生物组学研究。目前担任Frontiers in Microbiology副主编、中国工程院院刊Engineering、Engineering in Life Science等SCI期刊编委,以及Critical Reviews in Environmental Science Technology、Journal of Environmental Sciences、Environmental Science & Ecotechnology、The Innovation、iMeta等期刊青年编委,曾担任加拿大自然科学与工程理事会(NSERC)国际评审专家。曾获Engineering“编委年度贡献奖”(2021)、中国生态学会“水云天微生物生态青年科技创新奖-特等奖”(2018)、香港科学会“青年科学家奖”(2016)、香港大学“杰出研究生奖”(2015)等奖项。近三年主持或参与国家级或省部级科研项目4项(含国家重点项目1项、省杰青1项)。目前参编中英文专著6本,以第一或通讯作者在Nature Communications、ISME Journal、Advanced Science、Environmental Science & Technology、Water Research、Environment International国际知名学术期刊等发表SCI论文60余篇,引用 4200余次。
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








