点击蓝字 关注我们
构建肝脏转录组学细胞特异性表达模型结合临床参数解析慢乙肝患者肝内炎症的形成因素

研究论文
● 期刊: iMeta (IF 23.7)
● 原文链接DOI: https://doi.org/10.1002/imt2.221
●2024年7月4日,深圳国家感染性疾病临床医学研究中心卢洪洲、德国埃森大学医院病毒研究所陆蒙吉、复旦大学附属华山医院国家传染病医学中心张继明和江南大学附属无锡五院临床医学研究中心王俊等团队在iMeta在线联合发表了题为“Cell-type-specific expression analysis of liver transcriptomics with clinical parameters to decipher the cause of intrahepatic inflammation in chronic hepatitis B”的研究文章。
● 本研究结果表明,巨噬细胞分泌趋化因子如 CCL20,招募耐受的 T 细胞进入肝组织;同时,肝细胞的先天免疫受到抑制,阻碍了 ISGs 的抗病毒效应。
● 第一作者:王俊、李倩
● 通讯作者:卢洪洲 (luhongzhou@szsy.sustech.edu.cn)、陆蒙吉 (mengji.lu@uni-due.de)、Daniel Hoffmann (daniel.hoffmann@uni-due.de)、张继明(jmzhang@fudan.edu.cn)
● 合作作者:邱源旺、Simo Kitanovski、汪成、张晨霞、李发红、李晓光、张振锋、黄利华
● 主要单位:深圳市第三人民医院,国家感染性疾病临床医学研究中心;杜伊斯堡-埃森大学,埃森大学医院病毒学研究所;江南大学附属无锡五院,临床医学研究中心 ;杜伊斯堡-埃森大学,生物信息学与计算生物物理学中心;复旦大学附属华山医院,国家传染病医学中心
亮 点
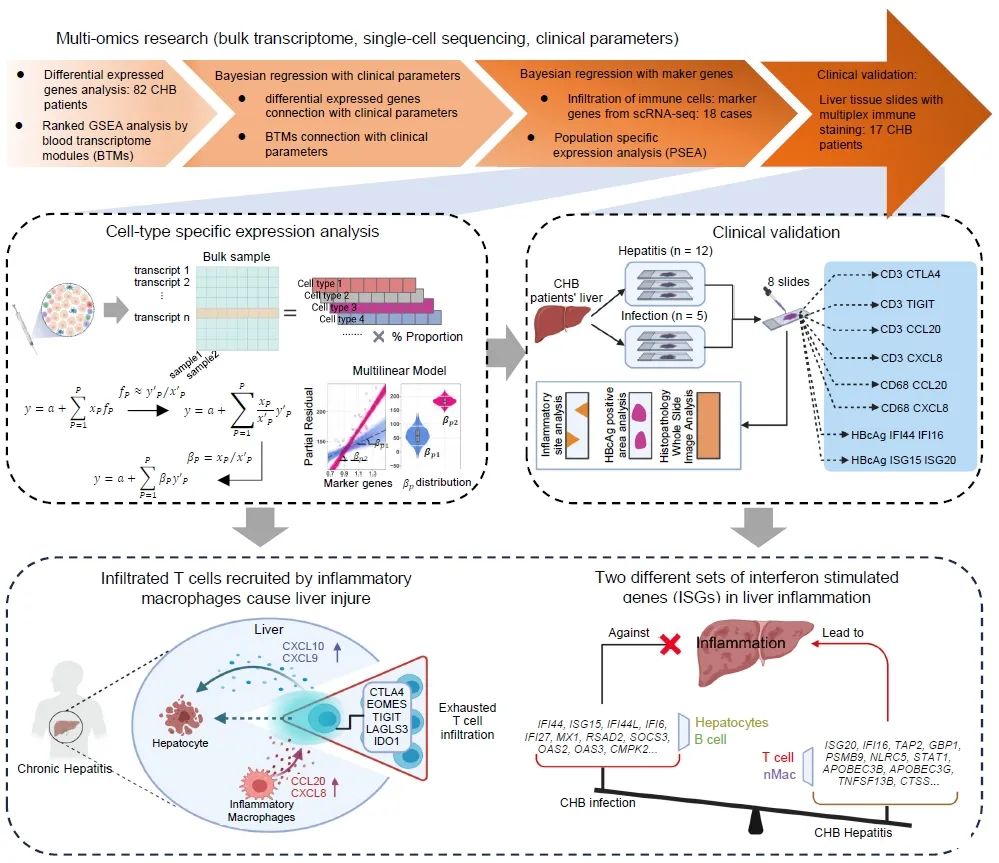
● 本研究结合肝脏穿刺组织转录组数据、单细胞测序数据和临床数据,通过贝叶斯回归分析,从多组学角度探索慢性乙型肝炎(CHB)肝内炎症的形成因素;
● 巨噬细胞分泌如CCL20和CXCL8等趋化因子,招募免疫耗竭型T淋巴细胞 (高表达CTLA4、TIGIT等)进入肝脏组织;
● 慢乙肝炎症期,肝细胞内的先天免疫受到抑制,阻碍了干扰素刺激基因(ISGs)启动抗病毒效应;
● 炎症期浸润肝组织的T细胞和巨噬细胞中天然免疫通路的激活,如干扰素通路的活化,进一步加剧了炎症的形成。
摘 要
由于缺乏针对肝内炎症的直接干预方法,实现慢性乙型肝炎(CHB)的功能性治愈仍然充满挑战。多组学研究方案则为深入理解CHB的肝脏炎症机制提供了可能性。本研究利用贝叶斯线性模型将基因表达与临床参数联系起来,通过群体特异性分析(PSEA)将整体基因表达细化到不同临床阶段的特定细胞类型。利用这些模型我们分析关键生物学因素,如炎症细胞、免疫活化、T细胞耗竭、趋化因子及其受体和干扰素刺激基因(ISGs)。此外,我们进一步通过对CHB患者肝组织样本的多重免疫染色验证了分析结果。在CHB患者中,免疫细胞活化和迁移相关的基因表达增加。巨噬细胞、T细胞、免疫负调节因子、趋化因子和ISGs等基因与血清丙氨酸氨基转移酶(ALT)水平呈正相关,但与HBV DNA水平不相关。PSEA模型确认T细胞为细胞耗竭相关的调节因子的来源,而巨噬细胞主要贡献了趋化因子的表达。在肝脏炎症期,上调的ISGs(ISG20、IFI16、TAP2、GBP1、PSMB9)与T细胞和巨噬细胞浸润相关,并与ALT水平呈正相关。相反,另一组主要由肝细胞和B细胞表达的ISGs(IFI44、ISG15、IFI44L、IFI6、MX1)与ALT的水平无关。我们的研究通过整合 CHB 患者的肝内转录组学、单细胞测序数据和临床数据,解析了 CHB 中肝内炎症的形成因素。研究结果支持,巨噬细胞分泌趋化因子如 CCL20,招募耐受的 T 细胞进入肝组织;同时,肝细胞的先天免疫受到抑制,阻碍了 ISGs 的抗病毒效应。
视频解读
Bilibili:https://www.bilibili.com/video/BV1EJ4m1M7V6/
Youtube:https://youtu.be/KPnH94G3sys
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
全球约有2.6亿人感染乙型肝炎病毒(HBV),每年因肝硬化和肝细胞癌导致超过100万相关死亡人数。在HBV的持续感染中,宿主免疫反应无法控制病毒,可能导致进行性肝损伤、肝硬化和癌症。然而,慢性乙型肝炎(CHB)中肝脏炎症形成的机制尚未完全了解。虽然核苷(酸)类似物联合干扰素等抗病毒疗法被广泛使用,但这些治疗方法无法根除病毒,由于缺乏直接针对肝脏炎症的干预方法,实现CHB的功能性治愈仍是一大挑战。
多组学研究是目前揭示肝脏炎症形成机制的最有前途的方法之一。最初,Vanwolleghem等人在CHB患者的外周血转录组数据中发现,在免疫活动期B淋巴细胞和天然免疫反应被激活,而T淋巴细胞在CHB的所有阶段都被激活。Lebosse等人进一步在CHB患者的肝组织中进行了转录组研究,报道了在HBeAg阳性患者中,天然免疫相关干扰素刺激基因 (ISGs),如CXCL10、GBP1、IFITM1、IFNB1、IL10、IL6、ISG15、TLR3、SOCS1和SOCS3,受到了抑制。2021年,van Buuren等人报道,在免疫活动期的CHB患者中,干扰素信号通路上调,肝内免疫细胞数量增加,强调了免疫调节在实现HBV功能性治愈中的重要性。2022年,Montanari等人也报道,在CHB的免疫活动期,干扰素刺激基因、免疫检查点基因(CTLA4、PDCD1、ICOS)和趋化因子基因(CXCL9、CXCL10)上调。由于基因的上调可能是由于肝组织中免疫细胞数量增加,导致与免疫细胞相关的基因平均表达水平增加,因而使用平均表达量的分析方法则无法在细胞水平上完全阐明参与炎症的免疫通路以及ISGs在不同细胞类型中的特定表达差异。单细胞测序技术则提供了一种可能有效方法来解决上述问题。2023年,Zhang等人对来自23名CHB患者不同临床阶段的肝组织CD45+免疫细胞进行了单细胞测序。他们发现免疫耗竭型CD8+ T细胞和FCGR3A+巨噬细胞在炎症形成中的重要作用。然而,在免疫活动期的患者样本量较少(五例),这使得在构建与临床数据的相关的统计模型时存在挑战。
基于上述背景与领域研究局限,本研究采取将肝脏组织转录组数据与单细胞测序数据和临床数据相结合,从多组学角度分析CHB中肝脏炎症的形成因素。我们开发了一个贝叶斯线性模型,用于研究基因表达和临床参数之间相关性。此外,通过从CHB患者和健康肝组织获得的单细胞测序数据筛选出细胞类型标志基因,我们进一步更新了群体特异性表达分析方法(PSEA),在不同临床阶段将整体基因表达拆分为特定细胞类型的表达。我们将这两个模型整合到一起分析之前报道的与肝脏炎症相关的免疫过程,如炎症细胞浸润、免疫细胞活化、T细胞耗竭、趋化因子及其受体表达、先天免疫活化和干扰素刺激基因(ISGs)表达,绘制了一张更全面的CHB肝脏炎症发生图谱。此外,通过对CHB患者肝组织样本进行多重免疫染色加以验证。
结 果
CHB患者在四个临床阶段的聚类
前期已从临床筛选出82名CHB患者,并进行了肝穿刺样本的组织学检查,排除了脂肪肝和其他肝病。CHB患者依据乙型肝炎病毒e抗原(HBeAg)状态、HBV病毒血清载量和丙氨酸氨基转移酶(ALT)水平的变化分为4个自然史时期。在HBeAg阳性的慢性感染中,HBV DNA血清载量往往较高,但没有肝炎。相反,在HBeAg阳性的慢性肝炎患者中,HBV血清DNA载量通常低于前者,但是血清ALT升高,肝脏出现明显炎症。本研究中,我们使用HBeAg状态、坏死性炎症评分(G评分)、HBV DNA水平和血清ALT水平将患者分为四组:19名HBeAg阳性慢性感染患者,38名HBeAg阳性慢性肝炎患者,11名HBeAg阴性慢性感染患者,以及14名HBeAg阴性慢性肝炎患者(图1A)。HBeAg阳性和阴性慢性感染患者的G评分较低(0−1分)。HBeAg阳性慢性感染患者的病毒DNA载量较高(107–109拷贝/mL),但ALT水平正常。大多数HBeAg阴性慢性感染患者的DNA低于检测限,只有两名患者的水平较高,但也维持较低水平,低于2 × 103拷贝/mL。除三名患者外,所有慢性感染患者的ALT水平均正常。对于慢性肝炎患者,大多数(78%)的G评分为2−4分,HBV DNA载量为103−107拷贝/mL,所有慢性肝炎患者的ALT水平均升高(> 40 IU/mL)(图1A)
慢性肝炎中上调的肝内基因
主成分分析(PCA)结果显示,根据各组间差异表达基因(DEGs),可以将慢性肝炎患者与慢性感染患者区分开来(图1B)。然而,患者的HBeAg阳性或阴性状态在PCA图中并未明显区分。我们将排名前35的差异表达基因进行GO分析。结果显示与免疫过程相关的通路被显著富集(图1D)。进一步的基因富集分析揭示(图1E),在多种已知的肝损伤途径中(包括焦亡、程序性坏死、凋亡和自噬性细胞死亡),凋亡相关的通路具有最高的富集分数。这些发现确认了宿主免疫反应与慢性肝炎阶段肝损伤之间的关联。
为了将DEGs注释到CHB肝脏中的特定免疫功能和细胞类型特异性过程,我们使用了外周血转录组模块(BTMs)的富集分析。每个BTM包含一组具有相似表达模式并注释为相同生物功能的基因。GSEA使用按FC预排序的基因列表对所有BTMs进行富集。与ALT和AST水平正相关的富集BTMs包括细胞周期、抗原呈递、T细胞、单核细胞/树突细胞、NK细胞、趋化因子、Toll样受体(TLR)信号、补体、DNA感应和IRF2靶基因模块。大多数B细胞相关的BTMs与ALT或AST水平之间没有明显的相关性。

图1. 慢性乙型肝炎(CHB)患者在四个临床阶段的聚类
(A) CHB患者在四个临床阶段中的无监督聚类。(B) 差异表达基因的主成分分析。(C) 82名CHB患者在四个临床阶段的肝内整体转录分析到细胞类型特异性分析的流程图。(D) 根据倍数变化(肝炎期vs感染期)进行基因集合富集分析(GSEA)。Chord图关联了前35个上调基因和前10个富集的GO通路。(E) 根据GSEA标准化富集评分和倍数变化进行肝损伤通路的富集分析。通过临床参数将82名CHB患者无监督地聚类为四个自然史时期:免疫耐受期(HBeAg+ 感染)、免疫活动期(HBeAg+ 肝炎)、非活动携带期(HBeAg- 感染)和再活动期(HBeAg- 肝炎)。差异基因表达分析和富集分析揭示了在肝脏炎症期上调的基因主要富集在与免疫相关的通路中,特别是与T淋巴细胞招募相关的通路。
定义肝内各细胞类型的标志基因
从人类肝脏获取的三个10×基因组单细胞RNA测序(scRNA-seq)数据集(GSE115469,GSE186343,GSE124395),合并后使用ST_transform方法进行标准化处理。该数据集包括6例慢性乙型肝炎(CHB)肝脏炎症阶段和12例健康供体肝脏(图2A,2B)。使用UMAP数据降维的方法映射和可视化了13种大的细胞类型,并分别被标记为肝细胞、胆管细胞、T细胞(αβ T细胞、γδ T细胞1、γδ T细胞2、NK样细胞)、B细胞(B细胞、浆细胞)、巨噬细胞(非炎症性和炎症性)、以及内皮细胞,它们代表了肝脏中的主要细胞亚群。T细胞在CHB患者肝脏中的比例较高(图2C)。αβ T细胞在肝脏T细胞中占据最大的一部分,并且它们在CHB肝脏中的数量比正常供体肝脏中更多。已知的标志基因(CD8A、CD3D、NKG7、GZMB、CD68、CD163、S100A8、CD79B、IGKC、ALB、PCK1和CYP2E1)被投影到UMAP图上(图2D)。使用层次聚类法识别肝内细胞类型标志基因,共发现92个标志基因(图2E)。

图2. 基于单细胞RNA测序数据的肝脏细胞类型特异性标志基因
(A) 左图:使用UMAP对肝脏10×基因组scRNA-seq数据的可视化和聚类。右图:使用UMAP映射和可视化的二十种细胞类型。(B) 左图:代表人类肝脏主要细胞类型的聚类。右图:CHB患者和健康供体肝脏中主要细胞类型的比例。(C) T细胞、巨噬细胞和B细胞亚型的比例。(D) 已知标志基因在UMAP图上的投影。(E) 通过层次聚类对肝脏主要细胞类型的92个肝内标志基因进行热图分析。使用UMAP识别并可视化了十三种细胞类型。这些聚类包括肝细胞、胆管细胞、T细胞亚型(αβ T细胞、γδ T细胞1、γδ T细胞2、类似NK细胞)、B细胞(B细胞、浆细胞)、巨噬细胞(非炎症性和炎症性)和内皮细胞,代表了肝脏中的主要细胞群体。CHB患者肝脏中的T细胞比例显著增加,其中αβ T细胞是最丰富的T细胞亚型,且其数量在CHB肝脏中较正常供体肝脏显著升高。
慢性乙型肝炎(CHB)中的免疫细胞特征及其与肝损伤的相关性
在肝炎阶段的肝组织样本中,T细胞(αβ T细胞、γδ T1、γδ T2)、NK样细胞、成熟B细胞、浆细胞B细胞以及炎症性和非炎症性巨噬细胞(iMac和nMac,分别)的标志基因均表达上调。与慢性感染的样本相比,肝细胞标志基因的表达在肝炎患者中较低(图3A,左侧)。细胞标志基因表达与血清ALT水平和HBV DNA载量的关系图显示,ALT与大多数T细胞、iMac和nMac细胞标志基因表达呈正相关,而与肝细胞标志基因表达呈负相关(图3A,右侧)。对四例慢性肝炎患者的肝组织切片进行多重免疫组化染色,结果显示淋巴细胞在胆管浸润部位的比例最高,且该部位的T细胞和B细胞比例大于整张切片(图3B)。CHB肝炎患者肝内特征在于肝内各种免疫细胞的浸润(图3C)。
在肝炎阶段肝脏中免疫相关基因的表达增加可能是由于免疫细胞浸润增多或免疫细胞激活导致胞内基因表达上调。之前的研究表明,线性模型可以用于大多数基因的差异表达分析。特定细胞类型的基因表达可以描述为与该群体大小呈线性关系。在临床样本中准确测量不同细胞类型的群体大小通常是不可能的。然而,Kuhn等人开发了一种多重线性回归方程,根据标志基因的表达水平描述特定细胞类型的相对细胞群体大小。我们进一步发展为贝叶斯多重线性回归模型,一个相对于特定标志基因的 β 值代表某一细胞类型中标准化的基因表达。图 3D 显示了 HBeAg 阳性感染阶段和相应的肝炎阶段的最小二乘拟合,其中直线的斜率是从 PSEA 方程中得出的 β 值。小提琴图显示了不同临床阶段每个基因的斜率分布及其 75% 置信区间(图 3E)。

图3. 将整体基因表达解析为特定细胞类型在不同CHB阶段的人群特异性表达分析
(A) 左图:显示82名CHB患者(左侧列)肝脏中13种主要细胞类型的标记基因(行),分别在肝炎(G3S3,炎症病理评分和阶段:G分数=3,S分数=3)和感染阶段(G1S1,炎症病理评分和阶段:G分数=1,S分数=1)中的表达情况(顶部的树状图)。右图:血清参数(ALT、HBV DNA)与标记基因表达之间的斜率(边际后验的95%可信区间);红色和蓝色泡泡分别表示持续为正和负斜率(B) 顶部:来自4名CHB患者的一个代表性肝切片的多重免疫染色,显示CD3、CD68、CD20和NKP46的表达情况。底部:来自四个CHB肝脏的代表性图像中CD3+、CD68+、CD20+和NKP46+细胞在整个标本和炎症区域中的比例。(C) 免疫细胞从门脉区浸润到肝脏的示意图。(D) 顶部:来自肝活检样本的整体转录组,显示不同细胞类型的百分比。底部:通过标记基因的表达水平描述相对细胞种群大小的多线性方程(即特定细胞类型的表达分析,PSEA)。(E) 局部残差图和组别中拟合的线性模型,蓝色代表感染阶段,红色代表肝炎阶段的相应最小二乘拟合线。小提琴图显示了β值分布及其各基因在两个阶段的75%置信区间,用于比较感染阶段(蓝点)和肝炎阶段(红点)中基因的特定表达。由于炎症期间免疫细胞浸润增加,基因上调源于细胞活化和免疫细胞数量增加,导致免疫细胞特异性基因表达水平升高。传统方法无法辨别这些对基因表达的影响。而我们更新的PSEA方法到基于贝叶斯多线性回归模型,利用特定细胞标记基因参考进行分析,评估不同临床阶段特定细胞类型的基因表达差异(在不同临床阶段的 β 值变化)。
趋化因子和免疫耗竭标志物与肝损伤密切相关
了解相关免疫调节分子、细胞因子和趋化因子的表达是否与血清ALT水平和/或HBV DNA载量相关联很重要。贝叶斯线性模型结果显示免疫负调基因,相关趋化因子及受体基因,C1Q家族的基因和特定细胞因子的基因表达与ALT和AST水平呈正相关,与血清HBV DNA水平不相关(图4A-B)。所有正相关基因均在CHB炎症期患者肝内高表达。通过PSEA,我们将这些基因的表达拆解到不同的细胞类型和临床阶段。根据这一分析,我们发现:(i)免疫负调节基因,包括CTLA4、EOMES、LGALS3、TIGIT、IDO1和CD274等的表达主要来自T细胞, HAVCR2和LGALS9主要来自非炎性巨噬细胞,BTLA来自于B细胞;(ii)趋化因子基因包括CXCL9、CXCL10、CXCL11、CCL5、CCL8、CCL4和CCL18等主要表达于T细胞;(iii)趋化因子基因CCL20和CXCL8的表达主要来自iMac和T细胞(图4B,左侧)。绘制的图表显示了从HBeAg阳性感染阶段到肝炎阶段的系数变化(β值)(图4B,右侧)。
在肝炎和感染阶段的T、iMac和nMac细胞中,使用部分残差图和斜率(β值)分布来比较选定基因的表达。在慢性肝炎患者的T细胞中,CXCL9、CXCL10、CXCL11和TIGIT的斜率较高,高于HBeAg+慢性感染和HBeAg-慢性感染患者(图5C),表明这些基因的表达在相应细胞类型中增加。作为T细胞耗竭指标的CTLA4、EOMES、LGALS3的斜率分布在慢性肝炎患者中明显高于HBeAg-慢性感染患者,但在HBeAg+慢性感染患者中通常重叠(图4C)。类似地,nMac中HAVCR2和LGALS9的基因表达在慢性肝炎患者中与HBeAg+慢性感染患者互相重叠(图4D)。因此,iMac中CCL20和CXCL18的β值在慢性肝炎患者中显著高于慢性感染患者(HBeAg-和HBeAg+)(图4D)。
总体而言,我们的研究表明趋化因子,包括CXCL8和CCL20,在iMac中显著表达,促进了肝脏对T细胞的招募,这些T细胞表现出较高的CTLA4、EOMES、TIGIT、LAG3和IDO1的基因表达(图4E)。招募的T细胞进一步释放趋化因子CXCL9和CXCL10,可进一步促进T细胞的浸润。

图4. T细胞和巨噬细胞中与肝损伤相关的趋化因子和免疫负调节分子
(A) 左图:显示CHB四个阶段中所有82名患者(列)的免疫调节、趋化因子、补体、细胞因子和干扰素基因表达的热图(行)。右图:左侧中基因表达的ALT和AST斜率的95%可信区间。彩色泡泡表示正斜率(红色)或负斜率(蓝色)。(B) 左图:慢性肝炎患者中不同参考细胞类型基因的PSEA系数值 (β_P) 的雷达图。右图:比较慢性肝炎与HBeAg阳性慢性感染期间不同参考细胞类型基因系数的变化。(C, D) 局部残差图及拟合后的后验线和慢性肝炎(红色)及HBeAg阳性慢性感染(蓝色)中来自拟合线的斜率分布小提琴图,显示T细胞和iMac细胞对基因表达的贡献。利用建立的PSEA模型,分析了上调的免疫检查点基因和趋化因子基因,以识别其在不同临床阶段中的主要基因表达的细胞类型和表达变化。(E) 我们的研究结果表明,在免疫活跃期间,免疫检查点基因(如CTLA4、EOMES、TIGIT、LGALS3)主要由浸润的T淋巴细胞表达,暗示T细胞衰竭推动了炎症过程。此外,与ALT水平强相关的趋化因子基因,如CCL20和CXCL8,主要由巨噬细胞表达,表明巨噬细胞分泌趋化因子以招募免疫耗竭的T淋巴细胞进入肝组织。(F) 临床组织病理学验证工作流程:分析来自12名慢性肝炎阶段和5名感染期慢性HBV患者的肝切片。在8个连续切片上进行多重免疫染色,使用蓝框中列出的参数。机制图和流程图使用Biorender创建的(https://biorender.com/)。
T细胞耗竭标志物及巨噬细胞活化相关趋化因子的病理验证
为了验证多组学分析得到的结果,我们将来自17名慢性乙型肝炎(CHB)患者的肝组织切片分为两个不同的临床组:12名HBeAg阳性肝炎患者和5名HBeAg阳性但无肝脏炎症的感染患者。我们在这些切片上进行了CTLA4和TIGIT与CD3的双重免疫组化染色。在炎症部位,CTLA4和TIGIT与CD3共定位,表明在肝炎组中,耐受的CD3+ T细胞广泛浸润肝脏的炎症部位(图5A-D)。
此外,我们还在来自HBeAg阳性肝炎患者的肝穿刺样本上进行了CCL20和CXCL8与CD68和CD3的双重染色。在炎症部位,CCL20广泛表达于CD68+的巨噬细胞(区域I),并部分表达于CD3+ T细胞(区域II)(图5E, F)。类似地,CXCL8在CD3+ T细胞中部分表达(区域II),并且在CD68+的巨噬细胞中表达较高(区域I)(图5F)。CXCL8在相同区域的表达比CCL20低。

图5. T细胞耗竭标志物和巨噬细胞活化趋化因子的病理验证
(A) HBeAg阳性肝炎患者和HBeAg阳性感染患者肝标本中CTLA4与CD3的双重免疫组化染色。(B) 显示CHB肝炎、CHB感染和CHB肝炎炎性浸润部位的CTLA4+和CD3+CTLA4+细胞的柱状图。(C) HBeAg阳性肝炎患者和HBeAg阳性感染患者肝标本中TIGIT与CD3的双重免疫组化染色。(D) 显示CHB肝炎、CHB感染和CHB肝炎炎症浸润部位的TIGIT+和CD3+TIGIT+细胞的柱状图。(E) HBeAg阳性肝炎患者的肝标本中CCL20(红色)与CD68(绿色)和CD3(绿色)的双重标记。(F) 显示CHB肝炎、CHB感染和CHB肝炎炎性浸润部位的CCL20+、CD3+CCL20+和CD68+CCL20+细胞的柱状图。(G) HBeAg阳性肝炎患者的肝标本中CXCL8(红色)与CD68(绿色)和CD3(绿色)的双重标记。(H) 显示CHB肝炎、CHB感染和CHB肝炎肝内炎症浸润部位的CXCL8+、CD3+CXCL8+和CD68+CXCL8+细胞的柱状图。CTLA4和TIGIT与CD3在炎症部位共定位,表明在肝炎期间,耐受的CD3+ T细胞广泛浸润肝脏的这些部位。CCL20和CXCL8等趋化因子的释放主要来自CD68+巨噬细胞,在肝炎组和CD3+ T细胞浸润的炎症部位表达高水平,促进肝内炎症的形成。
上调的干扰素刺激基因在肝炎炎症中的不同作用
在HBV慢性感染中,干扰素刺激基因(ISGs)是否被激活仍然不是很清楚。为了探究ISGs在慢性HBV感染中的作用,我们在CHB肝脏炎症期和感染期(无肝内炎症)的患者肝脏组织样本中比较了341个ISGs的表达情况。其中,79个ISGs在CHB肝炎患者中表达上调,包括STAT1、STAT2、ISG15、ISG20、APOBEC3G、IFI16、IFI35、IFI44、OAS2和MX1(图6A)。我们使用PSEA的方法分析了所有CHB患者中不同肝细胞类型对这些ISGs的表达贡献。这些ISGs的B细胞、肝细胞和T细胞的β值如图6A左侧所示。
为了确定ISG表达是否对病毒控制和肝细胞损伤有关联,我们进行贝叶斯线性模型分析了ALT水平与ISGs表达的相互关系。结果显示,ISG20、IFI16、TAP2、GBP1、PSMB9、NLRC5和STAT1等ISGs与T淋巴细胞和巨噬细胞与ALT及AST水平呈强正相关。相反,B淋巴细胞和肝细胞表达的大多数ISGs(如IFI44、ISG15、IFI44L、IFI6、MX1和RSAD2)与ALT及AST水平无关(图6A,B)。而HBV DNA水平与大多数ISGs之间没有显著相关性。
我们在不同临床阶段比较了这些ISGs的β值。图6B,根据其与ALT水平的平均斜率(底部),ISGs在x轴上从大到小依次排列。进一步检查不同临床阶段细胞特异性表达差异,结果显示在免疫活动期(HBeAg+ 肝炎、HBeAg- 肝炎)期间,肝细胞ISG表达最低(图6B)。在这一阶段,ISGs主要由非炎性巨噬细胞和T淋巴细胞表达,少部分由B淋巴细胞表达。此外,T淋巴细胞表达的ISGs倾向于与ALT水平正相关,而B淋巴细胞表达的ISGs与ALT水平不相关(图6B)。图6C和D显示了HBeAg阳性感染期和肝炎期间这些ISGs在不同细胞类型中的β值变化。与ALT水平相关的排序靠前的ISGs表达,包括APOBEC3G、BTN3A3、CTSS和IFI16,主要由T细胞和nMac细胞表达。在HBeAg阳性感染期相比肝炎期间,肝细胞中高表达的ISGs,如IFI44、ISG15、IFI44L、MX1和OAS3,与ALT水平无关(图6C、D)。
总体而言,我们推测由肝细胞和B细胞表达的ISGs,如IFI44、ISG15、MX1和OAS3等不引起炎症反应,可发挥抗炎的作用。相反,ISGs如ISG20、IFI16、APOBEC3G等在T细胞和巨噬细胞中表达,参与肝脏炎症,损伤肝细胞,与血清ALT水平呈正相关(图6E)。

图6. 在CHB肝炎中细胞类型特异性干扰素刺激基因的不同作用
(A) 在CHB四个阶段中79个ISGs的表达情况(热图)。左侧:肝细胞(Hep1),T细胞和B细胞的β值。右侧:每个基因与ALT的线性模型的斜率概率分布(95%置信区间),红色点表示最大及最小β值均大于0。(B) 顶部:不同细胞类型中79个ISGs在慢性肝炎(左)、HBeAg阳性慢性感染(中)和HBeAg阴性慢性感染(右)中的PSEA系数(β值)。底部:慢性肝炎患者中79个ISGs上调,并按与ALT的平均斜率排序;斜率分布的95%可信区间。(C) 相对于HBeAg阳性感染组,慢性肝炎组(HBeAg阳性和阴性)中ISGs的PSEA系数(β)变化。(D) HBeAg阳性感染组与HBeAg阴性感染组中ISGs的PSEA系数(β)变化。(E) 假设机制是,诸如IFI44、ISG15、MX1和OAS3等在HBeAg阳性感染阶段主要由肝细胞表达,在HBeAg阴性感染和肝炎阶段主要由B细胞表达的ISGs不引起肝炎。相反,其他ISGs,包括ISG20、IFI16、APOBEC3G、CXCL10和CXCL11,主要在T细胞和nMacs中表达,参与引起CHB肝损伤。图片由Biorender(https://biorender.com/)创建。
慢性乙型肝炎(CHB)肝脏中不同细胞类型特异性干扰素刺激基因的病理验证
我们对17例CHB患者的肝活检标本进行了IFI44与IFI16、ISG20与ISG15的双重免疫组化染色。在CHB感染组中,IFI44和ISG15的表达较高,而在CHB肝炎组中,IFI16和ISG20的表达较高。此外,IFI16和ISG20主要在淋巴细胞浸润的炎症部位表达。相反,IFI44在炎症部位的表达较少,但在广泛分布于肝细胞区域(图7A-D)。此外,我们进行了多重免疫荧光染色,将ISGs与乙型肝炎病毒核心抗原(HBcAg)共染。IFI44与HBcAg高度共定位。为了阐明HBcAg在CHB肝脏切片中的空间分布差异,我们使用AP-time软件确定了HBcAg阳性细胞的分布。该软件评估了HBcAg阳性细胞与ISGs(IFI44、IFI16、ISG20和ISG15)阳性细胞在不同HBcAg阳性区域内的空间分布差异(距离 < 100 µm)(图7H)。此外,HBcAg分布热图进一步标记了HBcAg阳性的区域(图7G)。
结果显示,IFI44在c抗原阳性区域高表达及共定位,提示HBV在不同临床状态下刺激特异的干扰素刺激基因表达。

图7. 慢性乙型肝炎(CHB)肝脏中不同细胞类型特异性干扰素刺激基因的病理验证
(A) 对HBeAg阳性肝炎患者和HBeAg阳性感染患者的肝脏切片进行IFI44(红色)和IFI16(绿色)的双重免疫组化染色。(B) 对HBeAg阳性肝炎患者和HBeAg阳性感染患者的肝脏切片进行ISG15(红色)和ISG20(绿色)的双重免疫组化染色。(C) 左图:基于多重免疫荧光(mIF),展示肝炎切片和炎症部位IFI44和IFI16表达水平的热图。红色表示较高的表达,蓝色表示较低的表达。右图:展示CHB感染、CHB肝炎和CHB肝炎切片中渗透部位的IFI44和IFI16阳性细胞的柱状图。(D) 左图:基于mIF,展示肝炎切片和炎症部位ISG15和ISG20表达水平的热图。红色表示较高的表达,蓝色表示较低的表达。右图:展示CHB感染、CHB肝炎和CHB肝炎切片中渗透部位的ISG15和ISG20阳性细胞的柱状图。(E) 对HBcAg阳性CHB患者的肝脏切片进行IFI44(红色)和IFI16(紫色)与HBcAg(绿色)的双重免疫组化染色。(F) 对HBcAg阳性CHB患者的肝脏切片进行ISG15(红色)和ISG20(紫色)与HBcAg(绿色)的双重免疫组化染色。(G) 左图:展示HBcAg阳性切片中HBcAg阳性细胞与IFI44、IFI16、ISG20和ISG15的细胞间关联的互动图。右图:直方图展示在100 µm半径内,IFI44、IFI16、ISG20和ISG15阳性细胞与HBcAg阳性细胞的平均距离。每个条表示不同距离下阳性细胞的百分比。(H) 左图:基于mIF,展示HBcAg阳性细胞分布的热图。热图用于展示HBcAg阳性细胞的分布,以识别HBcAg阳性区域。红色表示HBcAg阳性细胞的较高细胞计数,蓝色表示较低的细胞计数。右图:直方图展示双阳性细胞的平均百分比。直方图展示在整个切片和HBcAg阳性区域内,HBcAg+ISG15+、HBcAg+ISG20+、HBcAg+IFI44+和HBcAg+IFI16+双阳性细胞的平均百分比。x轴表示不同组合的双阳性细胞,y轴表示这些双阳性细胞的平均百分比。
方 法
患者队列和转录组数据集
我们先前的研究中,从上海华山医院、瑞金医院和上海市公共卫生中心招募了82名慢性乙型肝炎(CHB)患者纳入研究。这些患者的临床特征和伦理指南已在之前的研究论文中描述。我们使用R包pheatmap进行无监督层次聚类分析患者的临床参数(血清ALT、HBV DNA、HBeAg、坏死性炎症评分(0-4))并进行显示。为了进行临床验证,我们使用了来自CHB患者的肝脏切片,这些样本是存储在病理科的患者检查剩余样本,已获得深圳市第三人民医院研究伦理委员会批准并按照1975年《赫尔辛基宣言》的伦理指南执行。CHB患者被分成两组,即肝炎组(HBeAg阳性,G ≥ 2)和感染组(HBeAg阳性,G < 2),进一步对17份肝脏切片进行了多重免疫染色。
差异基因表达分析
使用Limma包评估了差异的整体基因表达。为了提高统计分析的可靠性,我们遵循了微阵列质量控制(MAQC)项目的建议,并要求显著的差异基因在两个比较组之间具有校正后p值 < 0.01和倍数变化> 2。使用R包factoextra对82名患者的差异基因的前2个主成分进行了可视化。为了将差异基因转化为特定的免疫功能和细胞类型相关的注释,我们的分析还使用了外周血液转录组模块(BTMs)分析。每个BTM包含一组具有相似生物功能的基因。
定义每种肝内细胞类型的标记基因
我们合并了来自人类肝脏的三个10×基因组学单细胞RNA测序数据集(GSE115469、GSE186343、GSE124395),并使用ST转换方法进行了归一化处理,包括6例肝炎期CHB肝和12例健康供体肝。使用UMAP方法映射和可视化了13种细胞类型。从所有细胞簇的差异基因(p值 < 0.01)中筛选候选标记基因。然后,使用层次聚类方法(pheatmap包)探索了仅在单个簇中表达的基因及其相关的细胞类型,这些基因进一步被选为下一步分析的细胞标记基因。
基于标记基因的表达分解成细胞类型的贝叶斯多元线性模型
先前的研究表明,线性模型可以捕捉大多数基因的行为。具体而言,特定类型细胞群体中的RNA表达可以描述为线性依赖于该群体的大小。由于在人类临床或尸检样本中准确测量不同细胞类型的群体大小通常是不可能的,Alexandre等人提供了一种多线性方程,描述了通过标记基因的表达水平(PSEA)来计算相对细胞群体大小的模型:

(y,组织样本中某基因的表达;y'p组织样本中细胞类型 p 的细胞标志基因的表达;x'p,假设组织样本仅包含细胞类型 p 的细胞标志基因的表达;x',由细胞类型 p 贡献的特定基因的表达;βp,特定基因相对于细胞类型 p 的细胞标志基因标准化后的表达;𝑎,背景;𝑃,组织样本中细胞类型的数量)
贝叶斯线性回归连接基因表达与临床参数
为了比较来自PSEA获得的给定基因的细胞类型特异性表达在不同临床阶段中的差异,构建了贝叶斯多元线性回归模型。同时还构建了贝叶斯线性回归模型用于探索了临床血清参数(ALT、AST和HBV DNA)与BTMs、不同细胞类型的标记基因和免疫相关基因(如免疫抑制剂、TLR、ISGs、趋化因子和受体、细胞因子和干扰素)的表达之间的相互关系。我们使用Stan通过R包rstanarm的glm函数实现了贝叶斯线性回归。
多重免疫荧光染色及多重免疫染色切片分析
对四个肝脏切片进行了多重免疫染色。依次进行了主要抗体的优化浓度,并与相应的二级抗体结合,这些抗体由5种不同的荧光IHC试剂盒检测。
利用AP-TIME图像分析软件(3D Medicines, Inc.),导入图像并进行了严格的检查。该软件便于量化细胞密度,表示为每平方毫米阳性染色细胞的数量(cells/mm^2)。此外,软件计算了阳性染色细胞占DAPI染色或有核细胞总数的百分比,包括整个肝脏切片和特定感兴趣区域的细胞百分比。根据需要,该分析考虑了单通道或多通道的荧光。
为了阐明免疫细胞在炎症部位内的空间分布偏好,软件测量了在不同细胞病变区域内感兴趣细胞之间的空间接近度(< 100 µm的距离内)。通过建立特定标记物的细胞阳性阈值,软件实现了精确的细胞分割。对于每个患者样本,提取了包括每个细胞特征和空间坐标在内的综合数据。为了增强可视化效果,该软件生成了基于收集数据的感兴趣区域复合图像。后续数据分析使用GraphPad Prism软件(版本8,Inc.; San Diego, CA, USA)进行。使用Mann-Whitney检验确定两组之间的统计显著性。结果的调整p值 < 0.05被视为统计显著。符号*、**、***、****和分别表示p < 0.05,p < 0.01,p < 0.001和p < 0.0001。
讨 论
理解宿主免疫反应、肝损伤与病毒控制之间的关系对慢性乙型肝炎(CHB)的研究至关重要。宿主免疫反应对于控制病毒复制至关重要,但在持续性CHB中,免疫反应未能有效控制HBV,导致肝损伤。因此,直接干预肝脏炎症的方法对于实现CHB的功能性治愈至关重要。
免疫细胞在肝组织中的增加浸润可能导致活检组织中免疫细胞比例显著增加。肝脏炎症组织中基因表达上调,可能是由于增加的免疫细胞数量,导致与免疫细胞相关基因的平均表达水平上升。分析组织基因差异表达的另一个局限性是无法在细胞水平阐明参与炎症的免疫途径,以及不同细胞类型中干扰素刺激基因(ISGs)的特定表达模式。单细胞测序技术提供了解决上述限制的一种有效的方法。2023年,张等人从23名CHB患者的肝组织CD45+免疫细胞中进行了单细胞测序,跨越不同临床阶段。他们的研究突显了免疫耗竭的CD8+ T细胞与FCGR3A+巨噬细胞在炎症形成中的重要角色。然而,免疫活跃期中患者样本量较少(仅5例),这给构建与临床数据相关的相关性统计模型带来了挑战。肝脏中每种细胞类型的变化可以通过细胞类型特异性标记基因进行定量描述,基于最近的单细胞分析研究,在此基础上,肝损伤的临床参数可以与免疫细胞标记物相关联。因此,我们构建了一个贝叶斯线性模型,将基因表达与临床参数联系起来,并使用来自单细胞测序数据集的标记基因,通过PSEA模型将基因表达拆解到不同的临床阶段的特定细胞类型。
我们整合了肝脏组织转录组数据、单细胞测序数据和临床数据,从多组学的视角分析了引发慢性乙型肝炎(CHB)肝脏炎症的因素。我们的分析结果与早先的研究建议一致,即增加免疫细胞向肝脏的招募导致肝损伤,但不能有效抑制HBV复制,持续性HBV感染导致免疫细胞耗竭。来自CD68+巨噬细胞的CCL20和CXCL8招募耐受型T细胞(CTLA4、EOMES、LGALS3、TIGIT)到肝脏,从而诱导肝细胞非特异性损伤。在CHB肝炎阶段,肝细胞内的先天免疫被抑制,阻碍了ISGs发挥抗病毒效果,免疫细胞被招募并在肝脏中活化,是表达特定ISGs的主导细胞群体,在浸润的免疫细胞中激活先天免疫途径进一步加剧了炎症形成。因此,肝内细胞群体异质性很强,随时间动态变化,并在肝组织中显示不均匀的空间分布。这也解释了不同ISGs的表达及其与ALT值的相关性。
在本研究中,我们的分析未能证实免疫浸润与HBV DNA水平之间存在显著相关性。这一发现与之前的假说一致,即并非招募到肝脏的免疫细胞都对HBV的控制有贡献。在特异性T细胞反应不足以控制HBV时,可能会招募到像巨噬细胞和非特异性T细胞这样的免疫细胞。非特异性免疫细胞不能有效抑制HBV的复制,而是通过产生细胞毒性因子导致肝细胞损伤。因此,可以理解在CHB中免疫细胞标记物与HBV DNA水平之间未观察到直接相关性。我们得出结论,我们的肝内转录组分析为由耐受T细胞和巨噬细胞驱动的肝损伤提供了证据,但未能确认肝内T细胞反应在CHB中对控制HBV复制的效果。T细胞中激活的ISGs在肝炎中也起到诱导或者参与作用。B细胞和肝细胞中激活的ISGs可能对肝脏的损伤有保护作用。
代码和数据可用性
本研究的支持数据可从NCBI基因表达综合数据库(NCBI Gene Expression Omnibus)获取,网址:https://www.ncbi.nlm.nih.gov/geo/,GEO系列登录号为:GSE65359、GSE115469、GSE186343、GSE124395 以及补充数据。使用的脚本保存在GitHub上,网址为:https://github.com/JunWang-Lab/iMeta。补充材料(方法、图表、图形摘要、幻灯片、视频、中文翻译版及更新材料)可在在线DOI或iMeta Science网站上找到,网址为:http://www.imeta.science/。
引文格式:
Jun Wang, Qian Li, Yuanwang Qiu, Simo Kitanovski, Cheng Wang, Chenxia Zhang, Fahong Li, et al. 2024. "Cell-type specific expression analysis of liver transcriptomics with clinical parameters to decipher the cause of intrahepatic inflammation in chronic hepatitis B." iMeta 3: e221. https://doi.org/10.1002/imt2.221
作者简介

王俊(第一作者)
● 德国 University of Duisburg-Essen获得Ph.D.学位,深圳国家感染性疾病临床医学研究中心(深圳三院)博士后,江南大学硕士生导师,无锡市第一、第二届双百拔尖人才。
● 方向:生物信息学(感染与免疫)。第一或通讯作者发表SCI论文20多篇,如iMeta、Emerging Microbes & Infections、Cell Death & Disease、Clinical Translational Medicine等。主持国家或省部级以上课题三项。获得省科技进步三等奖,省预防医学科技二等奖,省新技术引进二等奖各一项。

李倩(第一作者)
● 深圳国家感染性疾病临床医学研究中心特聘研究员,德国Duisburg-Essen大学获Ph.D.学位。
● 方向:感染免疫与炎症致病机制研究。在iMeta、Cellular & Molecular Immunology、Cell Death & Disease、Emerging Microbes and Infections、Clinical Translational and Medicine、Journal of Immunology等SCI上发表论文多篇,主持国家级课题2项,十四五课题骨干,主持省市级课题3项。

卢洪洲(通讯作者)
● 国家教育部长江学者、美国微生物学院院士、深圳国家感染性疾病临床医学研究中心主任。
● 发表论文1500余篇,包括Nature、NEJM、Cell Research、Lancet等高影响力期刊,文章总引用31447次,H指数为60;主编专业参考书20部;主持国家“十一五”到“十四五”传染病重大专项、国家自然科学基金、美国 NIH 项目等 60多项科研课题;获国家科学技术特等奖、法国国家医学科学院“夏▪杜奖”;以第一完成人获得10多项上海科技成果一等奖。

陆蒙吉 (Lu, Mengji)(通讯作者)
● 1990年在德国马普所获得微生物专业博士学位,德国ESSEN大学医学院病毒研究所教授,博士研究生导师。
● 擅长病毒性肝炎动物模型,肝炎病毒分子生物学及基因免疫治疗等方向,在土拨鼠肝炎模型、抗乙肝病毒天然与特异性免疫应答作用、新的免疫调节治疗策略的探索等方面开展了创新性工作。持德国联邦科技部及自然科学基金会项目10余项, 合作获得国家自然科学基金项目多项,863课题、中德跨学科重大合作项目、国家重大传染病科技专项课题,共同发表300余篇论文,合作培养博士生60余名。

Daniel Hoffmann(通讯作者)
● 1996年博士毕业于柏林弗雷大学化学系,现任德国杜伊斯堡-埃森大学终身教授,生物学院及医学生物技术中心(ZMB),生物信息学与计算生物物理学方向负责人。
● 主要研究领域方向是生物信息学、蛋白质结构、贝叶斯建模及人工智能。发表论文150余篇,引用超4900次,H指数为36。主要开发生物软件包为:HAMdetector: 一种贝叶斯回归模型,用于整合信息检测HLA相关突变;IgGeneUsage: 免疫谱系中差异基因的使用情况。

张继明(通讯作者)
● 现任复旦大学附属华山医院感染科副主任、国家传染病医学中心副主任、上海市肝病研究所病毒性肝炎研究室主任, 中华医学会医学病毒分会常委等。
● 长期从事感染/传染病学临床、研究和防控工作。承担多项科技部重大专项子课题、国家自然科学基金项目及NIH国际合作课题。发表国际高水平SCI论文100多篇,包括GUT、Hepatology、Journal of Hepatology、PLoS Pathog、 Nature Communications、Clinical Infectious Diseases等。
更多推荐
(▼ 点击跳转)
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百千华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表所有领域高影响力的研究、方法和综述,重点关注微生物组、生物信息、大数据和多组学等。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!发行后相继被Google Scholar、ESCI、PubMed、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.7,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,同学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,是定位IF>10的高水平综合期刊,欢迎投稿!
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science








































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








