人类参考基因组38版与19版有什么区别?
人类参考基因组38版与19版有什么区别?_数据库_Genome_at
GRC
参考基因组联盟(Genome Reference Consortium, GRC)是一个国际学术和研究机构的集合体(如下),于2007年创立,具有基因组制图、测序和信息学方面的专业知识。
- The Wellcome Sanger Institute【维康桑格研究所,以前称为桑格中心和威康信托基金桑格研究所,是一家非盈利的英国基因组学和遗传学研究机构,主要由惠康基金会资助。】
- The McDonnell Genome Institute at Washington University【MGI,麦克唐纳基因组研究所,位于密苏里州圣路易斯华盛顿大学,隶属于华盛顿大学医学院和 Alvin J. Siteman 癌症中心,美国国立卫生研究院资助的三个大型测序中心之一。】
- The European Bioinformatics Institute【EBI,欧洲生物信息学研究所,是一个政府间国际组织的学术机构,致力于以信息学手段解答生命科学问题。该所建立于1994年,位于英国剑桥南部的维康信托基因园,位于剑桥附近欣克斯顿的惠康基因组校区,作为欧洲分子生物学实验室(European Molecular Biology Laboratory, EMBL)的一部分,专注于生物信息学的研究和服务,该所管理维护着世界最全面的分子生物数据库,其中很多是生物学家熟悉的数据库,例如核酸序列数据库(ENA)、Ensembl (housing whole genome sequence data,几个知名的基因组浏览器之一,用于检索基因组学信息)、UniProt(protein sequence and annotation database,蛋白质数据库)、InterPro(蛋白质家族/域/蛋白指纹等)、Protein Data Bank (PDB, protein and nucleic acid tertiary structure database,蛋白分子结构数据库)。】
- The National Center for Biotechnology Information【NCBI,位于美国马里兰州的贝塞斯达,建于1988年,是美国国家医学图书馆的一部分(该图书馆又是美国国家卫生研究所的一部分),NCBI设置了与生物技术和生物医学相关的一系列数据库,是生物信息学工具和服务的重要资源。主要数据库有DNA序列数据库GenBank、生物医学文献数据库PubMed、表观基因组数据库。所有这些数据库都可以通过Entrez搜索引擎在线获取。】
- Zebrafish Model Organism Database【在1994年冷泉港斑马鱼遗传和发育会议上,一组斑马鱼研究人员建立了斑马鱼作为模式生物的在线信息数据库Zebrafish Information Network(ZFIN)(https://zfin.org/),这是斑马鱼(Danio rerio)作为模式生物的遗传和基因组的数据库。ZFIN提供了大量专业的斑马鱼遗传和基因组数据,如基因、等位基因、转基因品系、基因表达、基因功能、表型、人类疾病模型、命名和试剂。】
- Rat Genome Database【RGD, 大鼠基因组数据库,于1999年创立,并迅速成为大鼠基因组学、遗传、基因组、表型和疾病相关数据的数据库。目前RGD已扩展至10个物种(包括大鼠、小鼠、人、龙猫、倭黑猩猩、多纹黄鼠、狗、猪、绿猴和裸鼹鼠)。RGD提供了一套可用于查询、分析和可视化这些基因组信息。】
官网:https://www.ncbi.nlm.nih.gov/grc/human
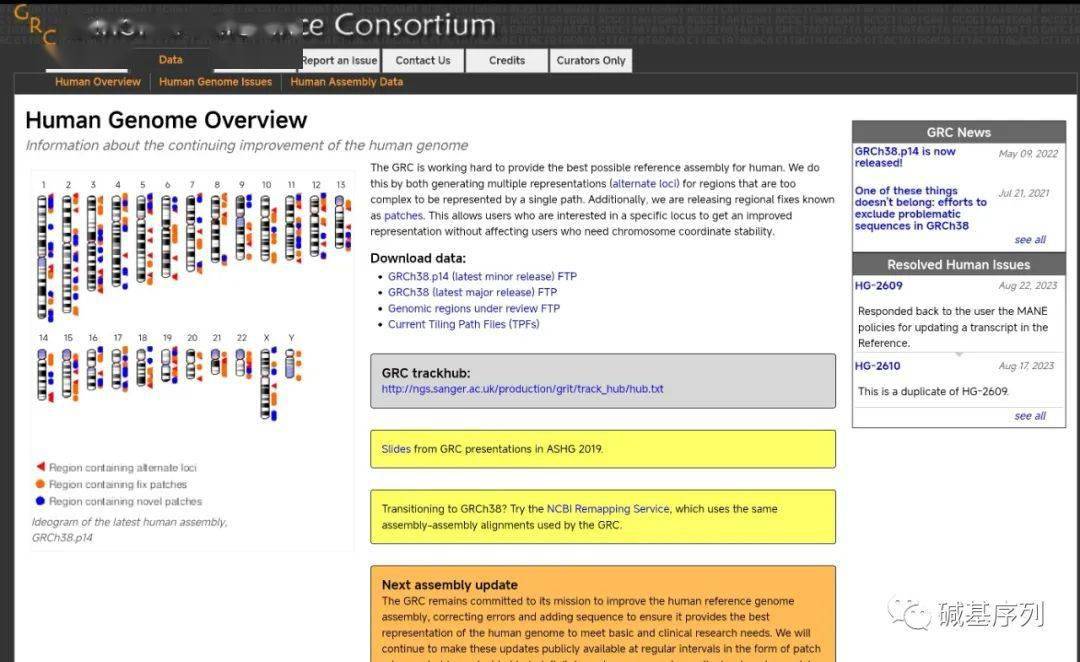
目前最新版本的人类参考基因组官名叫GRCh38 (Genome Reference Consortium Human Build 38),该版本由GRC于2013年12月释放。UCSC基因组浏览器也将该版本收藏,并称之为hg38,这个小名对于大多数人来说是更亲切熟悉的。GRCh38在GenBank中叫GCA_000001405.15,在 RefSeq 中叫GCF_000001405.26,虽然GRC组织建议在所有出版物和工具中使用该编号,但事实是前两种GRCh38和hg38 对生信分析更常见。
在不更改染色体坐标的情况下,向参考基因组添加或替换新序列,这种打补丁的方式,会在基因组版本后加 .p(patch)来命名。例如,目前最新的为2022年5月9号更新的GRCh38.14,第14个补丁,正式版本叫做 Genome Reference Consortium Human Build 38 patch release 14。
GRCh38优势
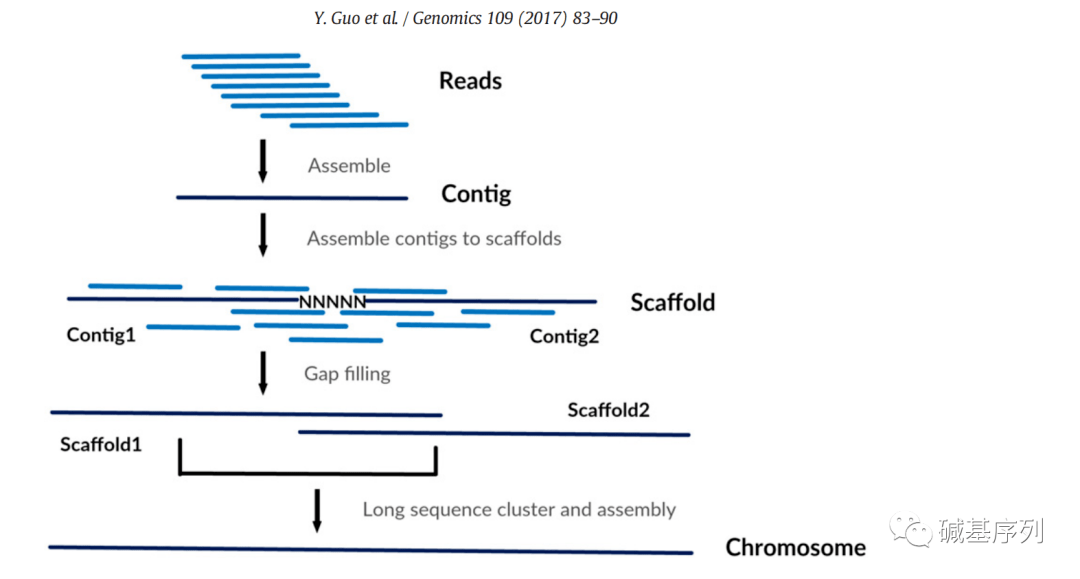
人类参考基因组是所有基于高通量测序的生物医学研究的基本必要条件。参考基因组的组装(genome assembly)是生物信息学领域的核心问题,就是把机器测序产生的读取片段reads经过序列拼接组装,生成基因组的碱基序列。
宏观来说,基因组组装可以分为从头组装(de novo assembly)和映射比对组装(mapping assembly)。从头组装是指不需要依靠任何已知的基因组信息;而映射比对组装就是需要把测序序列和参考基因组来比对,找到序列的对应位置再进行组装。
- reads:由于受目前测序水平的限制,基因组测序时需要先将基因组打断成DNA片段,然后再建库测序。reads指的是测序仪单次测序所得到的碱基序列,也就是一连串ATCGATCG之类的序列,不同的测序仪器,reads长度不一样。对整个基因组进行测序,就会产生成百上千万的reads。通常一代和三代的reads读长可达千个bp,二代测序的reads相对较短,平均是几十到几百bp。
- contig:中文叫做重叠群,就是通过不同reads之间的overlap交叠区,拼接成的序列,是基因组序列的连续长度,其中碱基的顺序具备高置信水平。
- scaffold:这是比contig还要长的序列,获得contig之后还需要构建paired-end或者mate-pair库,确定contig的顺序关系和位置关系,从而获得获得一定大小(如3Kb、6Kb、10Kb、20Kb)的两端序列,最后contig按照一定顺序和方向组成scaffold,其中形成scaffold过程中还需要填补contig之间的空缺(gap)。
基因测序报告中出现的专业词汇是什么意思?——名词解释
全外显子组测序数据分析流程中出现的专业词汇是什么意思?——名词解释(2)
高通量测序数据质量评价中涉及到专业词汇——名词解释(3)
基于OLC算法(Overlap Layout Consensus,先重叠后扩展)
OLC算法适用于较长reads的一代和三代测序。OLC算法最初成功的用于Sange测序数据的组装。OLC算法的整体步骤可以分为三步:
- Overlap:对所有reads进行两两比对,找到片段间的重叠信息,一般在比对之前会将reads做下索引,减少计算量。这里需要设定最小重叠长度,如果两个read的最小重叠长度低于一定阈值,那么可以认为两段序列顺序性较差。
- Layout:根据得到的重叠信息将存在的重叠片段建立一种组合关系,形成重叠群,即Contig。Contig进一步排列,生成多个较长的scaffold。
- 根据构成Contig的片段的原始质量数据,在重叠群中寻找一条权重最重的序列路径,并获得与路径对应的序列,即consensus序列。
- 通过consensus的多序列比对算法,就可以获得最终的基因组序列。
下面这个图更为形象的解释OLC算法
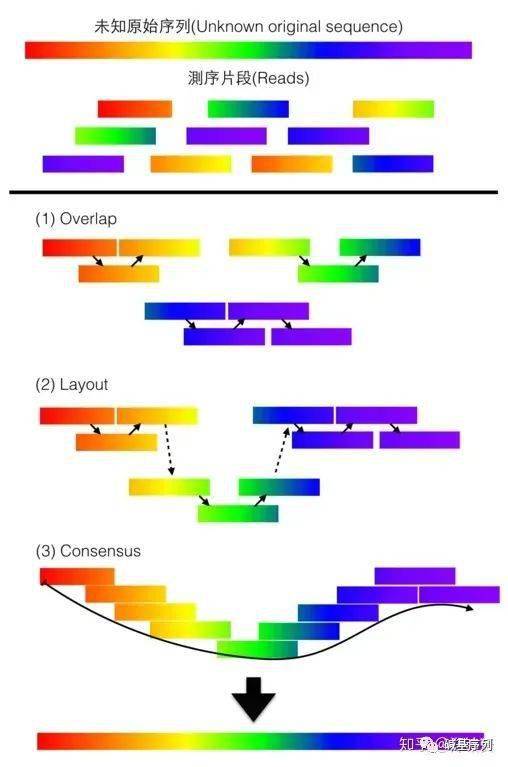
在实践中,构建一个完整和正确的人类参考基因组存在许多挑战。最困难的地方就是重复的DNA区域,如端粒,具有相当复杂的consensus序列。由于测序对GC含量有不同的敏感性,引起基因组的不均一性,导致scaffold之间的gap产生。研究人员一直在积极地应对这些挑战,并逐步改进人类参考基因组。
人类基因组计划于2001年组装了第一个人类参考基因组。
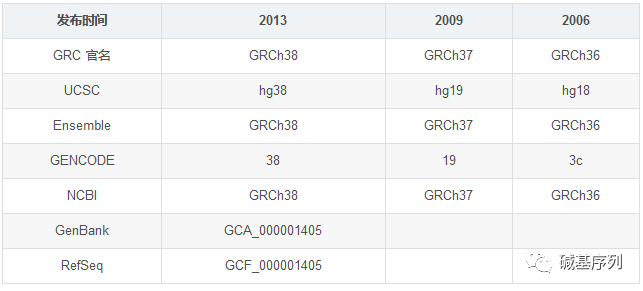
2009年,GRC发布了第19版人类基因组GRCh37,也常被称为hg19。GRCH37被广泛应用于数据分析。
2013年,GRC发布了第20版人类基因组GRCh38。但由于注释工具、数据库的不健全及升级基因组的工作繁杂,时至今日,GRCH37仍被相当程度地使用。

这篇文章主要研究了GRCh38和GRCh37之间的比较,以及GRCh38在基因组分析中的准确性和可靠性。文章通过比较两个版本的基因组,发现GRCh38在基因组分析中具有更高的准确性和可靠性,特别是在检测结构变异方面。文章还指出,GRCh38在基因组分析中的准确性和可靠性的提高,不仅得益于基因组组装的改进,还得益于参考基因组中的多样性。
GRCh38是目前(以文献发表时间算)测序最准确的人类基因组。测序使用的金标准是Sanger测序,可以产生长达1000个bp的reads,其准确性是高通量短读长测序的10倍。与GRCh37相比,GRCh38改变了8000个bp,纠正了几个错误组装的区域,填补了空白,增加了着丝粒序列,在178个区域组装了261条alternate loci,丰富了基因组的多样性。
为了比较GRCh38和GRCh37对基因组学分析的影响,作者使用这两种人类参考基因组分析了一组外显子组序列(N=30)。从染色体基本统计、比对、单核苷酸变量(single nucleotide variables, SNV)、小插入和缺失(small insertion and deletion, INDEL)、拷贝数变异(copy number variation, CNV)、结构变异等多个角度对GRCh38和GRCh37进行比较。
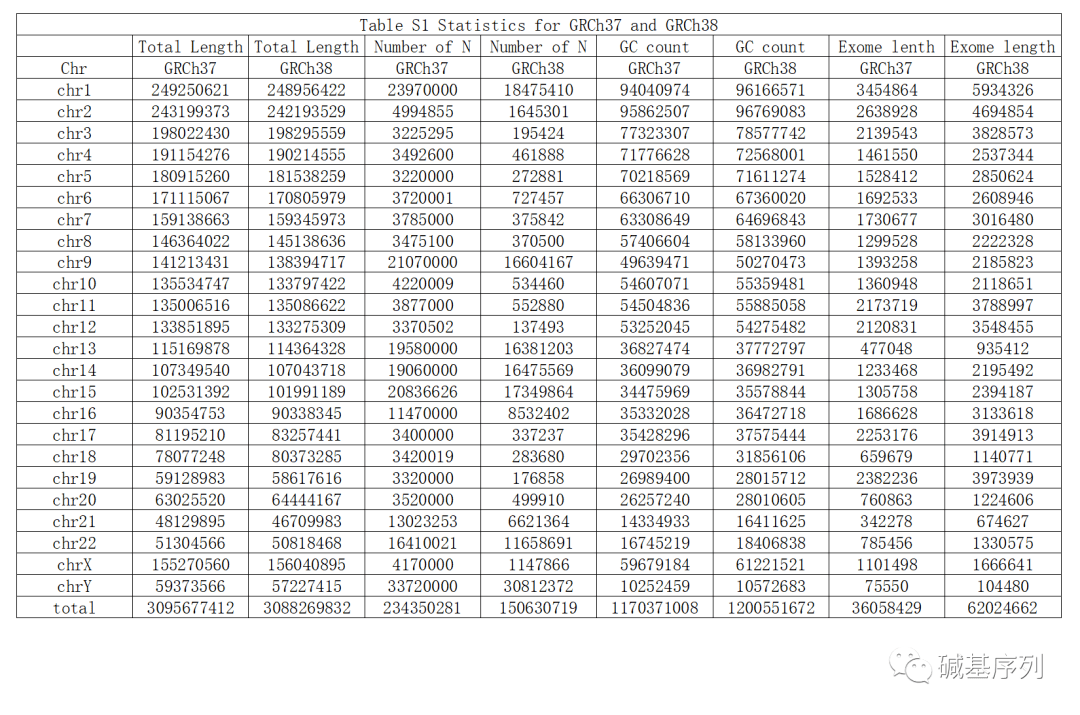
GRCh37总共有3,095 677,412个核苷酸,GRCh38总共有3,088,269,832个核苷酸,减少了7,407,580个核苷酸。不算线粒体DNA,最常用的线粒体基因组是1999年剑桥发布的rCRS,两者线粒体基因组是一样的。在检测的24条染色体中,16条GRCh38的核苷酸计数减少,8条增加。
字母“N”在参考基因组(FASTA文件)中表示序列间隙或未注释区域。GRCh37的Ns总数为234,350,281,GRCh38的Ns总数为150,630,719,减少了83,719,562 Ns。24条染色体均出现Ns数目减少。
GC含量是基因组中G和C核苷酸的百分比。已有研究表明,GC含量会影响Illumina测序的效率,并影响后续的分析,如CNV的检测,而CNV的检测严重依赖于深度覆盖。GC含量百分比因人类基因组的区域而异。GC位点总数从GRCh37的1,170,371,008增加到GRCh38的1,200,551,672,增加了30,180,664个核苷酸。当计算GC%时,由于GRCh38的Ns数目减少,24条染色体中有17条的GC%减少。
外显子组的定义在很大程度上依赖于基因组注释。分析了从Ensembl (GRCh37 v37.75, GRCh38 v38.82)下载的最新基因特征格式(Gene Feature Format, GTF)文件的外显子组大小。外显子组大小从GRCh37的75,231,228增加到GRCh38的95,505,476,增加了20,274,248个核苷酸,增加了26.90%。所有染色体的外显子组大小均增加。GRCh37为2.43%,CRCh38为3.09%。外显子组大小的增加可归因于以下3个原因:GRCh38的总外显子数量从327,058增加到457,748;每个基因的外显子中位数也从13增加到19;每个外显子的核苷酸中位数几乎从140增加到146。这些综合因素解释了GRCh38外显子组百分比增加的原因。
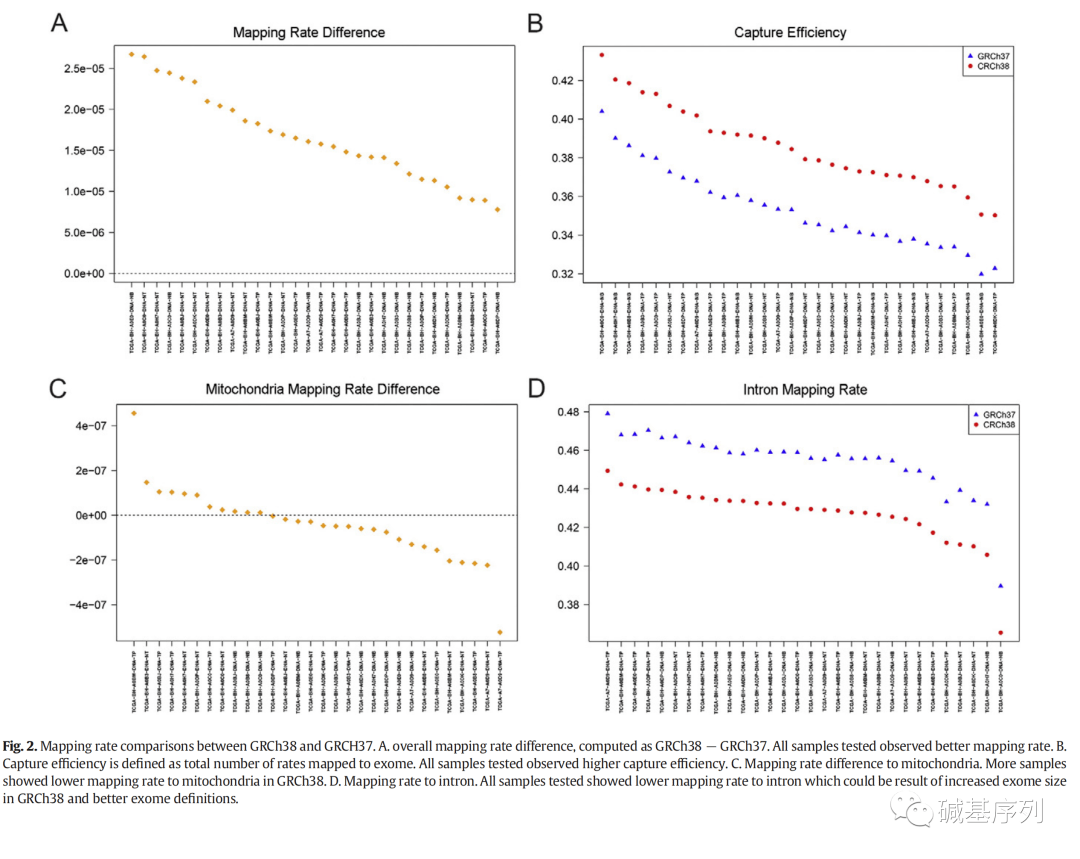
比对(alignment)是高通量测序数据分析中非常重要的一步。总有部分reads无法比对至参考基因组,有论文指出改进基因组可以提高比对率。从图2看出,30个WES样本的比对率都得到了提高,提高均值为0.0017%。外显子区域的比对率明显提高,约为3.22%,主要原因是外显子区域扩大,相应地内含子的比对率降低了2.70%。线粒体基因组的比对率略有下降,平均下降0.0005%,30个样本中有19个样本比对到线粒体DNA的reads数略有减少。由于mtDNA参考在GRCh37和38之间保持不变,这种降低可能是由于线粒体基因组的核拷贝数(nuclear copies of the mitochondrial genomes, nuMTS)的改变。
总体而言,使用GRCh38发现的SNVs较少。使用GRCh37鉴定出4,656,461个SNVs, GRCh38鉴定出4,617,859个SNVs。这表明,整体上改进了人类参考基因组,GRCh38产生的假阳性SNVs更少。
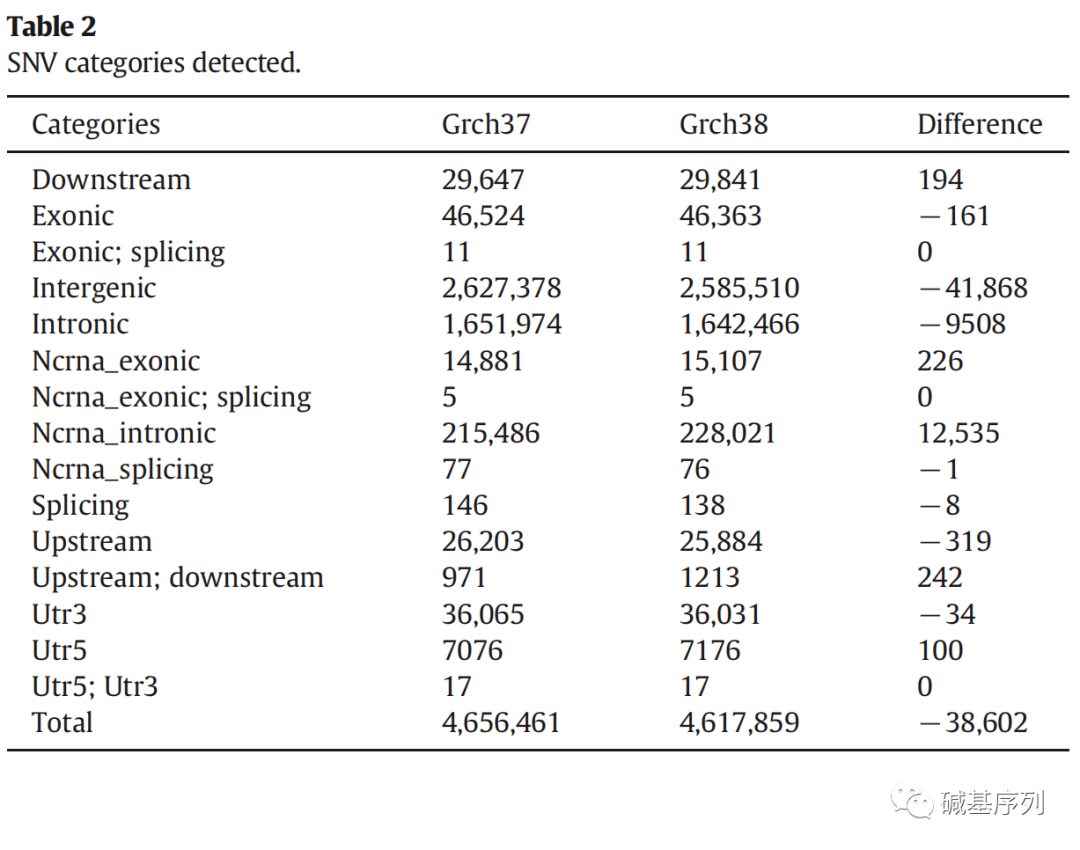
非同义变异(nonsynonymou,stopgain, stopploss)是我们关注的重点,SNVs总数变少,但GRCH38比GRCH37多了22,796个非同义变异,主要原因是外显子区域增加。使用LiftOver转化参考基因基因组坐标后显示,两种参考基因组中93%SNVs是一致的,且质量值和覆盖度并无差异。
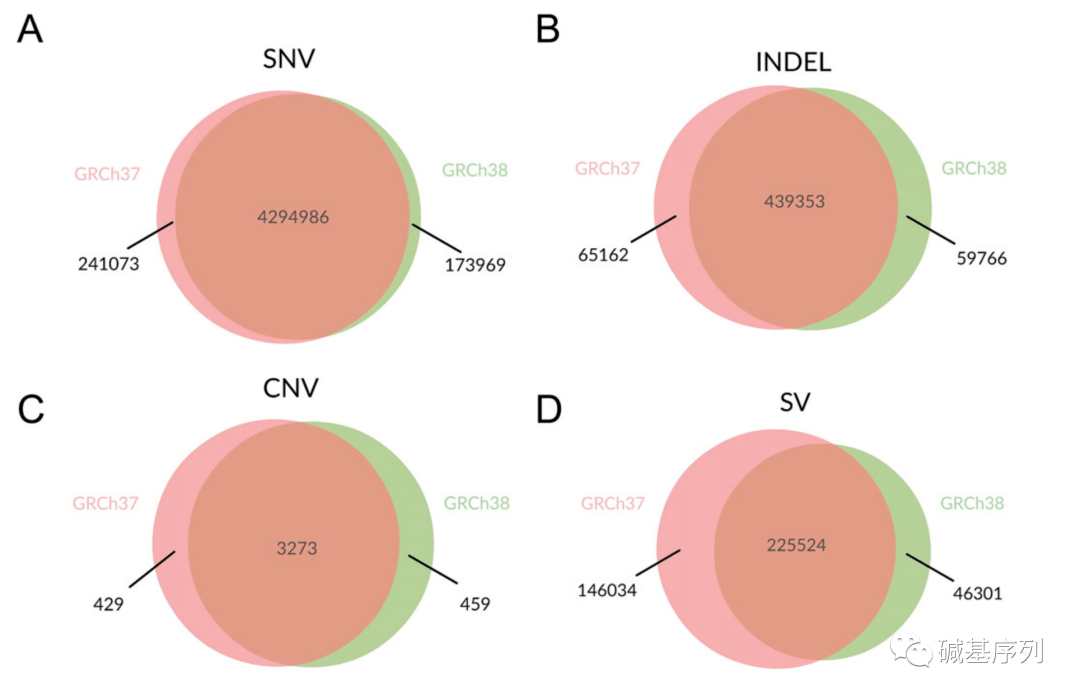
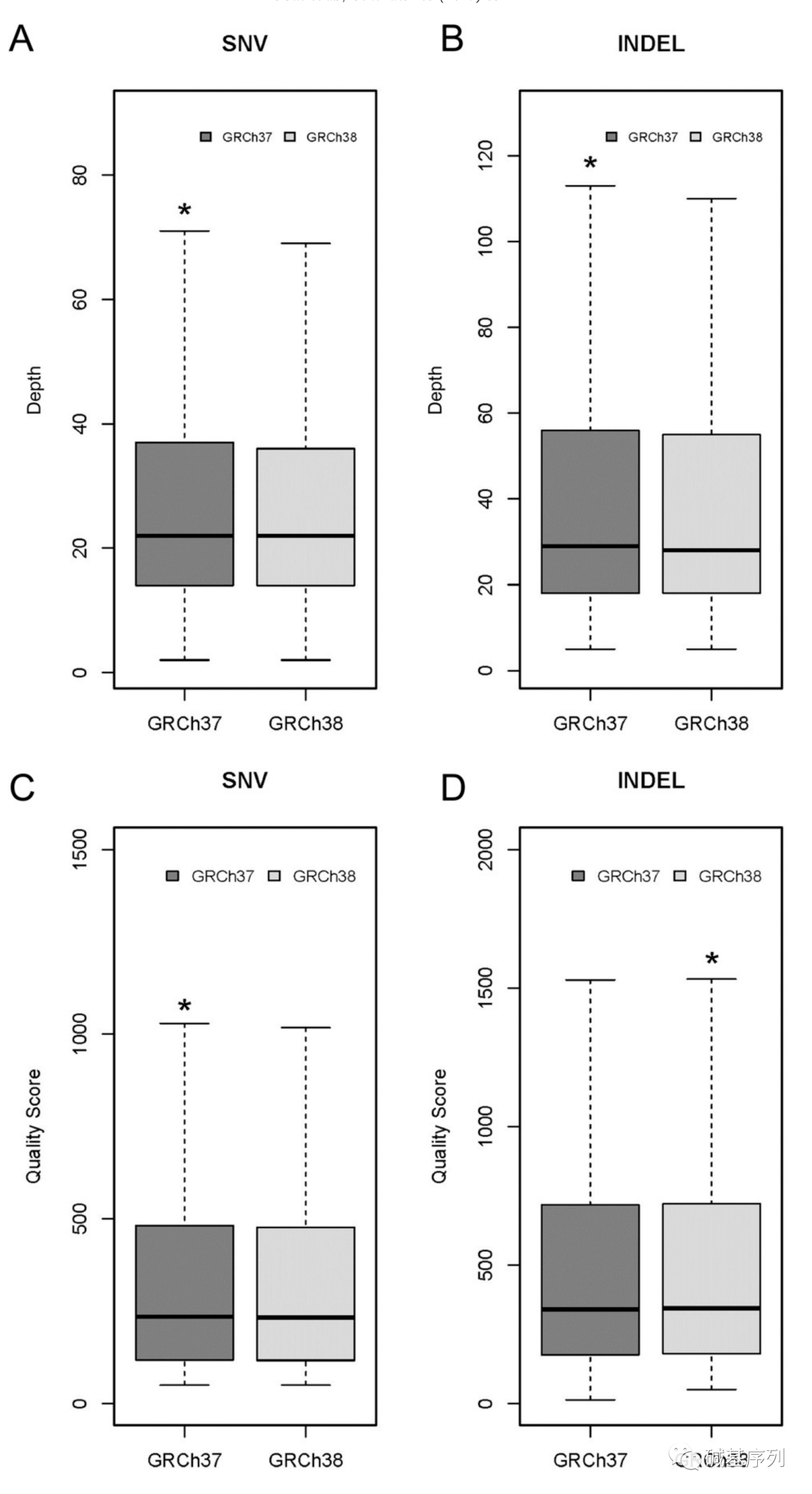
还确定了GRCh37的241,073个独特SNP和GRCh38的173,969个独特SNP。对于INDELs, GRCh37鉴定了504,515个,GRCh38鉴定了499,119个,两个参考基因组比对中88.0%的INDELs是一致的。在SNVs和INDELs的质量值和深度覆盖,GRCh37和GRCh38之间没有实质性的差异。
GRCh37鉴定出3702个CNVs, GRCh38鉴定出3732个CNVs, 88.4%的CNVs在两个参考基因组之间一致。这两个参考基因组都发现了重复片段明显>缺失片段。使用GRCh37,发现了135个缺失和3567个重复。使用GRCh38,发现了131个缺失和3601个重复。这些结果与之前的发现一致,即高通量测序容易识别出更多的重复CNVs,而不是缺失CNV。
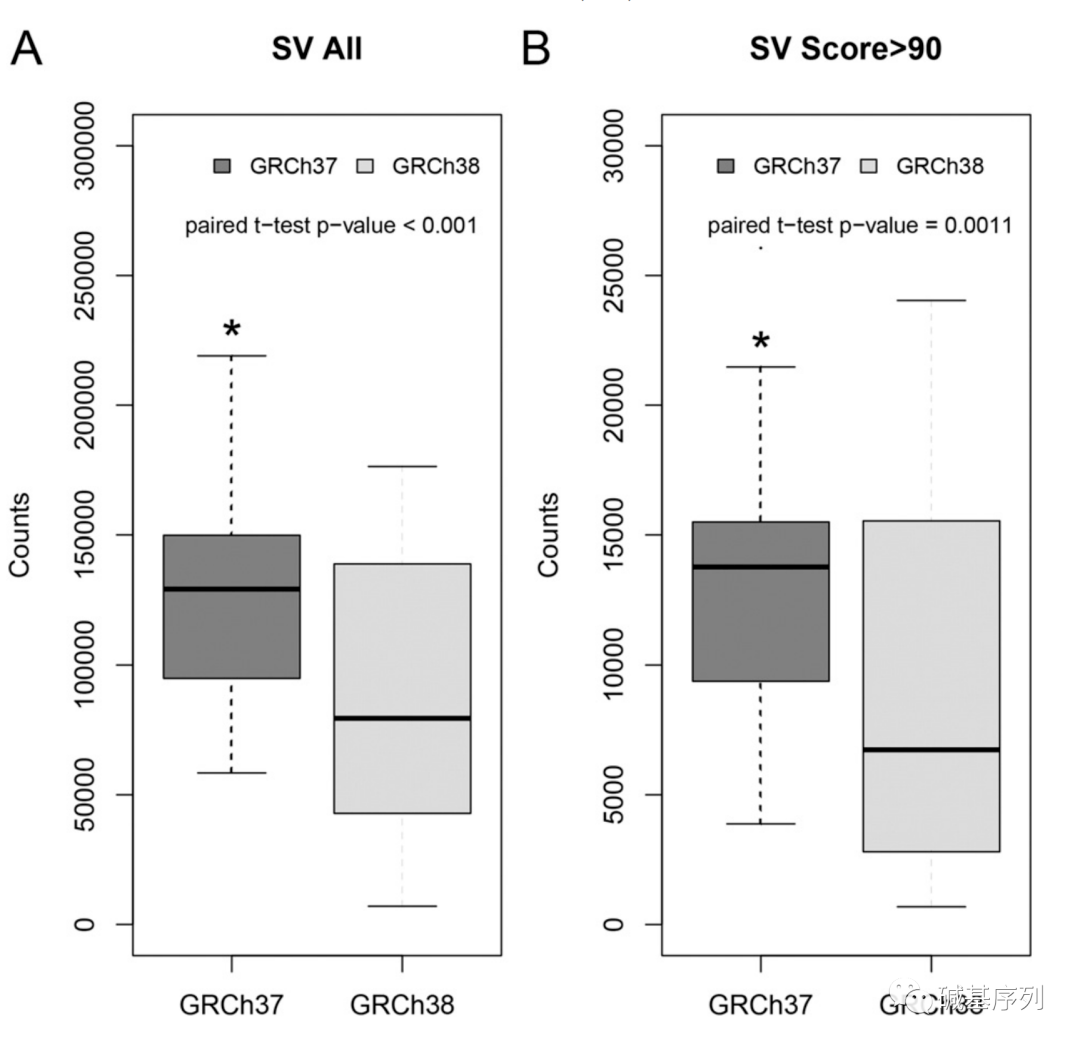
使用GRCh37,发现了371,558个结构变异,而使用GRCh38,发现数量少得多(少26.8%)的271,825个结构变异。83.0%的结构变异能同时在两个基因组中检测到。结构变异检测难度大,且有较高的假阳性率。与GRCh37相比,使用GRCh38可以降低了假阳性率。虽然作者没有使用金标准来计算真阳性率和真阴性率,但变异数量减少预示着假阳性率降低。
GRCh38对着丝粒区域序列的改进。从UCSC基因组浏览器中获得了GRCh38中的着丝粒坐标。GRCh37的着丝粒坐标是不完整的。提取所有比对到GRCh38着丝粒区域的reads,然后将这些着丝粒序列比对回GRCh37,发现41.5% ~ 53.3%的reads能被成功比对到GRCh37。然而,绘制的位置不一定是着丝粒区。
重组人类基因组是一项费时又费力的任务,截止2018年,人类基因组已经发布了20个版本。GRCh38的一个重要技术进步是葡萄胎的应用。葡萄胎没有从卵子中获得染色体,精子的染色体发生了复制,因此没有等位基因变异,可用于获得基因组上高度同源区域的reads。
GRCh38并不是完美的人类基因组,其主要缺陷在着丝粒区域。该区域包括数百万个碱基,序列高度重复。GRCh37着丝粒区域以gap形式存在,GRCh38建立模型推测,虽不准确,但还是向前迈进了一大步。
人类基因组仅代表在基因组位点上的1个等位基因位点。参考等位基因是根据一个小群体的基因组确定的,可能并不是主要等位基因(人群频率>50%)。在某些情况下,检测的目标人种没有参考等位基因存在。目前的检测软件,如GATK,Platypus都允许一个位置存在多种等位基因。
基于GRCh37和GRCh38的WES样本数据分析显示,GRCh38可以得到更准确的分析结果。GRCh38具有更好的比对效果,对后续CNVs、结构变异的检测都具有正面影响。综上所述,GRCh38是人类基因组从GRCh37迈出的一大步,基因组准确度的提升对于高通量测序数据分析具有明显的积极意义。





















 3616
3616

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








