参考文献: Cao L, Coventry B, Goreshnik I, et al. Robust de novo design of protein binding proteins from target structural information alone[J]. bioRxiv, 2021.
解读作者:吴炜坤
一、背景介绍
自从2012年以来,de novo设计靶向结合的蛋白的技术得到了长足的发展。在Rosetta社区过往的一系列文章中,我们可以看出好几个关键技术的交接点,在2020年以前主要以docking-match、motif-graft、fragment重组技术为主,配合酵母展示库的方法对初筛的weak binder进行优化,而在近两年,RIF和深度学习框架厚积薄发,逐渐开始关注全新PPI和高精度的设计方法,其中最具前沿和代表性的工作就是RIF和RFDesign。

那今天我们就主要来讲一讲基于RIF的通用设计框架的工作原理和成功应用的案例。
二、设计方法
2.1 RIF是什么?
在2011年-2015年期间,产生全新蛋白-蛋白相互作用设计的理念和实践就开始了,但使用的是常规的docking/match-design的方法,其存在最大的问题:hotspot和binding pose的组合空间巨大,通常不能将设计和刚体搜索同时做到, 一般只能先考虑1-3个hotspot的组合并优选少量的初始ppi侧链构象,其次再考虑scaffold和受体之间的粗糙的形状互补后进行热点残基和scaffold和“融合”,最后再通过多轮侧链设计和优化。因此只能在非常小的空间内进行搜索,并且该方法较为依赖多样的scaffold数据库。
而今天介绍的基于RIF(rotamer interaction field)的方法早在2014年间就进行了开发。有兴趣的同学可以看最古老的说明文档的PPT:
Legacy — rif 0.1.dev0 documentation
简单来说RIF分为rifgen和rifdock两个基本的步骤:rifgen负责在某些小分子或热点氨基酸附近以近乎遍历的方式进行rotamer的生成,特别地是使用rif可以针对极性残基也做优化,这些rotamer信息都被储存在hash table中。

rifdock:使用被对接到指定区域的scaffold库作为输入(可以理解为粗略的形状互补搜索),对每一个binding pose的主链xyz信息与rifgen中巨量的inversed-rotamer进行层级逐渐缩小范围的匹配搜索,对clash、rotamer等指标打分进行排名,优选较好的scaffold binding pose。

从此看来,RIF一定程度上解决了需要“同时对接和侧链设计”的痛点,rifgen等于在设计ppi上的残基(“打螺丝”),rifdock负责把预先生成好的稳定scaffold进行“安装”。
这套方法开发出来后,最先尝试的dou jiayi是在小分子中进行de novo binder设计,详见2018年发表的文章:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6275156/
再后来的故事大家也清楚,cao longxing大神在将此方法改进并发扬光大,并设计了对抗新冠的皮摩尔级亲和力的binder:
https://www.science.org/doi/10.1126/science.abd9909
2.2 RIF+MotifGraft横空出世
这套方法流程已经全部概述在下面一张图了。作者这里对新的方法做了一个生动的比喻:将rifgen产生的hotspot-stub比喻为踏脚石,将scaffold比作人,将设计binder比作攀岩的过程,通过地毯式打钉,把人挂在上面,只要挂得住,就把那一系列的好钉子留下,最后通过将高矮胖瘦不同的人去抓这些钉子,从而找到最好的攀岩姿势。

作者提出新的通用设计方法,其主要的亮点在于:
采用了更高效的helical extension产生高质量三螺旋骨架库(34507个);
-
由于蛋白-蛋白相互作用主要是疏水的,因此rifgen只考虑疏水的hotspo stub;
-
使用patchdock进行快速的形状互补后,接上rifdock,可以找到ddG和cms指标更好的pose;
-
采用快速的repack+min对rifdock产生的构象进行打分过滤,并且仅考虑Rosetta的疏水相互作用项使得优化速度提升了10倍;
-
在Interface sequence Design中引入cms(the contact molecular surface) score term,在优化时考虑形状互补以及非极性包埋残基,并且在蒙特卡洛过程中upweight处于ppi界面上氨基酸的score,使得PPI的质量进一步提升;
-
将质量好的binding motif(连续的)进行分离以及聚类,这一步相当于隐式地考虑了scaffold pose的体积,同时把最有可能结合的热点残基筛选;(2000个,有点像binding TERMs的操作)
-
采用motif-graft的细化搜索scaffold和展开新一轮的界面设计。
整个流程图(包括实验):

三、测试效果
作者在12个蛋白靶点上进行了相关的测试,均成功设计出能够特异性靶向结合的mini binder,少数的binder更是突破了pM级别,这些binder高度稳定,表面具有明显的净电荷分布。

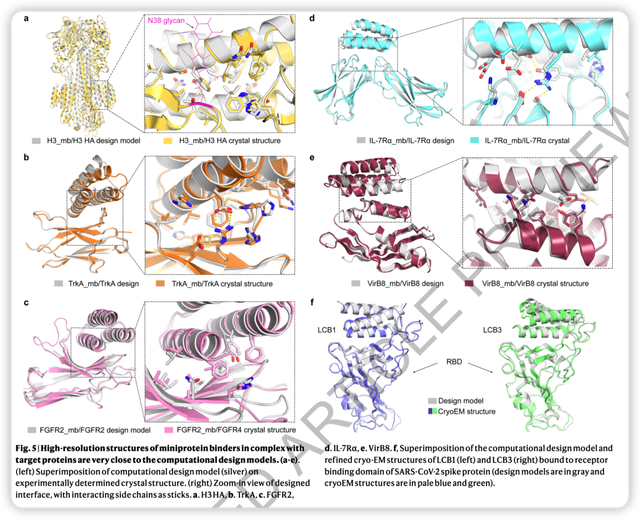
其中部分结构的解析表明,设计出来结合表位和计算机模型高度一致!
结语
作者开源发布所有的rosetta script和database!人人皆可设计binder,或许已经不再遥远。

RIF docking implementation:
-
https://github.com/rifdock/rifdock
design scripts:
-
http://files.ipd.uw.edu/pub/robust_de_novo_design_ minibinders_2021/supplemental_files/scripts_and_main_pdbs.tar.gz
-
http://files.ipd.uw.edu/pub/robust_de_novo_design_minibinders_2021/ supplemental_files/computational_protocol_analysis.tar.gz
-
http:// files.ipd.uw.edu/pub/robust_de_novo_design_minibinders_2021/ supplemental_files/experimental_data_and_analysis.tar.gz
database:
-
http:// files.ipd.uw.edu/pub/robust_de_novo_design_minibinders_2021/ supplemental_file s/scaffolds.tar.gz
models:
-
http://files.ipd.uw.edu/pub/ robustde_novo_design_minibinders_2021/supplemental_files/design models_pdb.tar.gz
-
http://files.ipd.uw.edu/pub/robust_de_novo_ design_minibinders_2021/supplemental_files/design_models_silent. tar.gz























 3303
3303

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








