背景
有一个老师提问:
邓老师,想问一下如何根据猪场子代父母代数据找出它们之间的系谱关系图呢,最好是方便统计的那种

这个问题,可以从以下三个方面解答:
- 1,根据三列系谱,得到15列系谱,这样每个个体,都可以网上追溯三个世代。
- 2,根据系谱计算A矩阵,然后提取感兴趣的个体,可以绘制热图和聚类图
- 3,对于感兴趣的个体,提取他们的三代系谱数据,作图
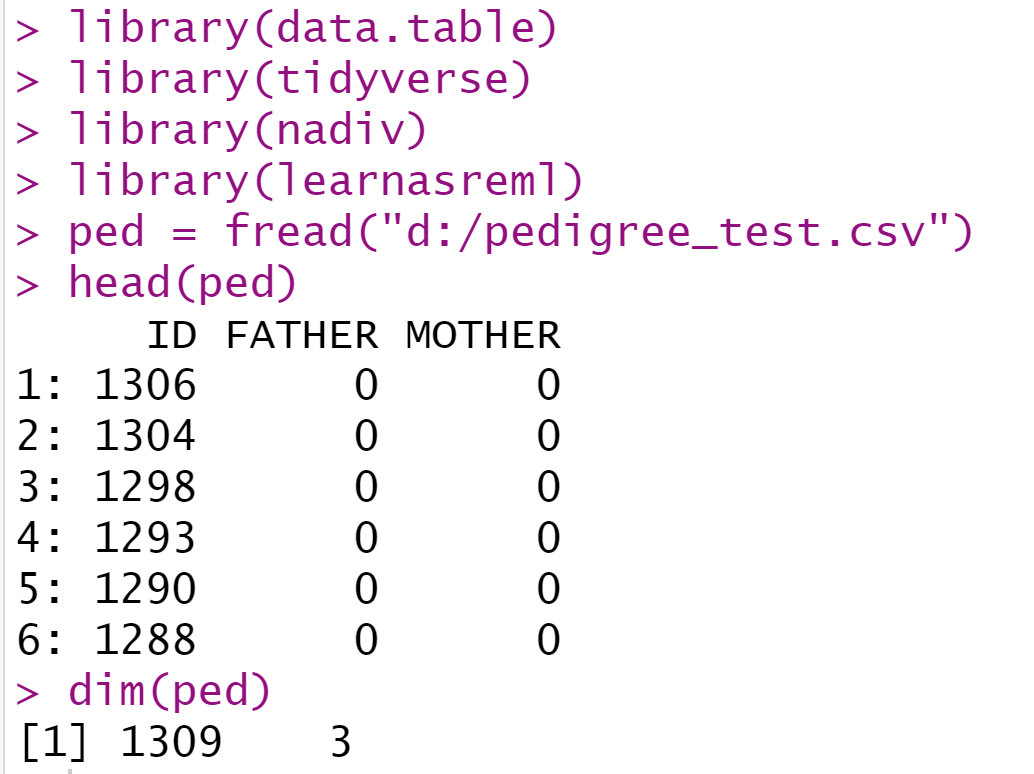
数据介绍:
library(data.table)
library(tidyverse)
library(nadiv)
library(learnasreml)
ped = fread("d:/pedigree_test.csv")
head(ped)
dim(ped)
下面,介绍一下这三种方法的具体实施方法。
1. 追溯3代系谱
首先,这是三列系谱数据,我们需要将其转为15列系谱数据,包括每个个体的三个世代的数据。用的是我写的包learnasreml中的pedigree_3_to_15函数:

如果我们想查看1092个体的三代系谱记录,可以找到ID列是1092的行,分别给出个体的三代系谱数据:
- 个体
- 个体的父本(S),爸爸
- 个体的母本(D),妈妈
- 个体的父本的父本(SS),爷爷
- 个体的父本的母本(SD),奶奶
- 个体的母本的父本(DS),姥爷
- 个体的母本的母本(DD),姥姥
- ……
- ……

如果我们想要筛选个体记录比较完整的个体,可以以DDD为条件,筛选一下:

然后,将其变为3列系谱。这里用到的是pedigree_15_to_3

2. 系谱可视化:聚类和热图分析
上一步中,得到的系谱还有937条,我们对着937条系谱进行可视化。
首先,计算亲缘关系A矩阵:
Amat = makeA(pped1) %>% as.matrix()
Amat[1:10,1:10]

对其进行可视化:

或者单独提取出聚类分析图:

也可以做成圈图的聚类可视化:

当然,上面品种较多,可视化不太直观,如果挑选出感兴趣的少数个体(比如20~50个),用上面的方法是非常方便的。
3. 系谱可视化
系谱可视化,可以使用visPedigree包中的visPedigree
library(visPedigree)
pped2 = visPedigree::tidyped(ped1)
visped(pped2)

可以非常只管的看出个体间的关系。
以上就是根据系谱查看个体间关系的三种方法,希望对大家有所帮助。
相关的数据和代码,我上传到了我的知识星球,欢迎感兴趣的小伙伴下载使用。
领取内容:pdf和配套数据代码


























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








