前言
这是一个数据挖掘选修课的作业,要求是类概念描述和特征化分析,两个词连搜都搜不到,如果你不知道我写的是什么,说实话我也不知道…
使用的语言为python,是直接在Jupyter中写的,中间会有一些输出方便随时进行验证
数据预处理
导入库
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from math import log
读取样本数据
# 读取三种癌症数据集进行分析,查看样本集的规模
BLCA = pd.read_csv(r'数据集/BLCA/rna.csv')
KIRC = pd.read_csv(r'数据集/KIRC/rna.csv')
LUAD = pd.read_csv(r'数据集/LUAD/rna.csv')
print(BLCA.shape)
print(KIRC.shape)
print(LUAD.shape)
(3217, 400)
(3217, 489)
(3217, 491)
# 查看其中一种样本数据的大致结构
BLCA.head(6)
| gene_id | TCGA-HQ-A2OF | TCGA-GU-A767 | TCGA-ZF-AA4R | TCGA-DK-A1AC | TCGA-DK-A3IT | TCGA-GC-A3RD | TCGA-BT-A0YX | TCGA-FD-A6TE | TCGA-E7-A5KE | ... | TCGA-BT-A0S7 | TCGA-K4-A6FZ | TCGA-E5-A2PC | TCGA-DK-AA6Q | TCGA-XF-AAMY | TCGA-CU-A0YO | TCGA-E7-A7PW | TCGA-S5-A6DX | TCGA-GD-A76B | TCGA-ZF-AA4V | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | A2BP1|54715 | -0.402918 | -1.236959 | -1.453230 | -1.211589 | -1.288438 | -1.262086 | -1.033565 | -1.183245 | -1.076254 | ... | -1.282740 | -1.023637 | -0.366722 | -1.161285 | -1.380649 | -1.205136 | -0.875586 | -1.059148 | -1.393928 | -1.215641 |
| 1 | A2ML1|144568 | 0.717502 | 0.737891 | 1.584643 | 1.471932 | 0.414015 | 1.821084 | 2.515898 | 0.723830 | 0.668703 | ... | 0.163903 | 1.318229 | 0.879135 | 0.513603 | 1.571557 | 1.679199 | 1.222708 | -0.079802 | 1.480393 | 1.947340 |
| 2 | ACTL6B|51412 | -1.185185 | -1.403906 | -1.453230 | -1.211589 | -1.288438 | -1.262086 | -1.196075 | -1.183245 | -0.937518 | ... | -1.282740 | -1.291842 | -1.036778 | -1.161285 | -1.380649 | -1.368904 | -1.567590 | -1.425863 | -1.393928 | -1.215641 |
| 3 | ADAM6|8755 | 0.373826 | 1.411279 | 2.092282 | 3.186438 | 2.967457 | 1.437113 | 2.723775 | 2.320012 | 0.736963 | ... | 2.375413 | 1.964369 | 1.202000 | 1.824419 | 2.185051 | 2.175849 | 1.446352 | 3.591872 | 3.266818 | 2.514385 |
| 4 | ADAMDEC1|27299 | -0.489828 | -0.894004 | -0.205169 | 1.015179 | 0.089540 | 0.114903 | 0.664465 | 0.138955 | -1.076254 | ... | -0.408256 | -0.063113 | -0.147173 | 0.177490 | -0.254007 | 0.173888 | -0.951712 | 0.592750 | 0.260382 | 0.758897 |
| 5 | ALDH1A3|220 | 0.420170 | 0.563404 | 1.064257 | 0.460861 | 1.518204 | 1.809320 | 1.230912 | 1.488275 | 0.809302 | ... | 1.180098 | 1.751919 | 0.034835 | 1.512029 | 0.851459 | 1.645271 | 0.398217 | 0.599149 | -0.064256 | 1.339531 |
6 rows × 400 columns
# 对三种癌症样本数据进行转置,之后每一行表示一个样本数据,每一列表示一个样本特征
BLCASet = BLCA.T
BLCASet = BLCASet.iloc[1:,:].astype('float32')
KIRCSet = KIRC.T
KIRCSet = KIRCSet.iloc[1:,:].astype('float32')
LUADSet = LUAD.T
LUADSet = LUADSet.iloc[1:,:].astype('float32')
BLCASet.shape
BLCASet.head(6)
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | ... | 3207 | 3208 | 3209 | 3210 | 3211 | 3212 | 3213 | 3214 | 3215 | 3216 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TCGA-HQ-A2OF | -0.402918 | 0.717502 | -1.185185 | 0.373826 | -0.489828 | 0.420170 | -1.185185 | 1.841492 | 1.327818 | -0.301422 | ... | 0.275846 | 1.228001 | -0.959773 | -0.530559 | 0.637047 | 1.339706 | 0.520305 | 0.235093 | -0.383992 | 0.795605 |
| TCGA-GU-A767 | -1.236959 | 0.737891 | -1.403906 | 1.411279 | -0.894004 | 0.563404 | -1.236959 | 1.309958 | 2.015829 | 0.416162 | ... | 0.364331 | 1.484526 | -1.403906 | 0.689511 | -0.474266 | 0.378506 | 0.994647 | 0.776598 | 0.334363 | 0.577015 |
| TCGA-ZF-AA4R | -1.453230 | 1.584643 | -1.453230 | 2.092282 | -0.205169 | 1.064257 | -1.267946 | 0.207475 | 0.764633 | -0.786075 | ... | 1.350011 | 1.031541 | -1.453230 | 0.867536 | -0.145515 | 0.229990 | 1.210068 | 0.303078 | 0.912720 | 0.709520 |
| TCGA-DK-A1AC | -1.211589 | 1.471932 | -1.211589 | 3.186439 | 1.015179 | 0.460861 | -0.995834 | 1.576376 | 0.546906 | -0.855604 | ... | 0.637691 | 1.190068 | -0.348651 | 0.034641 | 0.030973 | 0.093970 | 1.089523 | 0.583813 | 0.503198 | 0.733853 |
| TCGA-DK-A3IT | -1.288438 | 0.414015 | -1.288438 | 2.967457 | 0.089540 | 1.518204 | -1.159105 | -0.035145 | 1.575849 | 0.220414 | ... | 0.598191 | 1.479815 | -1.288438 | 0.921235 | -0.307700 | 0.486069 | 1.311522 | 0.483139 | 0.262508 | 0.589681 |
| TCGA-GC-A3RD | -1.262087 | 1.821084 | -1.262087 | 1.437113 | 0.114903 | 1.809320 | -0.993603 | 0.878451 | 2.058253 | 0.036055 | ... | 1.069556 | 1.383669 | -0.215558 | 0.234100 | -0.324477 | 0.379689 | 1.323748 | -0.026147 | 0.638123 | 0.096817 |
6 rows × 3217 columns
# 首先对第一种癌症和后两种癌症进行区分,添加一列作为标签
BLCASet.insert(loc=3217, column=3217, value='BLCA')
KIRCSet.insert(loc=3217, column=3217, value='KIRC')
LUADSet.insert(loc=3217, column=3217, value='LUAD')
BLCASet.head(6)
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | ... | 3208 | 3209 | 3210 | 3211 | 3212 | 3213 | 3214 | 3215 | 3216 | 3217 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TCGA-HQ-A2OF | -0.402918 | 0.717502 | -1.185185 | 0.373826 | -0.489828 | 0.420170 | -1.185185 | 1.841492 | 1.327818 | -0.301422 | ... | 1.228001 | -0.959773 | -0.530559 | 0.637047 | 1.339706 | 0.520305 | 0.235093 | -0.383992 | 0.795605 | BLCA |
| TCGA-GU-A767 | -1.236959 | 0.737891 | -1.403906 | 1.411279 | -0.894004 | 0.563404 | -1.236959 | 1.309958 | 2.015829 | 0.416162 | ... | 1.484526 | -1.403906 | 0.689511 | -0.474266 | 0.378506 | 0.994647 | 0.776598 | 0.334363 | 0.577015 | BLCA |
| TCGA-ZF-AA4R | -1.453230 | 1.584643 | -1.453230 | 2.092282 | -0.205169 | 1.064257 | -1.267946 | 0.207475 | 0.764633 | -0.786075 | ... | 1.031541 | -1.453230 | 0.867536 | -0.145515 | 0.229990 | 1.210068 | 0.303078 | 0.912720 | 0.709520 | BLCA |
| TCGA-DK-A1AC | -1.211589 | 1.471932 | -1.211589 | 3.186439 | 1.015179 | 0.460861 | -0.995834 | 1.576376 | 0.546906 | -0.855604 | ... | 1.190068 | -0.348651 | 0.034641 | 0.030973 | 0.093970 | 1.089523 | 0.583813 | 0.503198 | 0.733853 | BLCA |
| TCGA-DK-A3IT | -1.288438 | 0.414015 | -1.288438 | 2.967457 | 0.089540 | 1.518204 | -1.159105 | -0.035145 | 1.575849 | 0.220414 | ... | 1.479815 | -1.288438 | 0.921235 | -0.307700 | 0.486069 | 1.311522 | 0.483139 | 0.262508 | 0.589681 | BLCA |
| TCGA-GC-A3RD | -1.262087 | 1.821084 | -1.262087 | 1.437113 | 0.114903 | 1.809320 | -0.993603 | 0.878451 | 2.058253 | 0.036055 | ... | 1.383669 | -0.215558 | 0.234100 | -0.324477 | 0.379689 | 1.323748 | -0.026147 | 0.638123 | 0.096817 | BLCA |
6 rows × 3218 columns
计算过程
掌握知识有限,只知道如何计算两种类别的特征的信息增益,这里先对第一种和后两种癌症进行区分,之后再对后两种癌症做区分
把后两种癌症数据合并为一个数据集,并对它们做一些方便后续计算的处理
# 先对第一种和第二三种进行区分
First_Set = np.array(BLCASet)
Second_Set = pd.concat([KIRCSet,LUADSet],axis=0)
Second_Set = np.array(Second_Set)
First_Set
array([[-0.40291815996170044, 0.7175019979476929, -1.1851845979690552,
..., -0.38399189710617065, 0.7956054210662842, 'BLCA'],
[-1.2369587421417236, 0.7378911972045898, -1.4039058685302734,
..., 0.33436259627342224, 0.5770151615142822, 'BLCA'],
[-1.4532300233840942, 1.5846433639526367, -1.4532300233840942,
..., 0.9127196669578552, 0.7095199823379517, 'BLCA'],
...,
[-1.0591480731964111, -0.07980185002088547, -1.4258626699447632,
..., -0.03409137204289436, 0.35402581095695496, 'BLCA'],
[-1.3939282894134521, 1.4803926944732666, -1.3939282894134521,
..., 0.6251711845397949, 0.7505104541778564, 'BLCA'],
[-1.2156405448913574, 1.9473400115966797, -1.2156405448913574,
..., 0.7588967680931091, 0.9871674180030823, 'BLCA']],
dtype=object)
用来分类一个给定样本的期望信息的计算:
计算每个属性的熵
def I(s1, s2):
if s1==0 or s2==0:
return 0
prob1 = float(s1/(s1+s2))
prob2 = float(s2/(s1+s2))
return -prob1*log(prob1,2)-prob2*log(prob2,2)
计算给定的样本分类所需要的期望信息
# 计算给定的样本分类所需要的期望信息
I_First_Second = I(len(First_Set), len(Second_Set))
I_First_Second
0.8684114715162274
要计算每种属性特征的信息增益,对于离散值,我们可以直接统计每种类型的数量,进而计算熵和信息增益,而样本数据集每种属性是连续的值而非离散,这里采取的处理方法是:对于要计算信息增益的属性,遍历其所有的属性值,以每个属性值为分界将数据分成大于该值和小于该值两部分,对划分后的数据进行信息增益的计算,遍历完当前属性的所有取值,得到以每个属性值为分界所计算得到的信息增益,取这些结果中最大的信息增益作为当前属性的信息增益值
定义上述计算的函数:
# 对于离散值的处理,采用取值的方式,分为大于该值和小于该值两类
def Feature_Gain(First_Set, Second_Set):
Gain = np.zeros(len(First_Set[0])-2)
divPointSet = np.zeros(len(First_Set[0])-2)
for i in range(len(First_Set[0])-2):
for j in range(len(First_Set)-1):
maxG = float('-inf')
divPoint = First_Set[j][i]
s11 = 0
s12 = 0
featList = [example[0] for example in First_Set]
for value in featList:
if value < divPoint:
s11 = s11+1
else:
s12 = s12+1
s21 = 0
s22 = 0
featList = [example[0] for example in Second_Set]
for value in featList:
if value < divPoint:
s21 = s21+1
else:
s22 = s22+1
# 计算以divPoint划分下该属性的信息增益
G = I_First_Second-\
(s11+s21)/(len(First_Set)+len(Second_Set))*I(s11,s21)-\
(s12+s22)/(len(First_Set)+len(Second_Set))*I(s12,s22)
if G>maxG:
Gain[i] = G
divPointSet[i] = divPoint
maxG = G
return Gain, divPointSet
计算所有属性的信息增益
计算得到所有属性的信息增益
Gain, divPointSet = Feature_Gain(First_Set, Second_Set)
根据信息增益大小,根据大小对下标进行排序
tmp = np.argsort(Gain)
print(tmp)
[2073 515 1941 ... 1331 2299 2617]
以上排序结果说名,第2617个属性中存在令信息增益值最大的一个划分
Gain[2617]
0.018568131349944283
divPointSet[2617]
-1.2444435358047485
DataSet = pd.concat([BLCASet,KIRCSet,LUADSet],axis=0)
结果可视化
经过上述的计算,此时已经得到各属性可能的信息增益大小,可以利用可能信息增益较大的几种属性和可能信息增益较小的属性对几种癌症进行区分,对比它们的效果
选择可能信息增益较小的属性值对样本数据分布进行绘制
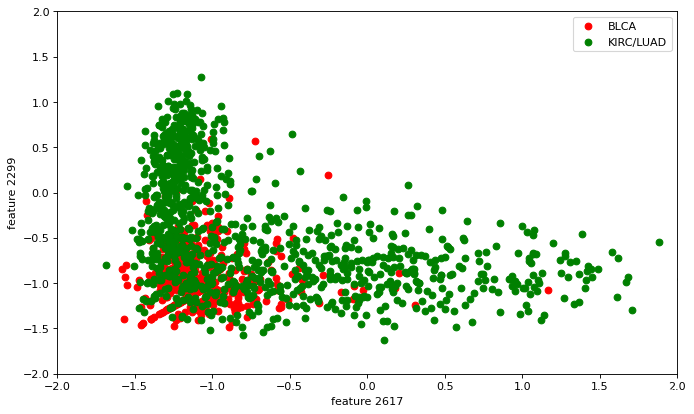
信息增益较大的属性下的结果
plt.figure(figsize=(10,6), dpi=80)
plt.xlim(-2,2)
plt.ylim(-2,2)
plt.scatter(First_Set[:,2617],First_Set[:,2299],label='BLCA', c='red')
plt.scatter(Second_Set[:,2617],Second_Set[:,2299],label='KIRC/LUAD', c='green')
plt.xlabel('feature 2617')
plt.ylabel('feature 2299')
plt.legend(loc='upper right')
plt.show()
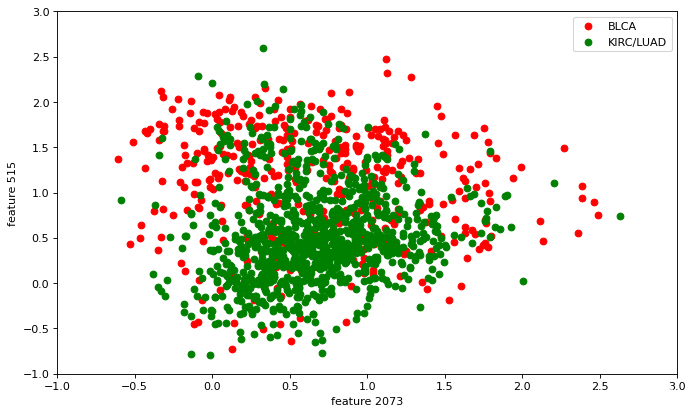
信息增益较小的属性下的结果
plt.figure(figsize=(10,6), dpi=80)
plt.xlim(-1,3)
plt.ylim(-1,3)
plt.scatter(First_Set[:,2073],First_Set[:,515],label='BLCA', c='red')
plt.scatter(Second_Set[:,2073],Second_Set[:,515],label='KIRC/LUAD', c='green')
plt.xlabel('feature 2073')
plt.ylabel('feature 515')
plt.legend(loc='upper right')
plt.show()
# 可以按照同样的思路对剩余的两种癌症进行区分
KIRC_Set = np.array(KIRCSet)
LUAD_Set = np.array(LUADSet)
I_KIRC_LUAD = I(len(KIRC_Set), len(LUAD_Set))
I_KIRC_LUAD
0.9999969833346999
Gain1, divPointSet1 = Feature_Gain(KIRC_Set, LUAD_Set)
tmp1 = np.argsort(Gain1)
print(tmp1)
[2659 1152 1156 ... 944 0 2781]
plt.figure(figsize=(10,6), dpi=80)
plt.xlim(-2,2)
plt.ylim(-2,2)
plt.scatter(KIRC_Set[:,2781],KIRC_Set[:,0],label='KIRC', c='red')
plt.scatter(LUAD_Set[:,2781],LUAD_Set[:,0],label='LUAD', c='green')
plt.xlabel('feature 2781')
plt.ylabel('feature 0')
plt.legend(loc='upper right')
plt.show()
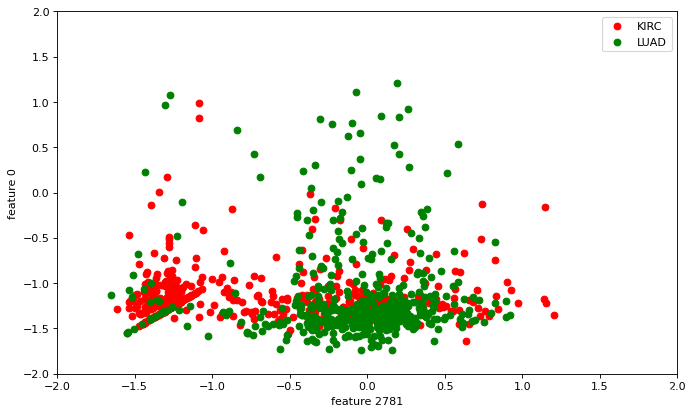
plt.figure(figsize=(10,6), dpi=80)
plt.xlim(-1,2)
plt.ylim(1.5,4)
plt.scatter(KIRC_Set[:,2659],KIRC_Set[:,1152],label='KIRC', c='red')
plt.scatter(LUAD_Set[:,2659],LUAD_Set[:,1152],label='LUAD', c='green')
plt.xlabel('feature 2781')
plt.ylabel('feature 0')
plt.legend(loc='upper right')
plt.show()
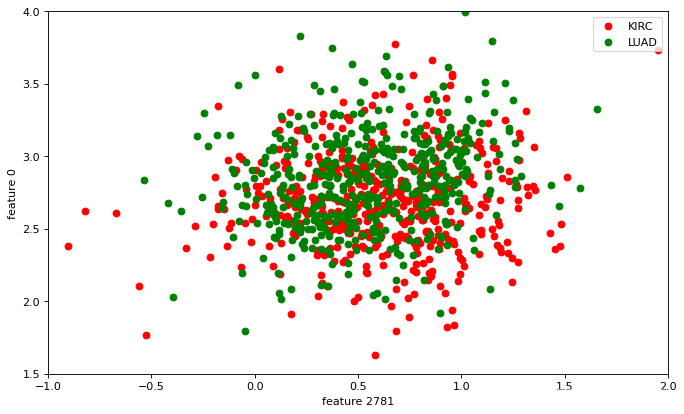
写在后面
至于这样的结果能说明什么,比较信息增益较大和较小的特征下的数据绘制的结果来看,使用信息增益较小的数据绘制出的图像,两种类型的样本点“均匀”分布,换句话说就是很难区分开;而使用信息增益较大的特征数据绘制出的图像,虽然也没好到哪去(特征维数太多了),但直观上可以感到比前者更易区分两种样本类型(大概)。这时因为信息增益较大的特征,意味着其不确定性下降更大,即更易于将两种事物区分开。








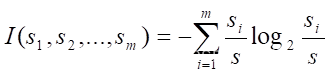














 1596
1596











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










