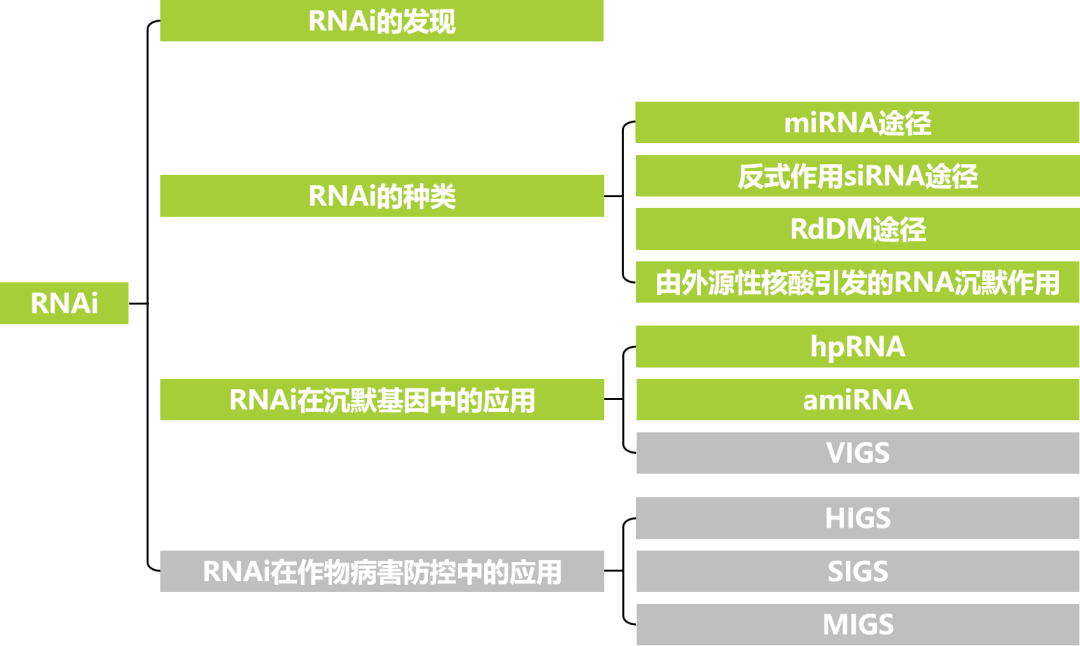
本文内容速览:
基因编辑的风近几年一直很热,热到大家似乎不怎么记得有一种技术叫RNAi,不知道有没有说到你的心坎里去。其实在植物基因功能研究中,基因编辑和RNAi都是常用的手段,它们各有优势和劣势。基因编辑(如CRISPR-Cas9)主要通过精确修改基因序列来改变基因功能,而RNAi是通过阻碍特定基因的转录或翻译来抑制基因表达。在实际的应用中选择哪种方法取决于研究目标和具体的实验条件。
在某些特殊情况下,例如某些与生长发育相关基因的纯合突变体会致死,又或者想要特异敲低同一基因多个可变剪切体中的特定剪切体,针对这些情况基因编辑往往不是最佳选择,此时我们应该将目光转向RNAi技术,因为RNAi作用于靶基因的mRNA而非DNA,可以有效解决上述问题。
今天这篇文章主要围绕RNAi技术的发现、类型以及RNAi在基因功能研究以及农业生产中的应用进行展开,帮大家重新认识一下我们的老朋友RNAi。
01
RNAi的发现与历史
第一篇关于RNAi(RNA interference,RNAi)的论文可能早在1928年就已发表(Wingard, S. A., 1928)。在这篇论文中,温加德描述了烟草植株的情况,即只有最初受感染的叶片会出现坏死和病变,而其他叶片则未受影响(图1)。上述叶片不知为何对烟草环斑病毒产生了免疫力,不仅没有表现出症状,对二次感染也具有抵抗力。当时,这种“恢复”现象令人困惑,无法找到合理的解释来说明这种针对二次感染的特异性抵抗力。随着对烟草环斑病毒这一案例的进一步研究,我们如今了解到,植物从病毒病害中恢复的过程涉及一种名为“RNAi”的机制,该机制能够特异性地靶向并降解病毒的RNA (Ratcliff F et al., 1997; Covey et al., 1997),关于RNAi的发展历程具体可见图1。
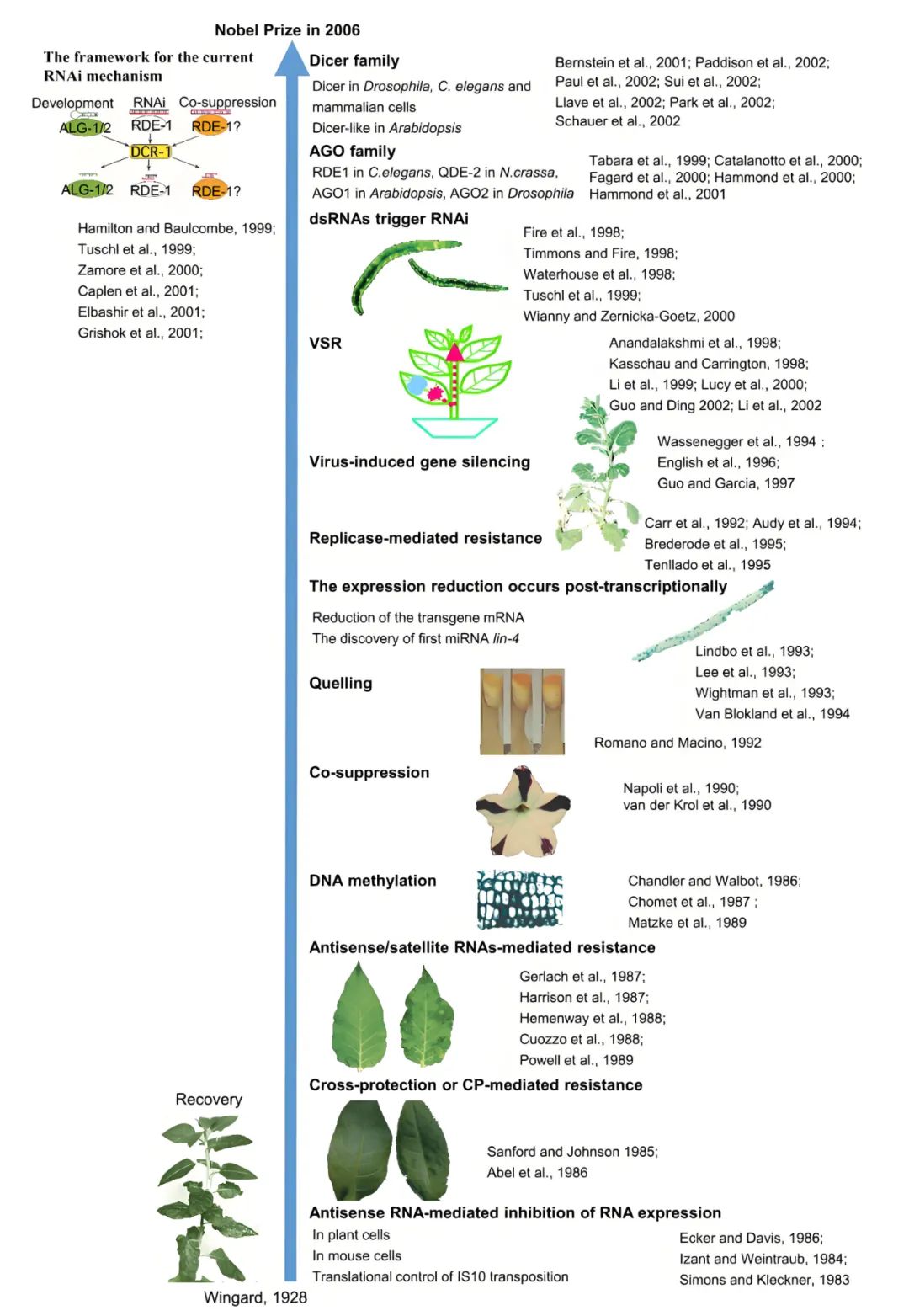
图1 RNAi通路发现和阐明的重要进展时间表(Zhao et al., 2022)。
注:图片中提到的参考文献大家感兴趣可至图1的原文献进行查看,本推文就不再一一列举了。
02
RNAi的类型
RNAi是实现基因沉默的一种经典途径,当RNAi途径激活时,就会导致基因沉默。在大多数真核生物中,RNAi是一种进化上保守且具有序列特异性的调控机制,它能在转录水平(转录基因沉默(TGS))或转录后水平(转录后基因沉默(PTGS))上调节基因表达。原则上,RNAi是由内源性或外源性双链RNA(Double-stranded RNA,dsRNA)或发夹结构RNA(Hairpin structured RNA,hpRNA)诱导产生的。RNAi途径在植物中高度分化,以适应不同的功能需求(David Baulcombe, 2004; Eamens et al., 2008)。根据dsRNA或hpRNA前体的来源以及小RNA(Small RNA,sRNA)的作用靶点,植物中的RNAi可划分为4种相互重叠但功能不同的途径:微小RNA(miRNA)途径、反式作用siRNA(tasiRNA)途径、RNA介导的DNA甲基化途径以及外源RNA沉默途径。
2.1
miRNA途径
miRNAs是由MIR基因的遗传位点衍生出的20-24nt的短RNA(David P. Bartel, 2009)。与蛋白质编码基因一样,MIR基因由RNA聚合酶II转录生成pri-miRNA(初级微小RNA转录本)。由于分子内序列互补的存在,pri-miRNA形成一个不完全配对的“回折”茎环或发夹结构,随后在细胞核中由DCL1与双链RNA结合蛋白DRB1或HYL1的协助下被加工成一个pre-miRNA(短的“茎环”前体)(Yukio Kurihara and Yuichiro Watanabe, 2004; Kurihara et al., 2006; Eamens et al., 2009)。接着pre-miRNA分子在细胞核内进一步被DCL1加工,产生一个长度为21nt的不完全配对RNA双链体。此双链体包含成熟的miRNA(引导链)和miRNA*(miRNA随从链)。
RNA双链体(miRNA:miRNA*)的3’末端核苷酸会在2’-O羟基上被HEN1(RNA甲基化酶HUAENHANCER1)甲基化,该酶的作用是保护miRNA:miRNA*双链免受降解(Li et al., 2005)。随后,miRNA:miRNA*双链会被转运至细胞质中,在那里成熟的miRNA会与AGO蛋白(主要是AGO1)结合形成RISC。与通常以不完全匹配靶向mRNA的3’非翻译区(UTR)来抑制蛋白质翻译的动物miRNA不同,植物miRNA通常与靶mRNA具有高度的序列互补性,并直接进行序列特异性RNA切割(Anthony A. Milar & Peter M. Waterhouse, 2005)。植物中miRNA的主要靶基因是调节基因,如转录因子基因。因此,miRNA在植物发育中起着关键作用。
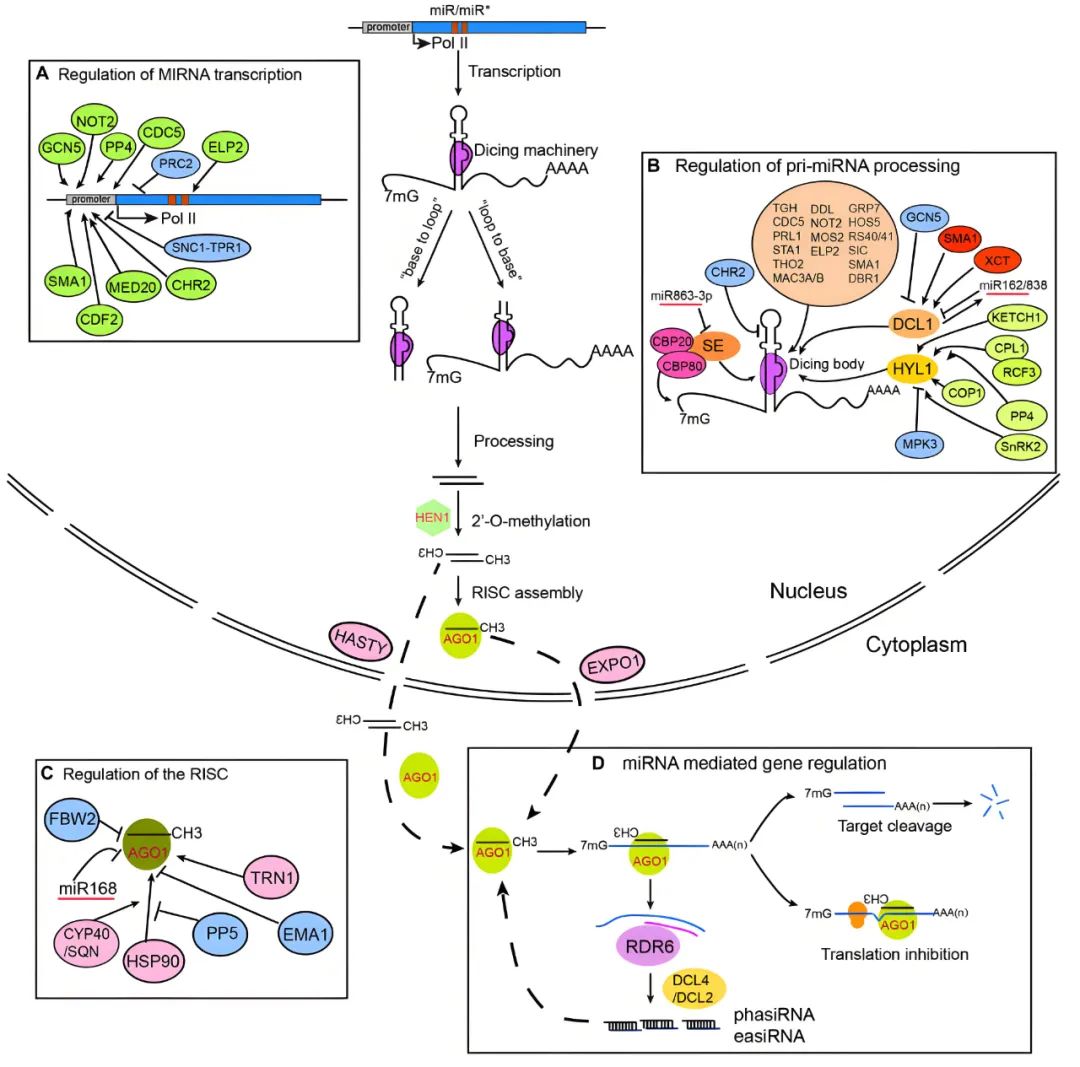
图2 植物中miRNA的生成调控、RISC加载以及其作用机制(Wang et al., 2019)。
2.2
反式作用siRNA途径
与miRNA类似,反式作用siRNA(tasiRNA)也是一类21nt非编码sRNA,它们由RNA聚合酶II从特定的TAS基因位点转录产生 (Vazquez et al., 2004; Allen et al., 2005; Peragine et al., 2004)。tasiRNA的生物合成由特定的miRNA启动,这些miRNA引导TAS前体RNA的切割。TAS转录本经miRNA切割后产生的片段,被RDR6(RNA依赖的RNA聚合酶6)转化为dsRNA。随后dsRNA被DCL4(Dicer-like 4)加工处理,从miRNA切割位点开始,以21nt的相位切割成21nt的siRNA。
对于启动tasiRNA的产生,miRNA的长度至关重要。目前发现只有22nt的miRNA能够触发tasiRNA的生物合成(Cuperus et al., 2010; Chen et al., 2010)。21-nt类别的miRNA,或者由能诱导tasiRNA的22nt miRNA改造而来的21nt人工miRNA变体,都无法触发tasiRNA的产生。与miRNA类似,tasiRNA会被HEN1甲基化修饰(Li et al., 2005),并与AGO1或AGO7相互作用,引导靶标mRNA的降解。
知识补充:
在拟南芥以及其他植物物种中,已发现了大量类似于tasiRNA的siRNA,统称为相位siRNA(phasiRNAs)(Johnson et al., 2009; Zhai et al., 2011; Fei et al., 2013; Creasey et al., 2014)。这些phasiRNAs与最初定义的tasiRNA相同,均为21nt,并需要22nt的miRNA、AGO1、RDR6和DCL4来生成。来自蛋白质编码基因和诸如转座子和重复DNA等非编码序列的RNA都可以作为phasiRNAs的模板。
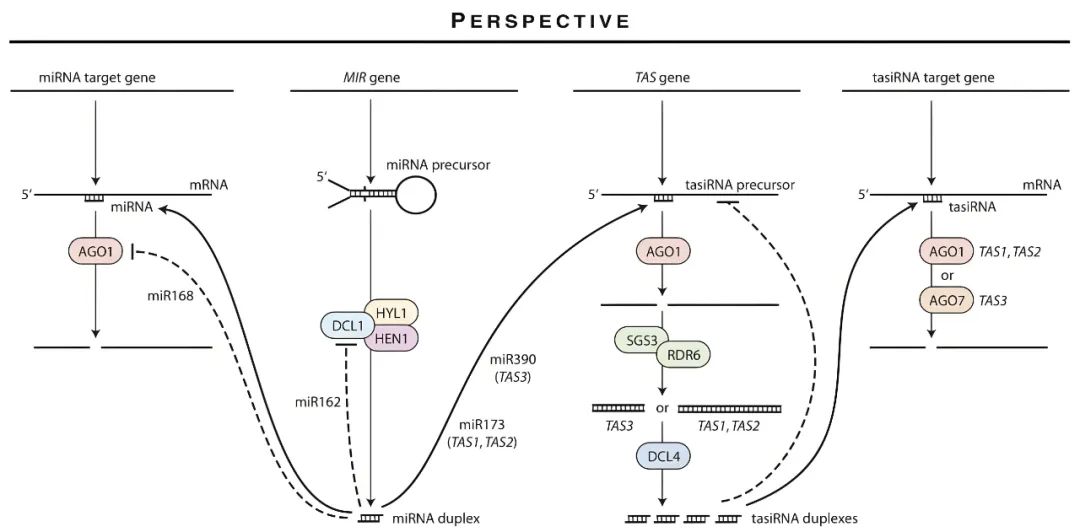
图3 miRNAs和tasiRNA生物合成和功能发挥的模式图(Hervé Vaucheret, 2005)。
2.3
RNA介导的DNA甲基化(RdDM)途径
RNA介导的DNA甲基化(RdDM)途径是植物所特有的,它在细胞核中介导de novo DNA甲基化(DNA从头甲基化)和转录沉默(Feng et al., 2010; Haag et al., 2011; Zhang et al., 2013; Matzke et al., 2015)。RdDM由24nt siRNA(24核苷酸小干扰RNA)所介导,这些siRNA由植物特异的RNA聚合酶IV(PolIV)、RDR2(RNA依赖的RNA聚合酶2)和DCL3(Dicer-like 3)协同作用产生。简而言之,PolIV转录甲基化的、高度重复的DNA,产生异常RNA,随后RDR2将此单链RNA(Single-stranded RNA, ssRNA)转化为dsRNA,dsRNA再由DCL3加工成24-nt siRNA。这些siRNA还会被HEN1(Li et al., 2005)在其末端核苷酸的3’羟基基团上进行甲基化修饰。24nt siRNA被装载到AGO4(Argonaute4)上形成RISC,此过程涉及细胞核和细胞质中的步骤(Ye et al., 2012)。
接着,AGO4-siRNA复合体与由另一种植物特异的PolV(RNA聚合酶V)从靶标DNA转录出的长链非编码RNA(Long non-coding RNA, LncRNA)相互作用,从而招募包括DRM2(结构域重排甲基转移酶2)在内的其他因子,最终导致de novo DNA胞嘧啶甲基化。在对称的CG和CHG(H代表A、C或T)位点上发生的de novo胞嘧啶甲基化,在DNA复制过程中分别由MET1(甲基转移酶1)和CMT3(染色体甲基化酶3)维持。然而,发生在非对称CHH位点上的de novo胞嘧啶甲基化无法在DNA复制过程中维持,因此完全依赖于RdDM通路。RdDM的主要功能是沉默TEs(转座元件)和重复DNA,以维持基因组稳定性。事实上,24-nt siRNA也被称为rasiRNA(重复序列相关siRNA),因为大多数这类siRNA来源于植物基因组中的转座元件和重复DNA。有关RdDM途径的运行及其调控的机制尚未完全探明,现有研究支持一种共转录模型(Marjori A. Matzke and Rebecca A. Mosher, 2014)(图4)。
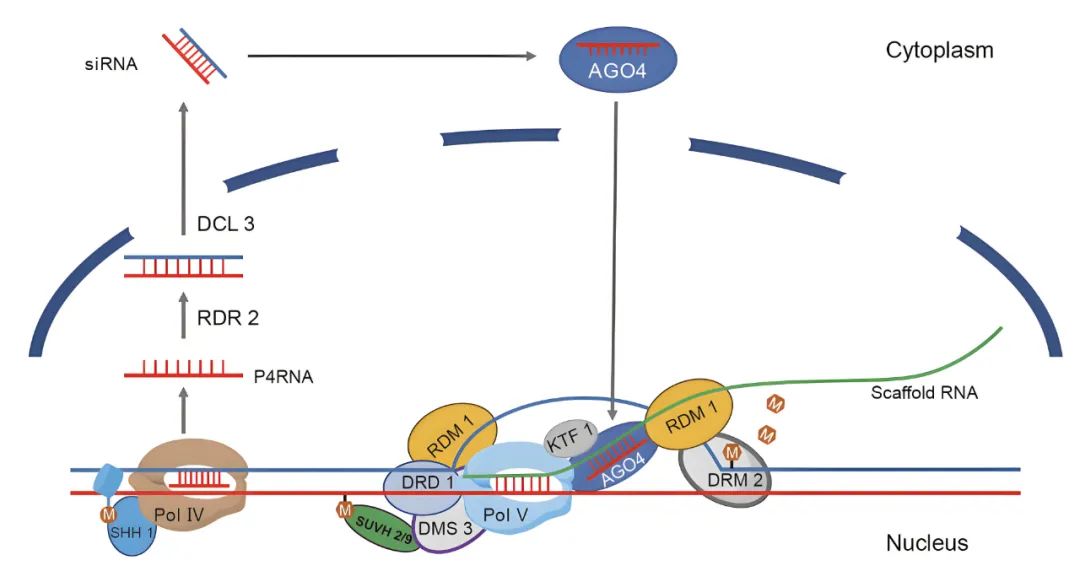
图4 RdDM的共转录模型(胡港和徐强,2021)。
2.4
由外源性核酸引发的RNA沉默作用
植物体内可通过入侵的核酸分子引发RNA沉默现象。在本文中“外源性RNA沉默”这一术语指的是由正义转基因(以天然编码方向(与内源基因相同)插入的转基因)和病毒所引发的RNA沉默现象。RNA沉默的现象最初是在对正义转基因的研究中被观察到的,研究结果表明,设计用于在番茄中过度表达色素合成酶的转基因不仅会自我沉默,还会导致内源性对应基因的沉默,从而导致花朵失去色素(Napoli etal., 1990; Van et al., 1990)(具体的案例小远在之前的推文“崩溃!超表达植株里的目的基因并没有被超表达??”中讲过,感兴趣的小伙伴可以去看看噢)。表明RNA为基因沉默诱导因子的首个证据也来自对植物中由正义转基因介导的病毒抗性的研究,在这些研究中,病毒衍生的转基因的表达会诱导序列特异性RNA降解,从而导致病毒抗性(Lindbo et al., 1993)。外源性RNA沉默与内源性siRNA和RdDM途径重叠。事实上,我们对这些内源性siRNA沉默途径的理解大多来自使用转基因和病毒作为模型的研究。
2.4.1 编码基因诱导的RNA沉默
正义转基因既可以在转录水平上被沉默(TGS),也可以在转录后水平上被沉默(PTGS)。当转基因以多拷贝重复的形式整合到植物基因组中时,这种沉默现象常常发生(David Baulcombe, 2004; Waterhouse et al., 2001)。目前对于TGS和PTGS的确切机制尚未完全阐明。一般而言,TGS与转基因启动子区域的DNA甲基化有关,而该甲基化很可能是由RdDM途径诱导的。事实上,人工表达靶向转基因启动子的长链发夹RNA(long hpRNA)能够诱导该启动子区域的DNA甲基化,从而导致转基因的转录沉默。多拷贝转基因重复序列有可能被PolIV和RDR2识别,从而产生触发RdDM的24-nt siRNA。另一种可能是,跨越多个转基因重复序列的连续转录会产生启动子转录本,这种转录本进而可以导致24-nt siRNA的产生并引发RdDM。
正义转基因的PTGS需要RDR6、DCL4、SGS3和AGO1的参与(Eamens et al., 2008; de Alba et al., 2013),因此与tasiRNA途径相似。PTGS的两个特性:传递性和系统性移动,都涉及到在主要靶位点之外区域产生21-nt siRNA(Vermeersch et al., 2013; Brosnan et al., 2007; Mlotshwa et al., 2008; Melnyk et al., 2011),这表明类似于tasiRNA的次级siRNA是PTGS的一个重要组成部分。
2.4.2 抗病毒RNA沉默
任何类型的病毒或亚病毒对植物的感染都会导致病毒sRNA的积累,这些sRNA是由相同的宿主植物siRNA生物合成机制从双链病毒RNA中加工而来,并利用相同的AGO蛋白直接降解单链病毒RNA(Shou-Wei Ding and Olivier Voinnet, 2007; Wang et al., 2012)。因此,病毒既是siRNA途径的诱导者,也是其靶向目标。实际上,RNA降解沉默已被认为是植物中一种天然的抗病毒防御机制(Waterhouse et al., 2001)。
03
RNAi在植物基因功能研究中的应用
自1998年发现dsRNA引起的基因沉默现象以及随后对植物中各种siRNA通路的阐明以来,人们已经开发出了多种基于转基因或病毒的基因沉默技术,以人工诱导植物中的RNA沉默。这些技术,特别是hpRNA、amiRNA以及VIGS技术,已被广泛应用于基因功能研究和植物遗传工程以改良作物品种。
3.1
hpRNA
1998年,研究人员首次证明了旨在表达发夹RNA(hairpin RNA,hpRNA)的转基因在植物中诱导RNAi的效果极为显著(Waterhouse et al., 1998; Ming-BoWang and PeterM.Waterhouse, 2000)。此后,hpRNA转基因被广泛用于抑制植物中的基因和病毒RNA(Watson et al., 2005)。典型的hpRNA载体由目标基因mRNA一部分的正向和反向序列作为反向重复序列构成,这些反向重复序列之间由非互补的间隔区隔开。间隔区主要用于在细菌细胞中克隆hpRNA时稳定构建体,因为完全反向重复的DNA在细菌中非常不稳定。此外,使用可剪接的内含子作为间隔区已被证明能提高植物中的RNA抑制效率(Smith et al., 2000)。转录的RNA中的正向和反向序列彼此互补,形成一个dsRNA臂。因此,由hpRNA转基因诱导的抑制不应需要RDRs来生成dsRNA,hpRNA主要由DCL4处理生成21nt的siRNA,21nt的siRNA与AGO蛋白结合形成RISC靶向mRNA导致基因沉默。
在植物基因功能研究中,我们常常需要对目的基因进行敲低以验证基因的功能,这种案例大家已经非常熟悉了,所以不再具体展示,这里小远想给大家介绍一种一次沉默多个基因的hpRNA载体的构建方法,具体的文献案例可能不是验证基因功能,而是培育抗病毒水稻,与这里的大标题稍稍有点不挂钩,但举一反三,重点在于具体的实验方法可为我们所用。
文献案例:
RNAi是植物抵抗病毒侵染最重要的途径之一,也是人工定向改良作物对病毒抗性的有效手段。通过表达针对病毒基因序列的发夹RNA,可产生大量特异性靶标降解病毒RNA的siRNA,显著提升植物对病毒的抗性。虽然已有研究通过RNAi技术获得了对病毒高抗的水稻株系,但以往获得的株系都是针对单一病毒,尚无同时针对多种病毒的高抗水稻株系。
2024年3月,中国农业科学院/浙江大学周雪平团队联合江苏省农业科学院周彤团队在Plant Biotechnology Journal杂志上发表了一篇题为“Development of a transgenic rice line with strong and broad resistance against four devastating rice viruses through expressing a single hairpin RNA construct”的研究论文,该文章报道了通过RNAi在水稻中转基因表达了针对水稻黑条矮缩病毒(RBSDV)、南方水稻黑条矮缩病毒(SRBSDV)、水稻条纹病毒(RSV)和水稻锯齿叶矮缩病毒(RRSV)等4种病毒基因序列的融合发夹,从而获得了同时对4种病毒高抗的水稻转基因株系ZJU-4K。
作者通过分析这4种病毒的关键蛋白编码序列,分别从中克隆出300bp的保守片段,将它们融合成单一的发夹RNA结构,并通过农杆菌介导的方式转化水稻愈伤(图5a)。在筛选鉴定后,成功获得了稳定遗传的T4代转基因水稻株系ZJU-4K(图5b)。sRNA测序结合RT-qPCR检测表明,ZJU-4K水稻植株中稳定产生了大量针对病毒基因保守序列的siRNA(图5c、d)。通过人工接种,研究团队系统地评价了ZJU-4K水稻对RBSDV、SRBSDV、RSV、RRSV等4种靶标病毒的抗性。与野生型受体水稻植株相比,ZJU-4K水稻展现出了对靶标病毒的极高抗性,发病率和病毒积累量都出现了极显著的降低(图5e-l)。特别是针对SRBSDV和RSV,ZJU-4K水稻可以取得近乎于免疫的效果。
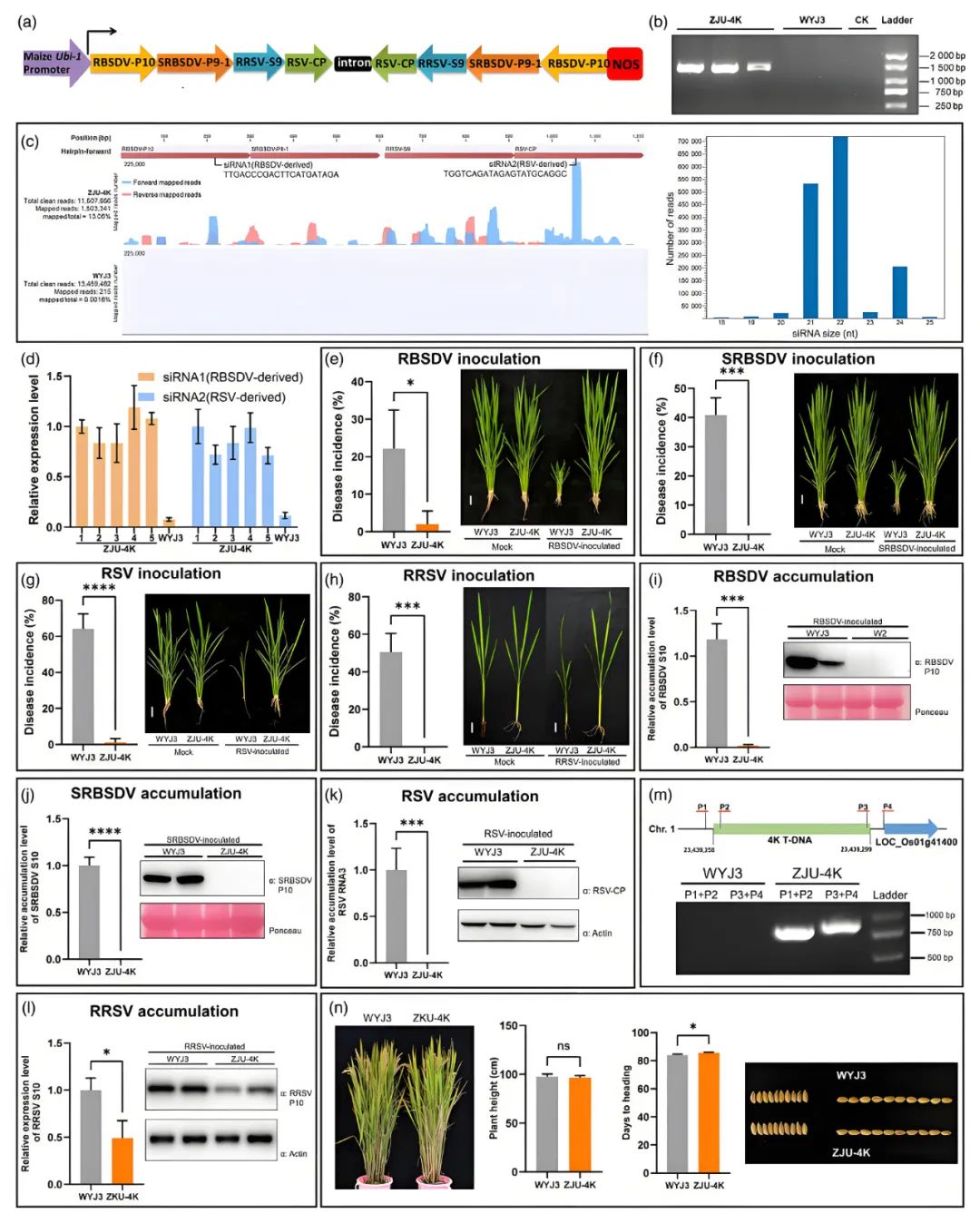
图5 培育出了一种对四种水稻病毒具有强大且广泛抗性的转基因水稻品系(Li et al., 2024)。(a)嵌合发夹RNA(hpRNA)结构示意图;(b)ZJU-4K(T4代)和野生型武育粳3号(WYJ3)水稻植株中hpRNA的PCR分析;(c)ZJU-4K(T4代)与WYJ3小RNA测序分析,左图:siRNA分布图谱,右图:hpRNA来源siRNA的长度分布直方图;(d)ZJU-4K(T4代)单株hpRNA来源siRNA的RT-qPCR检测;(e–h)ZJU-4K(T4代)与WYJ3接种四种病毒后的发病率及症状;(i–l)ZJU-4K(T4代)与WYJ3植株的病毒积累量检测;(m)ZJU-4K(T4代)的T-DNA插入位点验证;(n)ZJU-4K(T4代)与WYJ3的农艺性状比较。
对于同时沉默多个基因,我司还有另外两种载体构建方式可提供给大家参考(文献案例中是将多个基因的序列串在一起作为一个整体的干扰片段),如果大家在自己的科学研究中也需要同时沉默多个基因,可以从这些方法中任选其一噢!
方式一:

方式二:

3.2
amiRNA
在对拟南芥中miRNA的生成机制及miRNA前体结构有了深入理解之后,人们开发出了人工miRNA(amiRNA)这一技术,用以在植物中实现基因沉默(Schwab et al., 2006)。简而言之,amiRNA载体是通过将天然miRNA前体中的miRNA和miRNA*序列替换为相应的靶基因序列,同时保留原始前体的茎环结构而制成的。amiRNA载体中的miRNA链包含与靶mRNA相互补的序列,而miRNA*链的设计目的是维持原miRNA前体的miRNA:miRNA*双链结构(Schwab et al., 2006)。因此,amiRNA转基因是利用内源性miRNA途径来实现基因沉默的。
amiRNA技术相对于hpRNA技术的主要优势在于,其沉默作用由单一的sRNA介导,仅靶向一段21nt的短区域,因此具有更低的脱靶效应风险。然而,amiRNA的设计需要精心选择序列,以满足与AGO高效结合。此外,amiRNA介导的沉默过程不涉及二次siRNA的扩增,这使得amiRNA在抑制低丰度转录物方面可能比抑制高丰度转录物更有效。
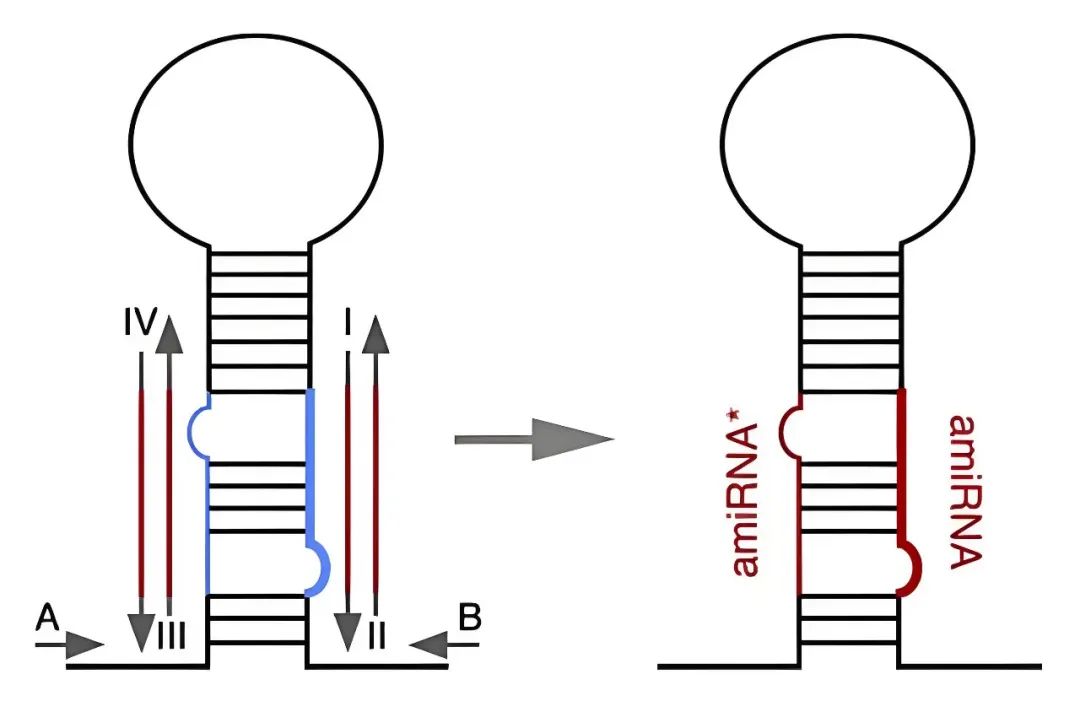
图6 amiRNA载体的构建方式(Schwab et al., 2006)。通过重叠PCR技术对内源性miRNA的前体进行定点突变。该技术使用寡核苷酸引物I至IV将miRNA和miRNA区域(蓝色)替换为人工设计的序列(红色)。引物A和B的设计基于模板质粒序列。通过将PCR产物A-IV、II-III和I-B在引物A和B存在下进行单管组合扩增,实现功能性miRNA前体的重新构建。
文献案例:
2025年4月,中国农业科学院生物技术研究所张海文联合衡阳师范大学等单位在Rice杂志上发表了一篇题为“OsERF2 Acts as a Direct Downstream Target of OsEIL1 to Negatively Regulate Salt Tolerance in Rice”的研究论文,该研究揭示了水稻ERF转录因子OsERF2在盐胁迫响应中的功能及其调控机制。研究发现,OsERF2作为乙烯信号通路关键转录因子OsEIL1的直接下游靶基因,负向调控水稻的耐盐性。该调控作用主要通过影响活性氧(ROS)的积累来实现,其中OsERF2直接结合并激活ROS产生关键基因OsRbohH的表达。这一发现阐明了水稻中一条新的乙烯信号介导的盐胁迫响应通路(OsEIL1-OsERF2-OsRbohH),为水稻耐盐遗传改良提供了新的理论依据和潜在靶点。
在这篇文章中作者为了验证OsERF2的功能,构建了OsERF2的amiRNA干扰株系(Ami-OsERF2,表达下调)和利用了一个已知的OsERF2功能获得性突变体nsf2857(组成性过表达)。在盐胁迫(将生长3周龄的苗用120mM NaCl处理8天)处理下,与野生型对照(Nip)相比,三个独立的Ami-OsERF2株系均表现出显著增强的耐盐性,存活率提高了约1.5倍(图7A,C)。相反,nsf2857突变体则对盐胁迫高度敏感,几乎无法存活,而其野生型对照(ZH11)仍有约60%的存活率(图7A,C)。此外,对OsERF2转基因株系中OsERF2的表达水平进行了定量检测,结果显示Ami-OsERF2株系表达量显著降低,而nsf2857显著升高(图7B)。类似的负调控现象在干旱胁迫实验中也得到验证。这些表型结果清晰地表明,OsERF2负向调控水稻对盐胁迫和干旱胁迫的耐受性。
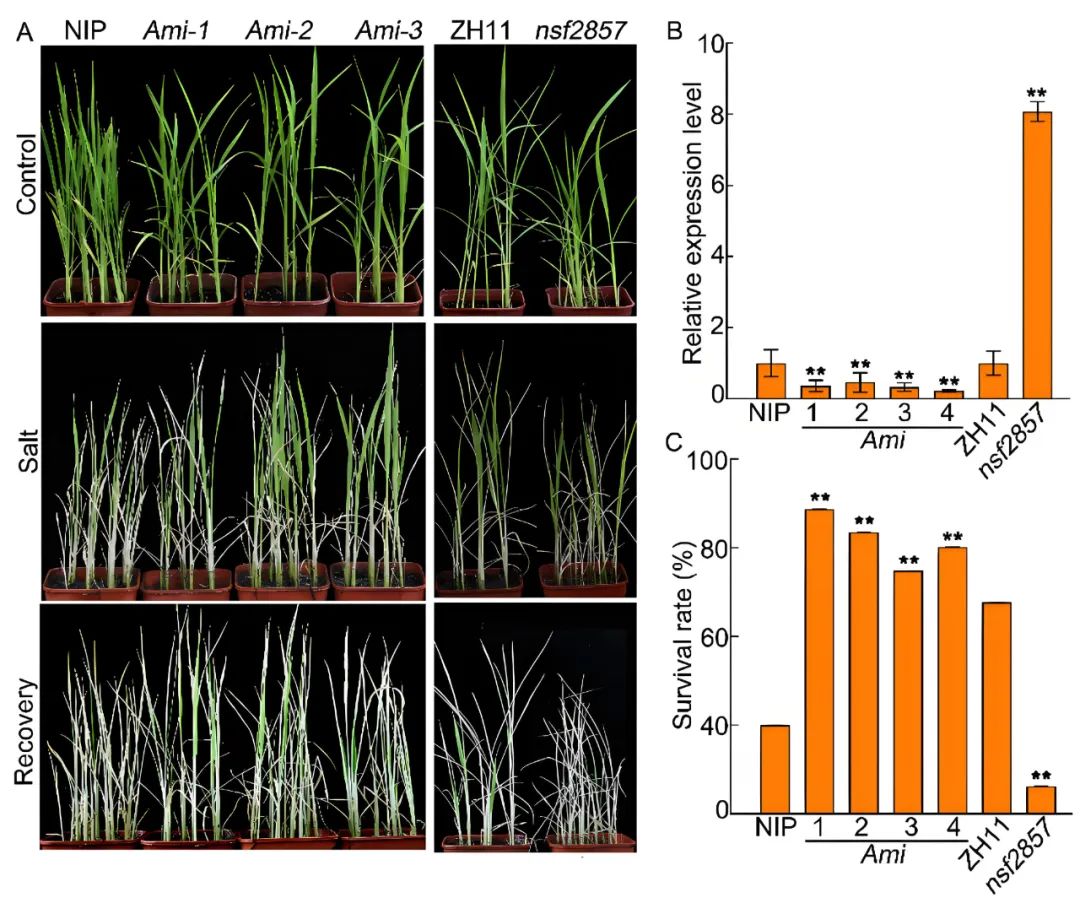
图7 OsERF2能够抑制水稻对盐分的耐受性(Zhou et al., 2025)。(A)野生型植株和OsERF2转基因植株在盐胁迫下的表型比较;(B)OsERF2在OsERF2转基因植株中的表达;(C)在恢复10天后,图(A)中所示植株的存活率。
关于amiRNA技术,大量的实验表明并非所有人工设计的amiRNA都能够有效地沉默目标基因。因此对于该技术的优化也一直不断,感兴趣的小伙伴可以去阅读“Artificial microRNA guide strand selection from duplexes with no mismatches shows a purine-rich preference for virus- and non-virus-based expression vectors in plants”和“Artificial microRNA guide strand selection from duplexes with no mismatches shows a purine-rich preference for virus- and non-virus-based expression vectors in plants”,这里就不再具体展开了。
由于篇幅的原因,本次的推文就给大家介绍到这里了,剩下的部分后续再为大家介绍,简单回顾一下本次推文的内容,主要为大家介绍了RNAi的发现与历史、RNAi的类型以及在沉默基因中的应用,该应用部分还有一个VIGS技术没有来得及讲,刚好与剩下的RNAi技术在作物病害防空中的应用技术HIGS、SIGS以及MIGS技术一起讲,好好比较一下它们之间的联系与区别。
References:
胡港,徐强.植物DNA从头甲基化的机制[J].科学通报,2021,66(15):1821-1834.
Allen E, Xie Z, Gustafson A M, et al. microRNA-directed phasing during trans-acting siRNA biogenesis in plants[J]. Cell, 2005, 121(2): 207-221.
Bartel D P. MicroRNAs: target recognition and regulatory functions[J]. Cell, 2009, 136(2): 215-233.
Baulcombe D. RNA silencing in plants[J]. Nature, 2004, 431(7006): 356-363.
Brosnan C A, Mitter N, Christie M, et al. Nuclear gene silencing directs reception of long-distance mRNA silencing in Arabidopsis[J]. Proceedings of the National Academy of Sciences, 2007, 104(37): 14741-14746.
Chen H M, Chen L T, Patel K, et al. 22-Nucleotide RNAs trigger secondary siRNA biogenesis in plants[J]. Proceedings of the National Academy of Sciences, 2010, 107(3 15269-15274.4):
Covey S N, Al-Kaff N S, Lángara A, et al. Plants combat infection by gene silencing[J]. 1997.
Creasey K M, Zhai J, Borges F, et al. miRNAs trigger widespread epigenetically activated siRNAs from transposons in Arabidopsis[J]. Nature, 2014, 508(7496): 411-415.
Cuperus J T, Carbonell A, Fahlgren N, et al. Unique functionality of 22-nt miRNAs in triggering RDR6-dependent siRNA biogenesis from target transcripts in Arabidopsis[J]. Nature structural & molecular biology, 2010, 17(8): 997-1003.
de Alba A E M, Elvira-Matelot E, Vaucheret H. Gene silencing in plants: a diversity of pathways[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2013, 1829(12): 1300-1308.
Ding S W, Voinnet O. Antiviral immunity directed by small RNAs[J]. Cell, 2007, 130(3): 413-426.
Eamens A L, Smith N A, Curtin S J, et al. The Arabidopsis thaliana double-stranded RNA binding protein DRB1 directs guide strand selection from microRNA duplexes[J]. RNA, 2009, 15(12): 2219-2235.
Eamens A, Wang M B, Smith N A, et al. RNA silencing in plants: yesterday, today, and tomorrow[J]. Plant physiology, 2008, 147(2): 456-468.
Fei Q, Xia R, Meyers B C. Phased, secondary, small interfering RNAs in posttranscriptional regulatory networks[J]. The Plant Cell, 2013, 25(7): 2400-2415.
Feng S, Jacobsen S E, Reik W. Epigenetic reprogramming in plant and animal development[J]. Science, 2010, 330(6004): 622-627.
Haag J R, Pikaard C S. Multisubunit RNA polymerases IV and V: purveyors of non-coding RNA for plant gene silencing[J]. Nature reviews Molecular cell biology, 2011, 12(8): 483-492.
Johnson C, Kasprzewska A, Tennessen K, et al. Clusters and superclusters of phased small RNAs in the developing inflorescence of rice[J]. Genome research, 2009, 19(8): 1429-1440.
Kurihara Y, Takashi Y, Watanabe Y. The interaction between DCL1 and HYL1 is important for efficient and precise processing of pri-miRNA in plant microRNA biogenesis[J]. RNA, 2006, 12(2): 206-212.
Kurihara Y, Watanabe Y. Arabidopsis micro-RNA biogenesis through Dicer-like 1 protein functions[J]. Proceedings of the National Academy of Sciences, 2004, 101(34): 12753-12758.
Li C, Wu J, Fu S, et al. Development of a transgenic rice line with strong and broad resistance against four devastating rice viruses through expressing a single hairpin RNA construct[J]. Plant Biotechnology Journal, 2024, 22(8): 2142.
Li J, Yang Z, Yu B, et al. Methylation protects miRNAs and siRNAs from a 3’-end uridylation activity in Arabidopsis[J]. Current biology, 2005, 15(16): 1501-1507.
Lindbo J A, Silva-Rosales L, Proebsting W M, et al. Induction of a highly specific antiviral state in transgenic plants: implications for regulation of gene expression and virus resistance[J]. The Plant Cell, 1993, 5(12): 1749-1759.
Matzke M A, Kanno T, Matzke A J M. RNA-directed DNA methylation: the evolution of a complex epigenetic pathway in flowering plants[J]. Annual review of plant biology, 2015, 66(1): 243-267.
Melnyk C W, Molnar A, Baulcombe D C. Intercellular and systemic movement of RNA silencing signals[J]. The EMBO journal, 2011, 30(17): 3553-3563.
Mette M F, Aufsatz W, Van der Winden J, et al. Transcriptional silencing and promoter methylation triggered by double‐stranded RNA[J]. The EMBO journal, 2000.
Millar A A, Waterhouse P M. Plant and animal microRNAs: similarities and differences[J]. Functional & integrative genomics, 2005, 5: 129-135.
Mlotshwa S, Pruss G J, Peragine A, et al. DICER-LIKE2 plays a primary role in transitive silencing of transgenes in Arabidopsis[J]. PloS one, 2008, 3(3): e1755.
Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans[J]. The plant cell, 1990, 2(4): 279-289.
Peragine A, Yoshikawa M, Wu G, et al. SGS3 and SGS2/SDE1/RDR6 are required for juvenile development and the production of trans-acting siRNAs in Arabidopsis[J]. Genes & development, 2004, 18(19): 2368-2379.
Ratcliff F, Harrison B D, Baulcombe D C. A similarity between viral defense and gene silencing in plants[J]. Science, 1997, 276(5318): 1558-1560.
Schwab R, Ossowski S, Riester M, et al. Highly specific gene silencing by artificial microRNAs in Arabidopsis[J]. The Plant Cell, 2006, 18(5): 1121-1133.
Smith N A, Singh S P, Wang M B, et al. Total silencing by intron-spliced hairpin RNAs[J]. Nature, 2000, 407(6802): 319-320.
Van der Krol A R, Mur L A, Beld M, et al. Flavonoid genes in petunia: addition of a limited number of gene copies may lead to a suppression of gene expression[J]. The Plant Cell, 1990, 2(4): 291-299.
Vaucheret H. MicroRNA-dependent trans-acting siRNA production[J]. Science's STKE, 2005, 2005(300): pe43-pe43.
Vazquez F, Vaucheret H, Rajagopalan R, et al. Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs[J]. Molecular cell, 2004, 16(1): 69-79.
Vermeersch L, De Winne N, Nolf J, et al. Transitive RNA silencing signals induce cytosine methylation of a transgenic but not an endogenous target[J]. The Plant Journal, 2013, 74(5): 867-879.
Wang J, Mei J, Ren G. Plant microRNAs: biogenesis, homeostasis, and degradation[J]. Frontiers in plant science, 2019, 10: 360.
Wang M B, Masuta C, Smith N A, et al. RNA silencing and plant viral diseases[J]. Molecular plant-microbe interactions, 2012, 25(10): 1275-1285.
Wang M B, Waterhouse P M. High-efficiency silencing of a β-glucuronidase gene in rice is correlated with repetitive transgene structure but is independent of DNA methylation[J]. Plant molecular biology, 2000, 43: 67-82.
Waterhouse P M, Graham M W, Wang M B. Virus resistance and gene silencing in plants can be induced by simultaneous expression of sense and antisense RNA[J]. Proceedings of the National Academy of Sciences, 1998, 95(23): 13959-13964.
Waterhouse P M, Wang M B, Lough T. Gene silencing as an adaptive defence against viruses[J]. Nature, 2001, 411(6839): 834-842.
Watson J M, Fusaro A F, Wang M B, et al. RNA silencing platforms in plants[J]. FEBS letters, 2005, 579(26): 5982-5987.
Wingard, S. A. Hosts and symptoms of ring spot, a virus disease of plants. [J]. Agric. Res. 37, 127–153(1928).
Ye R, Wang W, Iki T, et al. Cytoplasmic assembly and selective nuclear import of Arabidopsis Argonaute4/siRNA complexes[J]. Molecular cell, 2012, 46(6): 859-870.
Zhai J, Jeong D H, De Paoli E, et al. MicroRNAs as master regulators of the plant NB-LRR defense gene family via the production of phased, trans-acting siRNAs[J]. Genes & development, 2011, 25(23): 2540-2553.
Zhang H, He X, Zhu J K. RNA-directed DNA methylation in plants: Where to start?[J]. RNA biology, 2013, 10(10): 1593-1596.
Zhao J H, Guo H S. RNA silencing: From discovery and elucidation to application and perspectives[J]. Journal of integrative plant biology, 2022, 64(2): 476-498.
Zhou J, Fang S, Liu X, et al. OsERF2 Acts as a Direct Downstream Target of OsEIL1 to Negatively Regulate Salt Tolerance in Rice[J]. Rice, 2025, 18(1): 29.























 83
83

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








