前言
ESM3/ESMC模型仓库:
ESM3模型其实在今年年初就已经开源了,但是官方称ESM3主要是“专注于在医疗与其他应用方面的蛋白质的可控生成”,并且主要训练方法是mask prediction,所以用它来做序列嵌入生成的文章是比较少的(应该是)。
但今年的12月4号,ESMC模型开源了,并且开放了线上调用的接口,官方称ESMC是“专注于获取蛋白质隐藏生物信息的表征”的,是ESM2的上位替代,还列出了ESMC-600M可以上位替代ESM2-3B,表现和效率都优于ESM2,这下就不得不体验一把了。
替代表(官方认为的):
| ESMC | ESM2 | ESMC表现 |
| 300M | 650M | 相近 |
| 600M | 3B/15B | 上位替代 接近ESM2-15B |
| 6B | - | 远超ESM2 |
官方开源了ESM3、ESMC的代码,还开源了ESMC-300M和600M的参数,可以本地部署。
但既然有目前最优的蛋白质序列表征模型ESMC-6B,那肯定还是得试一试效果的。
ESMC-6B模型API的使用
首先pip install esm
然后官方给出了调用的代码:
from esm.sdk.forge import ESM3ForgeInferenceClient
from esm.sdk.api import ESMProtein, LogitsConfig
# Apply for forge access and get an access token
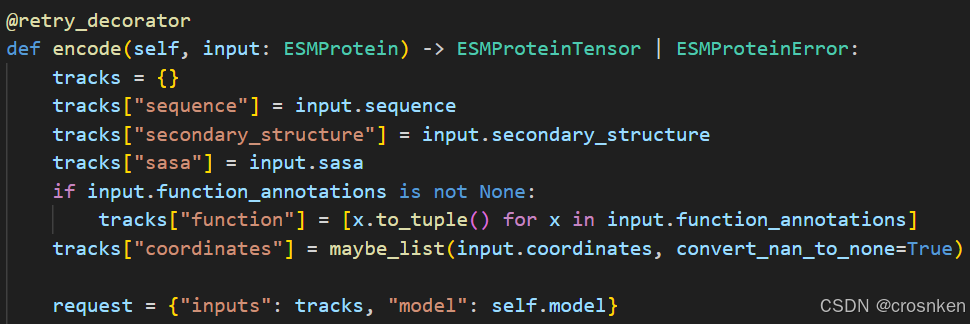
forge_client = ESM3ForgeInferenceClient(model="esmc-6b-2024-12", url="https://forge.evolutionaryscale.ai", token="<your forge token>")
protein_tensor = forge_client.encode(protein)
logits_output = forge_client.logits(
protein_tensor, LogitsConfig(sequence=True, return_embeddings=True)
)
print(logits_output.logits, logits_output.embeddings)但是这个代码需要获取到ESMC的使用token权限,需要申请,而且有点麻烦
https://forge.evolutionaryscale.ai.
有幸拿到访问许可之后就可以体验了。
ESMC-6B通过调用logits输出的embedding是一个 [1,序列长度,2560] 的tensor
一条序列嵌入所需的时间约为4~5s
如果你只需要跑某个物种的蛋白质数据集可以直接一条一条的查询,大概只用1~3天就能跑完
但如果要跑swissprot数据集(50w+),那可能需要跑上一个月,更别说还要对序列做mask数据增强。
并且输出的所占的空间很大,一般来说需要做一个平均池化或者什么池化
所以我就开始尝试能够一次性跑完一组序列的方法。
漫长的issue、源码、文档阅读
Issue1
首先是看到了这个issue
About Generating Protein Sequence Embeddings with Your Model · Issue #2 · evolutionaryscale/esm

这是开发者提出的一种方法
其实并没有解决batch预测的问题,只是利用了一下python语法的特性

本质上就是把多个序列拼接成一个,然后去跑模型
并且在调用encode API得到的序列依然只有一个<cls>和一个<eos>
没有根据序列来做tokenize
更加困惑的是,我们并不知道,如果将序列编码成
<cls>sequence1<eos><cls>sequence2<eos>...
这样的形势之后,ESMC的输出是否会受到不同序列间的数据特征的影响
毕竟它也是基于transformer的模型,n^2计算的自注意力层或多或少会导致不同序列间的影响。
(不过也可以试试看,后文我将API的encode()编码逆向出来了)
Issue2
然后就看到了这个Issue:make a batch to process multi protein at one time. · Issue #19 · evolutionaryscale/esm

虽然这个是针对ESM3生成式模型提出的一种batch方法
当时我也抱着死马当做活马医的态度试了一下
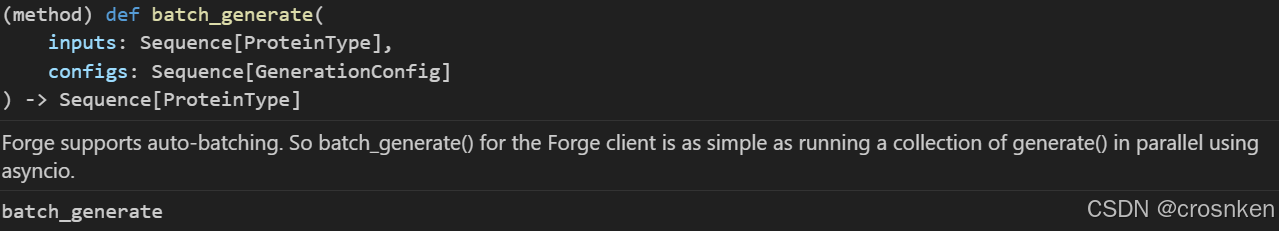
但是是过不了encode()的,因为encode()里面传入的内容只能是ESMProtein,不能使用其他的数据结构。
而batch_generate()输出是Sequence[ProteinType]
源码与文档分析
成功获取token后,可以看到reference文档,但是里面只写了http传输的数据包格式,并且再一次让我确定了,encode()和logits()都只能接受一个蛋白质序列。
但是根据源码,我们可以看出它是使用json传输request数据的,request里面就包含了我们要传过去的序列数据



源码中可以看出encode的输入格式已经被限制死了,ESMProtein中的seq数据只能是str
但是logits中传入的ESMProteinTensor还可以改变其中sequence的tensor形状
于是我决定尝试修改传入的protein_tensor.sequence的形状:
from esm.sdk.forge import ESM3ForgeInferenceClient
from esm.sdk.api import ESMProtein, LogitsConfig
import torch
protein = ESMProtein(sequence='ACDEFGHIKLMNPQRSTVWY_')
forge_client = ESM3ForgeInferenceClient(model="esmc-6b-2024-12", url="https://forge.evolutionaryscale.ai", token="")
protein_tensor = forge_client.encode(protein)
print(protein_tensor.sequence.shape)
protein_tensor.sequence = torch.stack([protein_tensor.sequence])
print(protein_tensor.sequence.shape)
logits_output = forge_client.logits(
protein_tensor, LogitsConfig(sequence=True, return_embeddings=True)
)
print(logits_output)
print(logits_output.logits, logits_output.embeddings.shape)但很不幸的是报错了,这次并不是encode()或者logits()的报错,而是网页端返回的报错,我们绕过了本地的所有数据类型限制,但是网页并不允许这样的tensor输入,以下是报错信息:

具体报错信息解释就是input.sequence[0]必须是一个合法的整数,而不是tensor
而我们改变形状之后,input.sequence[0]是第一个序列对应的tensor
于是
再看了一下ESMC模型的代码,发现模型主体部分是完全支持[....,.....,序列长度]的tensor输入的



logits函数调用的就是ESMC的forward
模型里面也没有多少玄学的操作,就一个Embedding、Transformer、regression
embedding不用说了,TransformerStack和RegressionHead(线性层+GELU+LN+线性)都是可以接入多维输入的


按道理来说使用ESMC模型端处理多维数据是没有任何问题的
这里就排除了ESMC模型端的报错
既然不是本地调用的问题,也不是远端模型的问题,最有可能的是服务器http解包的时候判断了读入数据的格式,避免了用户传入batch数据。
以上也只是我的猜想,但毕竟再怎么说server端代码肯定是不会开源的。
优化思路
尝试减小http包优化网络延迟
虽然说ESMC支持返回值采样forward_and_sample(),可以设置samplingconfig,直接获取蛋白质的平均值池化,显著地减小了http传输的数据量,但是这个功能在ESMC-6B模型中并不存在!以下是尝试使用的报错:

真是堵住了所有的路啊。。。。
尝试优化encode()减少http传输次数
最终成功的就只有这个了,但也优化不了多少,5秒一个序列查询优化到4.7秒左右
原理是通过检查encode()调用后返回的http报文,可以发现是有规律的
其实ESMC-6B远端模型使用的encode()和EsmSequenceTokenizer.tokenize()只有“_”的编码不同(encode是32,tokenize是3),其他的起始符<cls>:0, 占位符<pad>:1, 结尾符<eos>:2
all_amino_acid_number = {'A':5, 'C':23,'D':13,'E':9, 'F':18,
'G':6, 'H':21,'I':12,'K':15,'L':4,
'M':20,'N':17,'P':14,'Q':16,'R':10,
'S':8, 'T':11,'V':7, 'W':22,'Y':19,
'_':32}
def esm_encoder_seq(seq, pad_len):
s = [all_amino_acid_number[x] for x in seq]
while len(s)<pad_len:
s.append(1)
s.insert(0,0)
s.append(2)
return torch.tensor(s)这样就在本地进行encode(),减少一次http的RTT时间
但由于传输的数据量本来就很少,这个也优化不了多少时间,logits占的数据量才是最大的,结果它又不让优化。
结尾吐槽
拼尽全力,依然无法战胜
没能让ESM团队用出全力,真是遗憾呢
同学,你也不想reviewer知道你2025年还在用ESM-1b吧(其实还挺好用的)
但其实大家也不必着急(跑一个月也不是不能接受(?,毕竟一劳永逸,特征传三代,人走数据在),ESM团队正在开发ESMC-6B的batch输入功能了
在esm.utils.sampling中,有个类叫_BatchedESMProteinTensor,这个应该是以后会使用的数据传输格式,但是现在直接使用依然会报错,需要等团队修改server端代码的检查才能使用。
另外,本地也可以部署ESMC-600M的模型,而且是ESM2-3B的上位替代,ESMC-6B只是一个锦上添花的模型啦。


























 4517
4517

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








