转载自:
SMILES标准化方法以及其中的一个坑(手性)-CSDN博客
rdkit.Chem.MolToSmiles()方法是用于将RDKit分子对象转换为SMILES字符串的方法。它的参数如下:
mol:必需,要转换为SMILES字符串的RDKit分子对象。
isomericSmiles:bool类型,是否生成同分异构体SMILES,默认为False,即不生成同分异构体SMILES。
kekuleSmiles:bool类型,是否生成Kekule SMILES,默认为False,即生成熔合环上的芳香性保持未指定的杂化,如果是True,则生成使用Kekule表示法的SMILES。
canonical:bool类型,是否生成规范化的SMILES,默认为True,即生成规范化的SMILES。
allBondsExplicit:bool类型,是否为所有化学键添加显式的方向性和序数,即无论它们是单键、双键还是三键。默认为False。
allHsExplicit:bool类型,是否为所有氢原子添加显式的表示,默认为False。
sanitize:bool类型,是否在生成SMILES之前对分子进行净化,默认为True。净化操作将包括在分子中添加氢原子、移除未配对的电荷、设置正确的杂化和检查化学键长度等。
其中重要的一个参数:“isomericSmiles”,它表示是否保存原始分子的手性,当时做分子生成的时候坑惨了,我说怎么生成的分子都是不带有手性的呢???
from rdkit import Chem
# 创建一个 SMILES 字符串
smiles = 'O=C(N[C@@H](C)C1=CC=C(C(O)=O)C=C1)C2=C(CC3=CC=C(OC(F)F)C(OC(F)F)=C3)SC4=C2CCOC4'
# 将 SMILES 字符串转换为 RDKit 分子对象
mol = Chem.MolFromSmiles(smiles)
# 输出标准化后的 不带有手性SMILES
print(Chem.MolToSmiles(mol, isomericSmiles=False, canonical=True))
# 输出标准化后的 带有手性SMILES
print(Chem.MolToSmiles(mol, isomericSmiles=True, canonical=True))结果分析:
所以一定要保留手性:isomericSmiles = True
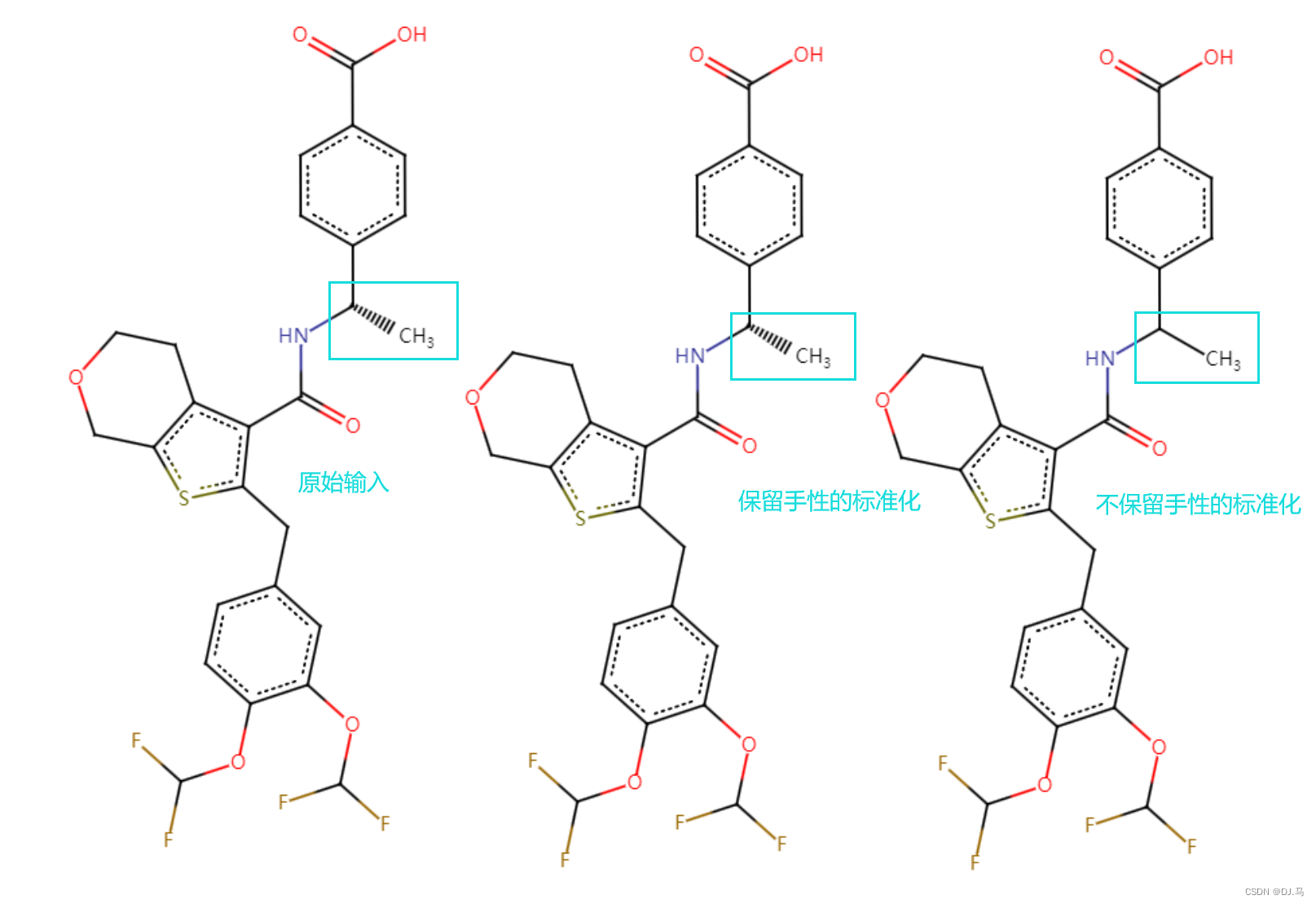
备注:手性
手性不会影响基本性质,例如MG,LogP....
手性中心个数(chiral_centers):过多的手性中心,会导致合成与纯化工艺难度的大幅提高。【chiral_center = len(Chem.FindMolChiralCenters(m, includeUnassigned=True))】
且会影响湿实验的结果,
我原来一直以为手性无关紧要,其实很有必要!!
"C@@H"和"C@H"区别以及和rdkit的标准化方法
"C@@H"和"C@H"这两种手性也是不一样的,以下两幅图的手性也是不同的,他们对应的化学反应可能也是不同的,
需要注意的是:带有"C@@H"的SMILES不一定就是第一幅图的实体黑色手性,它也有可能是第二附图的虚线手性,仅仅通过SMILES中的"C@@H"和"C@H"是无法判断手性是实体黑色还是虚线黑色,且带有"C@@H"的SMIELS和带有"C@H"的SMIELS可能是同一种物质【它们没有任何不同,也就说明rdkit的标准化方法可以很好的保留手性】:
标准化之前:O=C(N[C@@H](C)C1=CC=C(C(O)=O)C=C1)C2=C(CC3=CC=C(OC(F)F)C(OC(F)F)=C3)SC4=C2CCOC4
标准化之后:C[C@H](NC(=O)c1c(Cc2ccc(OC(F)F)c(OC(F)F)c2)sc2c1CCOC2)c1ccc(C(=O)O)cc1
























 8222
8222

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








