一、R语言中使用#来注释
#后面的内容都是注释掉的内容,不会执行。
二、R语言的安装包与加载包
1、R语言包的安装方法:从官网CRAN中下载,或自己导入(比如从官网下载到桌面再从桌面读取进来),一般使用第一种方法就够了。

2、直接输入包名,安装即可。

3、除了以上图形界面安装包方法外,还可以使用代码的方式安装R语言包。比如install.packages("tidyverse")。
install.packages("包名")4、安装完包后,每次重新打开R都需要library一下这个包,表示安装好后需要把这个包搬到R语言里头,就好比上战场打仗口袋里面装了很多子弹,但是我现在需要把它装在枪的弹夹里头。
三、了解TCGA数据库
1、TCGA数据库的建立和发展
TCGA数据库是由美国国家癌症研究所(NCI)和美国国家人类基因组研究所(NHGRI)于2005年联合启动的一项大规模癌症基因组研究项目,目的是通过全面、系统地研究癌症的基因组,揭示癌症的分子机制,以此推动癌症的早期诊断和个性化治疗的发展。
2、TCGA数据库的数据内容
TCGA数据库收录了来自33种主要癌症的约1.1万名患者的全基因组测序数据、蛋白质组数据、甲基化数据、mRNA数据、miRNA数据等多维度的癌症相关数据。其中,全基因组测序数据可以提供关于癌症患者基因的全面信息,蛋白质组数据可以揭示癌症细胞中的蛋白质表达和活性,甲基化数据可以反映癌症细胞的表观遗传状态,mRNA数据和miRNA数据则可以揭示癌症细胞的转录水平和调控机制。
3、TCGA数据库的应用
TCGA数据库的存在,可以帮助研究人员更好地理解癌症的遗传学和分子机制,为癌症的早期诊断、治疗策略的制定和药物的研发提供重要的数据支持。例如,研究人员可以通过分析TCGA数据库中的数据,发现癌症中常见的基因突变、拷贝数变异、结构变异等遗传变异,了解这些变异在癌症中的分布特征,这对于揭示癌症的遗传学机制具有重要价值。此外,通过分析TCGA数据库中的数据,研究人员还可以发现癌症的新靶点,为药物的研发提供方向。
4、TCGA数据库的挑战和前景
虽然TCGA数据库为癌症研究提供了丰富的数据资源,但是,如何有效地利用这些数据,挖掘出有价值的信息,仍然是一个挑战。这需要研究人员具有深厚的生物学知识、熟练的数据分析技能以及敏锐的科研洞察力。此外,随着科学技术的发展,TCGA数据库的数据内容和数据量都将进一步增加,如何管理和分析这些大规模的数据,也将成为未来的一个重要课题。然而,正是因为这些挑战,TCGA数据库的前景才更加广阔,它将继续为癌症研究和临床治疗提供重要的支持,推动癌症医学的发展。
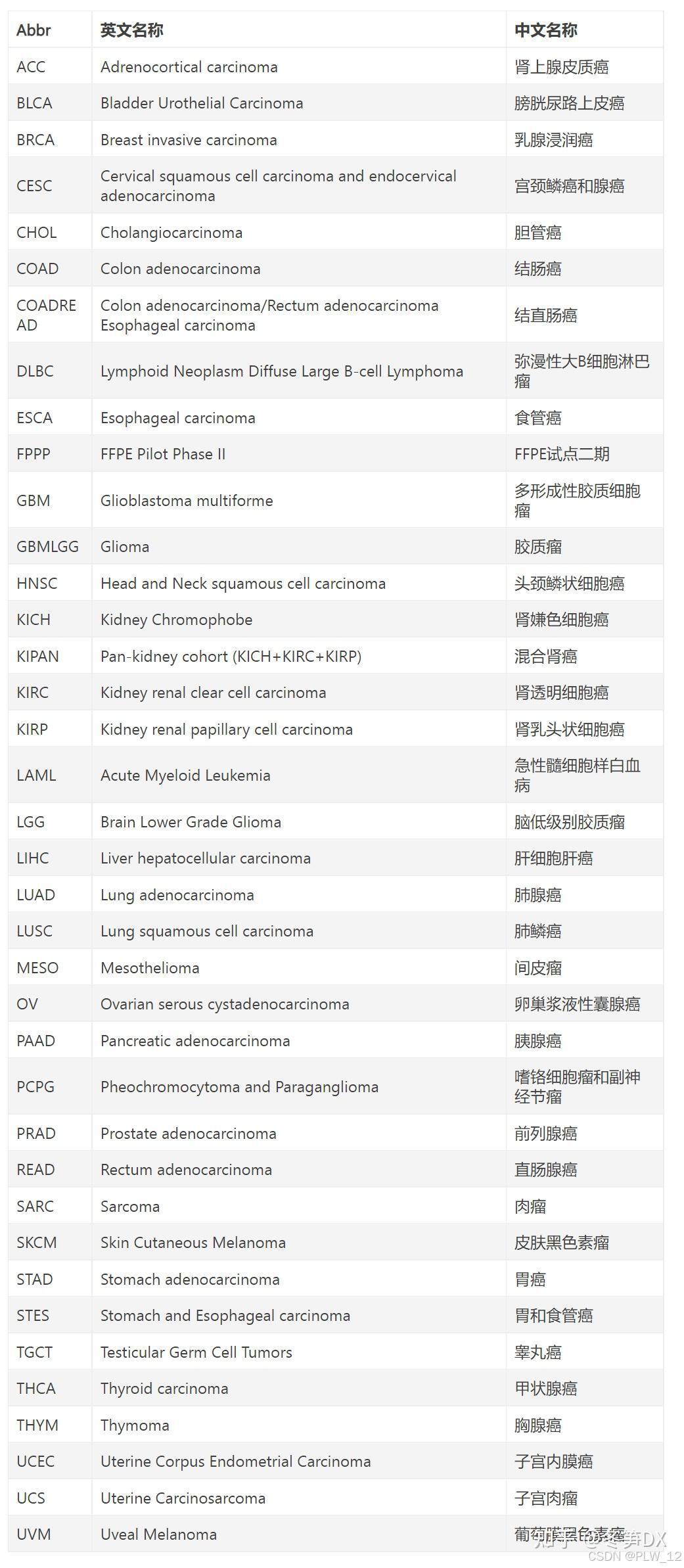
四、TCGA数据库中35 种癌种对应的中英文名称以及缩写
(图片来自知乎冬笋DX)
五、Xena是什么?
Xena是一个在线网站,由美国加利福尼亚大学开发,主要用于探索TCGA(The Cancer Genome Atlas)和其他公共数据库的数据。Xena提供了一个直观的平台,使用户能够快速下载特定基因在特定癌症中的表达量数据,并进行可视化分析。
六、在Xena网站下载TCGA对应癌种的数据库
1、Xena网站:https://xenabrowser.net/datapages/
2、在TCGA数据库中找到你所需要的癌症缩写,比如肝癌(LIHC),打开后可下载RNAseq中的Counts数据。



3、点击下载网址即可,就是速度比较慢,需要等待。






















 1067
1067

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








