图像分割相关的工作在实际项目中我们做得并不多,最近看到一篇高效的医学图像领域内的分割网络,觉得蛮有意思的就想趁着晚上忙完以后整体读一下,本文主要是自己论文阅读的记录,感兴趣的话可以参考一下,如果想要进一步了解研究内容详情的话可以移步阅读原英文论文,地址在这里,如下所示:

摘要
医学图像分割对于疾病诊断和监测至关重要。尽管当前的分割网络如UNet效果显著,但在捕捉长程特征方面存在困难。更准确的模型如TransUNet、Swin-UNet和CS-UNet具有更高的计算复杂度。为了解决这个问题,我们提出了GCtx-UNet,一种轻量级的分割架构,能够在性能上优于或与最先进的方法相媲美,同时捕捉全局和局部图像特征。GCtx-UNet使用视觉变换器,结合全局上下文自注意力模块和局部自注意力,以建模长程和短程空间依赖性。GCtx-UNet在Synapse多器官腹部CT数据集、ACDC心脏MRI数据集和多个息肉分割数据集上进行了评估。在Dice相似系数(DSC)和Hausdorff距离(HD)指标上,GCtx-UNet优于基于CNN和Transformer的方法,在复杂和小型解剖结构的分割中取得了显著的提升。此外,GCtx-UNet在模型大小、计算负载和训练及推理速度方面比最先进的方法更加高效,使其成为临床应用的实用选择。
1 引言
自动医学图像分割在提供预防、诊断、进展监测和预后等各种疾病的有价值信息以及定量病理评估方面至关重要。U形深度神经网络,包括编码器、解码器和跳跃连接,是目前最广泛使用的医学图像分割方法。尽管U形网络在许多医学图像分割任务中取得了最先进的性能,但它仍然存在局限性。一个主要限制是编码器有效提取和整合长程和局部特征的能力。基于卷积神经网络(CNN)的方法如UNet [26] 和 UNet++ [35] 擅长捕捉局部特征,但在建模数据中的长程依赖性方面存在困难。而基于Transformer的方法如Swin-UNet [6] 可以建模长程像素关系,但在建模局部信息时缺乏空间归纳偏置,导致结果不尽如人意。
过去的研究探索了CNN-Transformer混合架构如TransUnet [8] 以捕捉全局和局部信息,但这些模型通常显著增加了参数数量。这反过来转化为更高的计算复杂度,可能限制了它们的实际应用。最近,Hatamizadeh等人 [13] 提出了一种全局上下文视觉变换器(GC-ViT),利用全局上下文自注意力模块并与局部自注意力结合,以有效且高效地建模长程和短程空间交互。GC-ViT在图像分类、目标检测和语义分割任务中取得了最先进的结果。
在本文中,我们介绍了GCtx-UNet,一种用于医学图像分割的UNet类分割网络。GCtx-UNet通过使用GC-ViT [13] 编码器和解码器以及跳跃连接,有效捕捉长程和短程语义特征。这种架构在提高性能的同时需要更少的模型参数,具有更高的推理速度和更低的计算复杂度。
我们在多个医学图像数据集上评估了GCtx-UNet的分割和运行时性能,包括Synapse、ACDC和多个息肉图像数据集。我们的实验结果表明,GCtx-UNet在性能上优于或与最先进的分割算法(包括基于CNN、Transformer和混合分割网络)相当。此外,GCtx-UNet具有最小的模型大小,并使用最少的训练时间和推理时间。此外,我们在ImageNet和MedNet(从公共来源收集的20万张医学图像集合)上预训练了GCtx-UNet。在领域内图像(即MedNet)上预训练的GCtx-UNet比在自然图像(即ImageNet)上预训练的GCtx-UNet取得了更好的准确性。
2 相关工作
基于CNN的方法广泛用于医学图像分割,并被认为是最重要的方法之一。卷积神经网络(CNN),特别是基于编码器-解码器的架构如UNet [26] 及其衍生版本,在医学图像分割中显示出卓越的效果。例如,Att UNet [25] 通过注意力门增强了分割效果,而UNet++ [35] 引入了替代的跳跃连接机制,嵌套且密集,在一定程度上缓解了UNet各层次之间的语义差距。这种修改相对于UNet显著提高了性能。然而,UNet++无法捕捉全尺度的语义特征。Huang等人 [15] 提出了UNet3+,通过结合来自不同尺度的低级细节和高级语义,最大化全尺度特征图的使用。基于CNN的方法在各种医学图像分割任务中得到了应用,如视网膜图像分割 [11] 和皮肤分割 [33],展示了其在实现和训练中的良好性能和实用性。基于ResNet架构的分割算法在医学图像分割中确立了其地位 [19]。例如,Res-UNet [32] 通过加权注意力机制增强了视网膜血管分割。

图1:GC-ViT [13] 中局部和全局注意力机制的示意图。局部注意力仅在局部窗口内的特征补丁上计算(左)。全局注意力机制从整个输入特征图中提取查询补丁,聚合来自所有窗口的信息。全局查询与局部键和值令牌交互,从而允许捕捉长程信息。
Transformer固有的自注意力机制(MSA)使它们能够执行全局相关性建模,从而有效地处理长程依赖性。利用这一能力,Transformer在自然语言处理和计算机视觉任务中取得了显著进展,因其卓越的全局建模能力。一些开创性研究引入了基于Transformer的架构用于医学图像分割。Cao等人 [6] 提出了Swin-UNet,将Swin Transformer [21] 集成到U形分割网络中进行多器官分割。Azad等人 [2] 提出了TransDeepLab用于皮肤病变分割,通过多样化的窗口策略增强了DeepLab。此外,Huang等人 [16] 提出了MISSFormer,利用不同尺度的全局信息进行心脏分割,而Azad等人 [3] 提出了TransCeption,改进了补丁合并模块以捕捉单阶段内的多尺度表示。在编码器侧结合卷积操作和Transformer,Transclaw UNet [7] 实现了详细的分割和长距离关系学习。UNETR [12] 采用序列到序列预测进行3D医学图像分割。这些发展突显了基于Transformer的方法对医学图像分割的变革性影响,为更广泛的采用和深度学习进步铺平了道路。
Swin Transformer [21] 引入了局部窗口自注意力以降低成本,使其随图像大小线性增长,使用移位窗口注意力捕捉跨窗口信息,并通过分层架构利用多分辨率信息。然而,移位窗口注意力由于覆盖面积小而难以捕捉长程信息,并且缺乏像ViT [9] 那样的归纳偏置。
GC-ViT [13] 是一种分层架构,类似于Swin Transformer,但使用全局窗口注意力而不是移位窗口注意力,以有效捕捉长程信息。GC-ViT还使用卷积层进行下采样,为网络提供缺失的局部偏置和跨通道交互等理想属性。GC-ViT有4个阶段,每个阶段由局部和全局多头自注意力(MSA)层的交替块组成。如图1所示,在每个阶段,全局查询令牌通过使用融合倒残差块计算,该块包含来自不同图像区域的全局上下文信息。虽然局部自注意力模块负责建模短程信息,但全局查询令牌在所有全局自注意力模块中共享,以与局部键和值表示交互。
3 在MedNet数据集上的预训练
大多数基于CNN和Transformer的分割模型在自然图像如ImageNet上进行预训练。然而,由于自然图像和医学图像模态之间的语义差距 [27, 1],这对医学图像分割并不理想。在这项工作中,我们在一个名为MedNet的大型医学图像数据集上预训练了GC-ViT [13] 模型,特别是GCVit xxTiny。MedNet包含从多个公共数据集 [30] 和Kaggle [22, 23, 24] 收集的超过20万张医学图像。
MedNet包含不同类型的显微图像,如X射线、计算机断层扫描(CT)、光学相干断层扫描(OCT)和MRI。MedNet中的图像分为65个类别。与Stuckner等人 [29] 和Airfou等人 [1] 的方法类似,MedNet数据集分为训练集和验证集,每个类别有100张图像在验证集中,结果是96.75%/3.25%的训练/验证分割。使用每个类别100张图像进行验证足以获得可靠的准确性指标,并防止训练过程中过拟合。尽管验证集是平衡的,但训练集显示出一些类别不平衡。有几个类别,每个类别包含不到总图像的0.12%。三个类别包含6.2%的图像。大多数类别有超过2000张图像,占训练集的1%到2%。MedNet包括来自各种模态的图像,如X射线、CT、OCT和MRI,并涵盖了广泛的医学疾病,如肾癌、宫颈癌、阿尔茨海默病、Covid-19、肺炎、肺结核、猴痘、乳腺癌和疟疾。
我们使用AdamW优化器 [18] 对GC-ViT xxTiny进行了100个epoch的训练,初始学习率为0.0001,权重衰减为0.05,并使用余弦衰减调度器。训练数据通过albumentations库进行了增强,包括随机对比度和亮度的变化、垂直和水平翻转、光度失真和添加噪声。
训练过程持续到验证分数没有改善,采用早期停止标准,耐心为10个epoch。性能使用top-1和top-5准确性指标进行评估。Top-1准确性衡量测试样本中正确标签为最高预测的比例,而top-5准确性衡量测试样本中正确标签出现在前五个预测中的比例。GC-ViT xxTiny模型的top-1准确性为82.3%,top-5准确性为98.2%。
4 GCtx-UNet架构
GCtx-UNet的核心组件是GC-ViT块,其局部和全局注意力机制如图1所示。如图2所示,每个GC-ViT块包括局部和全局多头自注意力(MSA)、多层感知器(MLP)、全局令牌生成器(GTG)和下采样层。GTG组件为计算添加全局上下文。局部MSA只能在局部窗口内查询补丁,而全局MSA可以在窗口内查询不同图像区域。在每个阶段,全局查询组件是预先计算的。该块还在下采样层中引入了基于CNN的模块,以包含归纳偏置,这是ViT和Swin Transformer中缺失的有用特征。

图2:一个GC-ViT块具有局部和全局注意力、全局令牌生成器和下采样层。
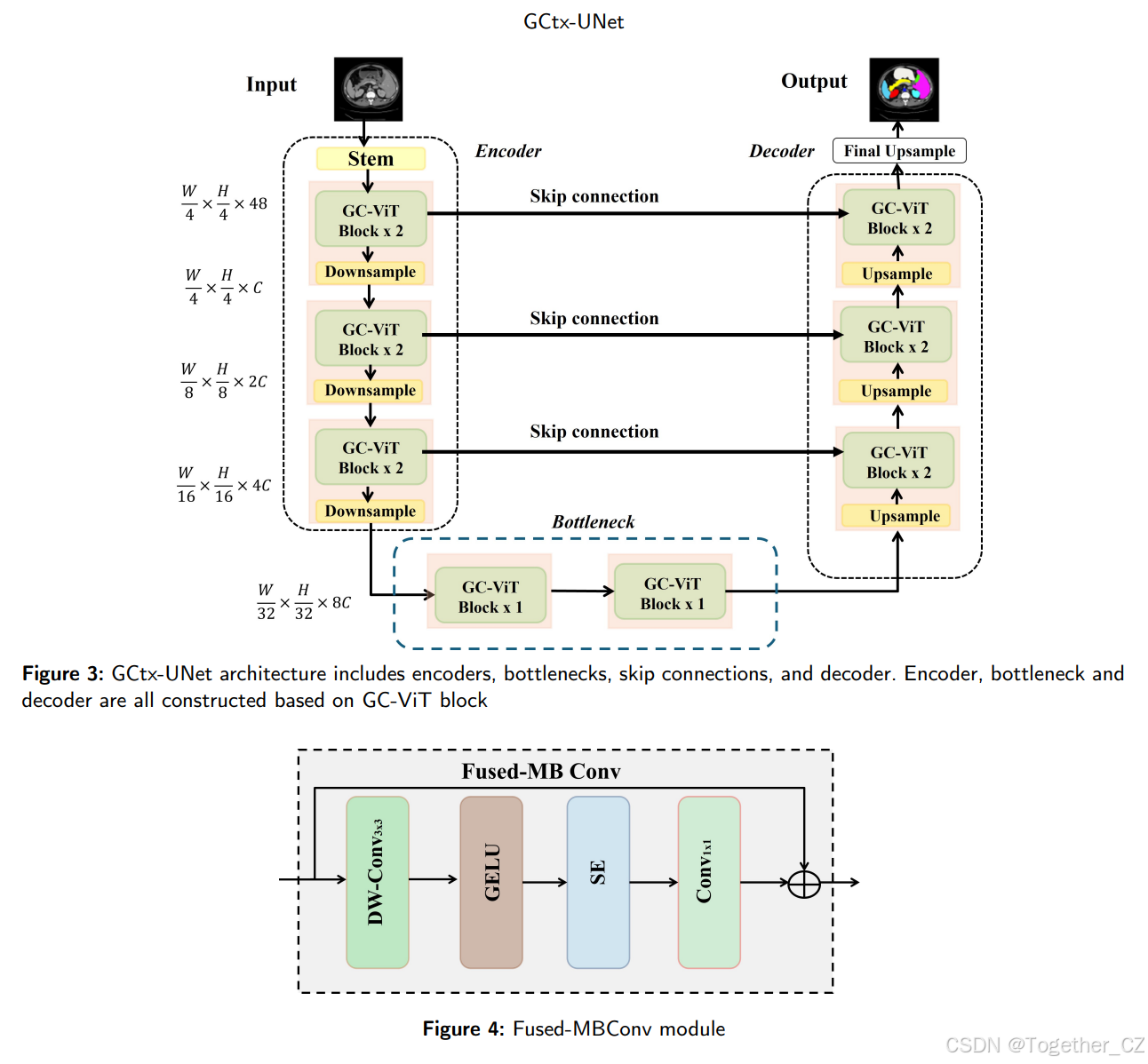
图3:GCtx-UNet架构包括编码器、瓶颈、跳跃连接和解码器。编码器、瓶颈和解码器均基于GC-ViT块构建。
图4:融合MBConv模块
GCtx-UNet是一种基于GC-ViT的U形编码器-解码器架构,具有跳跃连接,用于长程和短程语义特征学习。如图3所示,GCtx-UNet包括编码器、瓶颈、解码器和跳跃连接。
-
编码器和解码器都使用GC-ViT [13] 来建模长程和短程空间交互,无需计算注意力掩码或移动局部窗口等昂贵操作。
-
在每个阶段,GC-ViT编码器和解码器由交替的局部和全局自注意力模块组成,以提取空间特征。两者都在局部窗口内操作,类似于Swin Transformer。
-
跳跃连接将GC-ViT编码器的特征图与相应解码器阶段的特征图连接起来。瓶颈用于获取深度特征表示,在此组件中保持特征维度和分辨率不变。
-
编码器部分各阶段之间的下采样器和解码器部分各阶段之间的上采样器提供归纳偏置和建模通道间依赖性的理想属性。
在编码器中,初始图像被分割成四个补丁块,作为四阶段GC-ViT模块的输入。经过编码过程后,图像尺寸减小到(H/32)×(W/32)。在解码器中,上采样操作用于将图像尺寸增加2倍,并将通道数减少2倍。编码器各阶段的特征通过跳跃连接与解码器相应阶段的特征连接。解码器通过上采样完成其任务。
编码器
编码器采用分层GC-ViT方法,以在不同分辨率下获取特征表示。这是通过在4个阶段中减少空间维度并同时将嵌入维度增加2倍来实现的。首先,输入图像 x∈RH×W×3x∈RH×W×3 通过补丁化层进行处理。该层包括一个步幅为2的3×3卷积操作和填充,以生成重叠补丁。随后,这些补丁通过另一个3×3卷积层投影到维度为C的嵌入空间。在GC-ViT骨干的每个阶段之后,通过下采样层减少空间分辨率,同时增加通道数。这种下采样操作有助于在不同分辨率下提取分层特征。
下采样器
在下采样器中,我们加入了融合MBConv模块,通过注入归纳偏置和建模通道间相关性来生成分层表示。然后,使用核大小为3、步幅为2的卷积层将空间特征分辨率下采样2倍,同时将通道数加倍。如图4所示,融合MBConv包括DW-Conv3×3、GELU、SE和Conv1×1。融合MBConv操作可以通过以下方程定义:

其中,DW-Conv表示深度卷积,SE表示挤压和激励块,GELU表示高斯误差线性单元函数。
瓶颈
与Swin-UNet [6] 类似,瓶颈由两个GC-ViT块组成。瓶颈旨在促进深度特征表示的学习。在此结构中,特征维度和分辨率保持不变。
解码器
对称解码器与编码器相对应,使用GC-ViT变换器块构建。解码器镜像编码器设计,用未补丁块替换补丁块,用解嵌入层替换嵌入层,用上采样块替换下采样块。解码器的上采样块替换编码器的下采样块。上采样块将相邻维度的特征图重构为更高分辨率的特征图,并将特征维度减半。这种上采样块有效地增加了空间分辨率,同时细化和归一化特征表示,使其适用于解码和重建分割模型中的高分辨率特征。跳跃连接将编码器的特征与从上采样恢复的深度特征融合,从而减轻了下采样产生的空间数据损失。
表1: 我们实验中使用的方法列表

5 实验评估
我们将GCtx-UNet的性能与包括基于CNN、基于Transformer和混合算法在内的最先进算法进行了比较。表1总结了我们实验中使用的方法列表。我们在三种不同类型的医学图像数据集上评估了在MedNet和ImageNet上预训练的GCtx-UNet的性能,包括Synapse多器官分割数据集(Synapse)、自动化心脏诊断挑战数据集(ACDC) [5] 和息肉数据集,包括CVC-ClinicDB [4]、Kvasir-SEG [17]、CVC-300、ColonDB [31] 和ETIS-LaribDB [28]。
Synapse
Synapse多器官分割数据集(Synapse)包括30个病例的3779张腹部临床CT图像,其中18个病例用于训练,12个病例用于测试。该数据集包含8个腹部器官(主动脉、胆囊、左肾、右肾、肝脏、胰腺、脾脏和胃)。每个CT体积包括85~198张512×512像素的图像,体素空间分辨率为[0.54~0.54]×[0.98~0.98]×[2.5~5.0] mm [3]。
ACDC
自动化心脏诊断挑战数据集(ACDC) [5] 汇编了来自MICCAI 2017数据集的各种患者的MRI扫描结果。ACDC数据集包含100个心脏MRI扫描,每个扫描包含三个器官:右心室(RV)、心肌(Myo)和左心室(LV)。按照TransUNet [8] 的方法,我们将数据集分为70个训练病例、10个验证病例和20个测试病例。
息肉
我们使用了5个早期结直肠癌诊断图像的息肉数据集。CVC-ClinicDB [4] 和Kvasir-SEG [17] 数据集用于二值分割。CVC-ClinicDB数据集包含612张带有标记息肉的RGB结肠镜图像,像素分辨率为288×384。Kvasir-SEG数据集包含1000张息肉图像,像素分辨率范围为332×487到1920×1072,并附有相应的地面真值。按照PraNet [10] 的设置,我们使用了CVC-ClinicDB数据集中的900张图像和Kvasir数据集中的548张图像进行训练。剩余的64张CVC-ClinicDB图像和100张Kvasir图像用作测试集。为了评估模型的泛化性能,我们在三个未见过的数据集(CVC-300、CVC-ColonDB和ETIS-LaribDB)上测试了模型。
5.1 实现
GCtx-UNet使用PyTorch库实现,并在具有12GB内存的Nvidia GeForce GTX TITAN X上进行训练。输入图像大小被缩减为224×224像素。我们的模型使用批量大小24、学习率0.0001、AdamW优化器(动量0.9,权重衰减0.0001)进行训练。对于评估指标,我们使用了Synapse、ADCD和Polyp数据集的平均Dice相似系数(DSC)。此外,我们在Synapse数据集上使用了平均95% Hausdorff距离(HD)。HD指标提供了关于边界误差的更精确的性能估计。DSC值范围从0到1,较大的值表示更好的性能,而较小的HD值表示更好的性能。
表2: GCtx-UNet与最先进算法在Synapse上的比较(列分别为平均DSC(%)、平均HD(mm)和每个器官的DSC(%))。蓝色表示最佳结果,红色表示次佳结果。GCtx-UNet1在ImageNet上预训练,而GCtx-UNet2在MedNet上预训练。所有其他模型在ImageNet上预训练。

5.2 Synapse
我们将GCtx-UNet与当前最先进的方法在Synapse数据集上进行了比较,包括UNet、Att-UNet、TransUnet、SwinUnet、MISSFormer、TransDeepLab、HiFormer、GPA-TUNet和CS-UNet。表2总结了DSC和HD的性能比较。
在MedNet上预训练的GCtx-UNet在平均DSC(82.39%)和平均HD(15.94 mm)上排名第二,而在ImageNet上预训练的GCtx-UNet在平均DSC(81.95%)和平均HD(16.8 mm)上排名第四。请注意,这些指标与最佳平均DSC(83.27%)和平均HD(14.7 mm)相差不远,后者来自CS-UNet和HiFormer,它们具有更高的计算复杂度。还请注意,GCtx-UNet显著优于基于CNN的方法UNet和Att-UNet。例如,UNet的平均DSC为76.85%,平均HD为39.70 mm,而Att-UNet的平均DSC为77.77%,平均HD为36.02 mm。GCtx-UNet在性能上优于基于Transformer的方法,如Transdeeplab、MISSFormer和Swin-UNet,其平均DSC范围为79.13%到81.96%,平均HD范围为18.20到21.25 mm。GCtx-UNet的性能也与混合模型如TransUnet、GPA-TUNet、HiFormer和CS-UNet相当,其平均DSC范围为77.48%到83.27%,平均HD范围为14.70到31.69 mm。
器官级性能
图5显示了两个Synapse图像的定性比较,其中GCtx-UNet的性能优于Swin-UNet和CS-UNet。GCtx-UNet正确分割了大多数器官,但在胆囊区域有一些误分类。相比之下,Swin-UNet过度分割了脾脏(一些属于脾脏的区域被误分类为左肾),而CS-UNet过度分割了胰腺。我们推测这种改进是由于使用了GC-ViT,它引入了一个参数高效的下采样模块,并修改了融合MB-Conv块。这些修改解决了ViTs中缺乏归纳偏置的问题,使GCtx-UNet能够准确捕捉相对较大的区域,并在相邻器官的分割中表现良好。
具有清晰边界的大器官(如肾脏、胰腺和脾脏)的分割需要网络捕捉全局特征。我们推测这就是为什么基于Transformer的模型比基于CNN的模型更准确的原因。较小器官(如主动脉)的分割更多受益于局部特征的检测。这可能是为什么基于CNN的模型比基于Transformer的模型结果更准确的原因。具有复杂边界的大器官(如肝脏和胃)的分割需要捕捉局部和全局特征。这可能是为什么混合模型结果更准确的原因。
5.3 ACDC
我们将GCtx-UNet在ACDC数据集上的性能与一些最先进的方法进行了比较,包括基于CNN的方法(R50-UNet和R50-Atten-UNet)、基于Transformer的方法(R50-ViT、Swin-UNet、MISSFormer)和混合方法(TransUNet、GPA-TUNet和CS-UNet)。结果如表3所示。GCtx-UNet在平均DSC上优于所有其他方法,其中在MedNet上预训练的GCtx-UNet表现最好。特别是,GCtx-UNet在右心室(RV)和左心室(LV)上的表现更好。在MedNet上预训练的GCtx-UNet也比在ImageNet上预训练的GCtx-UNet表现更好。

图5:GCtx-UNet与地面真值、CS-UNet和Swin-UNet在Synapse数据集两个样本图像上的比较。请注意,GCtx-UNet1在ImageNet上预训练,而GCtx-UNet2在MedNet上预训练。红色矩形标识了Swin-UNet相对于GCtx-UNet和CS-UNet倾向于过度分割问题的区域。

图5:GCtx-UNet与地面真值、CS-UNet和Swin-UNet在Synapse数据集两个样本图像上的比较。请注意,GCtx-UNet1在ImageNet上预训练,而GCtx-UNet2在MedNet上预训练。红色矩形标识了Swin-UNet相对于GCtx-UNet和CS-UNet倾向于过度分割问题的区域。

图6:GCtx-UNet与地面真值和CS-UNet在ACDC数据集3个示例图像上的比较。请注意,GCtx-UNet1在ImageNet上预训练,而GCtx-UNet2在MedNet上预训练。橙色矩形框标识了GCtx-UNet1和CS-UNet相对于GCtx-UNet2存在过度或欠分割问题的区域。
表4: GCtx-UNet与最先进算法在息肉数据集上的比较(列分别为平均DSC(%))。蓝色表示最佳结果,红色表示次佳结果。GCtx-UNet1在ImageNet上预训练,而GCtx-UNet2在MedNet上预训练。

图6包括ACDC数据集中的3个示例图像,用于GCtx-UNet和CS-UNet的定性比较。在MedNet上预训练的GCtx-UNet能够比CS-UNet和在ImageNet上预训练的GCtx-UNet更准确地分割右心室和左心室。
息肉数据集
我们通过首先在两个已见数据集(CVC-ClinicDB和Kvasir)上训练GCtx-UNet,然后在三个未见数据集(CVC-ColonDB、ETIS-LaribDB和CVC-300)上使用训练好的模型来评估模型的泛化性能。表4比较了GCtx-UNet与最先进的基于CNN的算法(UNet、UNet++、PraNet)和混合方法(CS-UNet)的性能。尽管在MedNet上预训练的GCtx-UNet在Kvasir数据集上具有最佳的DSC指标,但在CVC-ClinicDB数据集上略逊于CS-UNet和PraNet,尽管差异相对较小。
泛化性
如表4(第3-5列)所示,GCtx-UNet在未见数据集(CVC-ColonDB、ETIS-LaribDB和CVC-300)上具有更好的泛化性。与其他方法相比,GCtx-UNet模型在CVC-ColonDB和ETIS-LaribDB数据集上具有最佳和次佳的DSC值,在CVC-300数据集上具有次佳的DSC值。GCtx-UNet的整体性能相当显著。
图7展示了各种方法的定性分割结果,包括在MedNet和ImageNet上训练的GCtx-UNet。选择了五个样本,每个数据集一个,以突出模糊边界和小息肉,便于区分分割性能的比较。在MedNet上预训练的GCtx-UNet显著减少了假阳性和假阴性。这种改进归因于其增强的区分息肉区域和正常区域模糊边界的能力。
6 计算复杂度
GCtx-UNet不仅具有良好的分割性能,而且比最先进的分割方法具有更低的计算复杂度。我们比较了GCtx-UNet与基于Transformer和混合方法的计算复杂度,包括模型参数数量、每训练epoch的浮点运算(FLOPs)、推理时间和模型大小。此评估在Synapse数据集上进行。如表5所示,GCtx-UNet的参数数量最少,为1234万,而参数数量最接近的模型TransDeepLab有2114万个参数。相应地,GCtx-UNet的模型大小最小,为49.75 MB,而第二小的模型TransDeepLab为86.343 MB。GCtx-UNet每训练epoch的FLOPs最少,为30.41G,而最接近的模型Swin-UNet为61.64G。同样,GCtx-UNet每epoch的训练时间最短,推理速度最高。

图7:GCtx-UNet与CS-UNet、PraNet、UNet和UNet++在息肉数据集5个示例图像上的比较。GCtx-UNet1在ImageNet上预训练,而GCtx-UNet2在MedNet上预训练。
表5: 基于模型参数、每训练epoch的浮点运算(FLOPs)、训练所需epoch数、每epoch训练时间(TTPE)、1568张轴向腹部临床CT图像的每秒帧数(FPS)推理时间和模型大小,比较GCtx-UNet、基于Transformer和混合算法。对于FLOPs和TTPE,批量大小为10。训练epoch数为最终结果所需的数量。

GCtx-UNet的低计算成本和小内存占用使其成为性能和模型复杂度之间最佳的权衡。它在性能上优于或与大多数最先进模型相当,而这些模型具有更大的模型大小和更高的计算成本。GCtx-UNet的高效设计突显了其在临床应用中实现更好分割结果的潜力。
7 消融研究
为了研究各种因素对模型性能的影响,我们在Synapse数据集上进行了消融研究。下面,我们讨论了上采样和训练模型的最佳超参数的影响。
表6:关于训练超参数对GCtx-UNet性能影响的消融研究。超参数包括损失函数(交叉熵损失和dice损失)和学习率。
表7:关于上采样类型对GCtx-UNet性能影响的消融研究

7.1 超参数调整
GCtx-UNet使用两种损失函数的组合进行训练,即dice损失和交叉熵损失,这与许多当前的分割方法一致。在训练过程中,我们通过选择dice和交叉熵损失的最佳组合以及最佳学习率来提高性能。我们进行了实验,以确定组合损失和学习率的最佳设置。表6比较了GCtx-UNet在各种超参数值下的Dice相似系数(DSC)和Hausdorff距离(HD)值的性能。当(dice损失,交叉熵损失)=(0.3,0.7)且学习率为0.0001时,达到了最佳的DSC和HD。此配置用于我们后续的所有实验。
7.2 上采样
与Swin-UNet [6] 类似,为了补充编码器中的下采样层,我们在解码器中专门设计了一个上采样层,以执行上采样和特征维度增加。为了评估此上采样层的有效性,我们在Synapse数据集上评估了GCtx-UNet,比较了使用双线性插值(带或不带SE(挤压和激励)块)的GCtx-UNet与使用转置卷积(带或不带SE块的融合MBConv模块)的上采样层的GCtx-UNet。表7中的结果表明,使用转置卷积(带SE块的融合MBConv模块)的上采样层的GCtx-UNet具有最佳性能。
8 结论
我们介绍了GCtx-UNet,一种U形网络,通过结合轻量级视觉变换器,有效捕捉全局和局部特征,从而增强医学图像分割。编码器和解码器组件之间的下采样和上采样块提供了归纳偏置并有效建模通道间依赖性。GCtx-UNet在各种医学图像数据集上的性能优于或与传统的基于CNN、基于Transformer和混合方法相当。同时,GCtx-UNet具有更低的模型复杂度,更少的模型参数、更小的模型大小、更短的训练和推理时间以及更低的训练FLOPs。GCtx-UNet建模长程空间依赖性的能力及其在分割复杂和小型解剖结构中的竞争性能使其成为临床应用的有前途工具。该架构的设计,包括带有跳跃连接的GC-ViT编码器和解码器,在保持比最先进方法更低的计算复杂度的同时,贡献了其高性能。在医学图像数据集MedNet上预训练并在多个医学成像任务上进行后续评估,展示了模型的鲁棒性和泛化能力,使GCtx-UNet成为医学图像分割的实用且强大的方法。作为未来的工作,我们计划引入GCtx-UNet 3D模型用于医学图像的体积分割。

























 3357
3357

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










