代码(基于Jupyter):https://github.com/williamdee1/LMPred_AMP_Prediction/tree/main
摘要
基于目前AMP是潜在的癌症治疗和高血压治疗的新药,同时新AMP的发现能够缓解耐药细菌问题,而常规的湿实验周期长、开销大,因此开发一种计算方法是解决这一问题的有效方式。本文作者提出一种新的AMP预测方法——LMPred,它由一个预训练好的语言模型(用于embedding)和CNN分类器(用于预测)组成,且在不同的数据集上取得了很高的预测精确度,同时优于目前最优秀的分类模型。
数据集
文中使用了两个数据集——Veltri dataset和LMPred dataset。Veltri dataset由1778个AMP和1778个non-AMP组成Antimicrobial Peptide Scanner;LMPred dataset由Veltri和Bhadra等人使用的数据集以及DRAMP 2.0 database组成,通过一定的筛选后最终获得3758AMPs。non-AMP则是通过使用前人文章中的以及UniProt database中的,通过筛选以及匹配AMP数据序列长度分布,最终也是得到3758个non-AMP。
模型
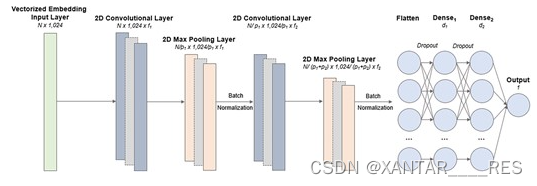
本文的LMPred方法结构分为两部分:pre-trained language model和CNN。
主要介绍一下language model(LM)。本文使用了三种在特定数据集上预训练好的语言模型——BERT、T5(Text-to-Text Transfer Transformer)和XLNet。它们分别在UniRef100、UniRef50和Big Fat Databse数据集上进行预训练,最终得到5组预训练模型:BERT、BERT BFD、T5XL_UNI、T5XL_BFD和XLNET。下面是对输入序列进行embedding操作的实例,

CNN分类器为每个LM设计了两种,但通过实验测试后发现两者最大差距仅1.47%。所有分类器中表现最好的模型为基于UniRef50预训练的T5,如下图所示,
总结
这篇文章提出了在AMP预测中一种新的序列输入预处理方法。通过使用预训练的LM模型embedding,最终使得分类任务优于目前已知的最优秀的方法。本文的结果也进一步的论证了NLP模型可以很好地应用到语言任务场景。










 文章介绍了LMPred,一种结合预训练语言模型(如BERT,T5,XLNet)和CNN进行抗微生物肽预测的方法。该方法在Veltridataset和LMPreddataset上表现优秀,优于现有最佳分类模型。预训练模型在不同数据集上训练,然后通过CNN进行分类,表明NLP技术在生物信息学领域的潜力。
文章介绍了LMPred,一种结合预训练语言模型(如BERT,T5,XLNet)和CNN进行抗微生物肽预测的方法。该方法在Veltridataset和LMPreddataset上表现优秀,优于现有最佳分类模型。预训练模型在不同数据集上训练,然后通过CNN进行分类,表明NLP技术在生物信息学领域的潜力。














 4万+
4万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








