文献链接:https://journals.asm.org/doi/10.1128/mSystems.00299-21
doi:10.1128/mSystems.00299-21
摘要
AMPs(Antimicrobial Peptides)潜在的抗菌特性近期受到了广大药物发现领域的学者的高度关注。然而,使用湿实验(wet-lab)筛选方法研发AMP的低效性和高花费是一个严峻的问题。因此,作者结合优秀的AMP数据集和深度学习开发了一种蛋白质编码方法,AI4AMP,它可以根据给定蛋白质序列预测AMP并且对蛋白质组进行筛选。
数据集
文章工作中使用了4个databases——APD3、LAMP、CAMP3和DRAMP。除去含有非常规氨基酸和长度短于10aa的,再筛选出重复的后,总共获得了6623个肽序列。对于non-AMP数据,其主要由真实世界中的肽(取自UniProt database)和人工合成的肽序列组成,最终也是获得了6623个肽序列。
实验方法
一、PC6编码方案
其核心是对每一个氨基酸的相关物理化学特性进行词嵌入(word embedding),得到一个矩阵,它每一行代表一个氨基酸的6个物理化学特征的值(经过归一化处理后的)。同时还使用6个值都为0的“X”做填充(padding)。最终得到一个200×6的矩阵(如下图A)。
补充说明,本文使用的6个化学特征分别为
- hydrophobicity(H1)
- the volume of side chains(V)
- Polarity(Pl)
- pH at the isoelectric point(pI)
- the dissociation constant for the COOH group(pKa)
- the net charge index of the side chain(NCI)
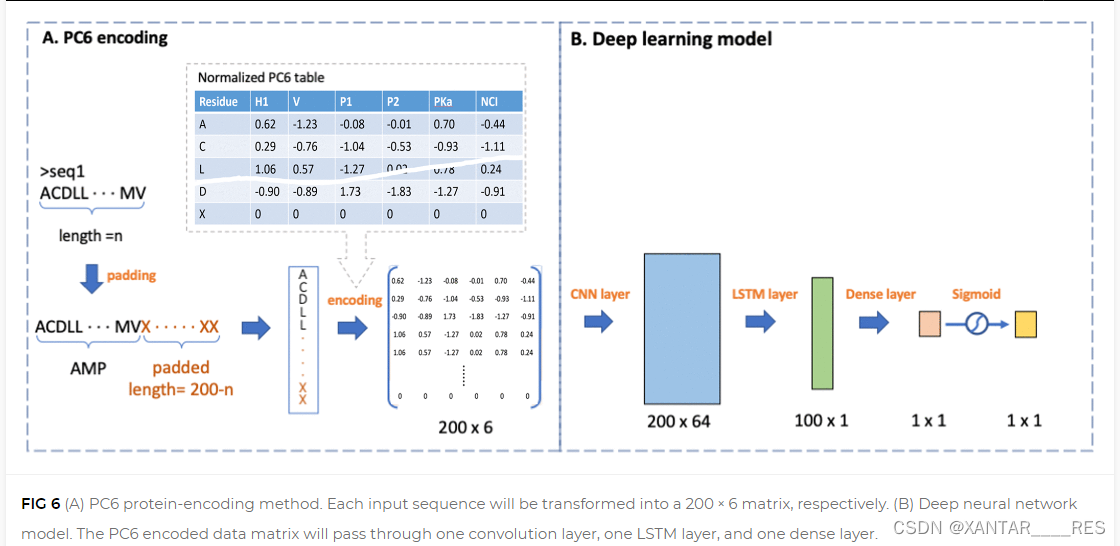
二、深度神经网络模型
本文的模型为NLP模型,由一层CNN、一层LSTM和一层全连接层组成(如上图B)。CNN层使用了64个包含16单元的过滤器(filter),然后使用矫正的线性激活函数接入LSTM。这里还使用Adam优化器,其学习率为0.0003。
三、模型评估
本文工作使用的评估指标为Accuracy、Precision、Sensitivity、Specificity、F1 score和MCC(Matthew correlation coefficient)。
























 445
445











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








