点击下方卡片,关注“CVer”公众号
AI/CV重磅干货,第一时间送达
添加微信号:CVer2233,小助手会拉你进群!
扫描下方二维码,加入CVer学术星球!可以获得最新顶会/顶刊上的论文idea和CV从入门到精通资料,及最前沿应用!发论文/搞科研/涨薪,强烈推荐!

3D医学图像分割方法已经取得了成功,但它们对大量体素级标注数据的依赖是一个需要解决的缺点,因为获取这些标注的成本很高。半监督学习(SSL)通过使用大量未标注数据和少量标注数据进行模型训练,解决了这一问题。最成功的SSL方法基于一致性学习,即通过最小化从扰动视图中获得的模型响应之间的距离来实现的。这些扰动通常在视图之间保持空间输入上下文的一致性,这可能导致模型从空间输入上下文中学习分割模式,而不是从前景对象中学习。在本文中,我们提出了TraCoCo,这是一种一致性学习SSL方法,它通过改变输入数据视图的空间输入上下文来进行扰动,使模型能够从前景对象中学习分割模式。此外,我们提出了一种新的“Confident Regional Cross entropy (CRC)”损失,该损失提高了训练收敛性,并保持了对共训练伪标签错误的鲁棒性。我们的方法在多个3D数据基准上达到了最先进的结果,例如左心房(LA)、胰腺CT(Pancreas)、和脑肿瘤分割(BraTS19)。我们的方法在一个2D切片基准——自动心脏诊断挑战(ACDC)上也取得了最佳结果,进一步证明了其有效性。

文章地址: https://ieeexplore.ieee.org/abstract/document/10695462
代码地址: https://github.com/yyliu01/TraCoCo
背景
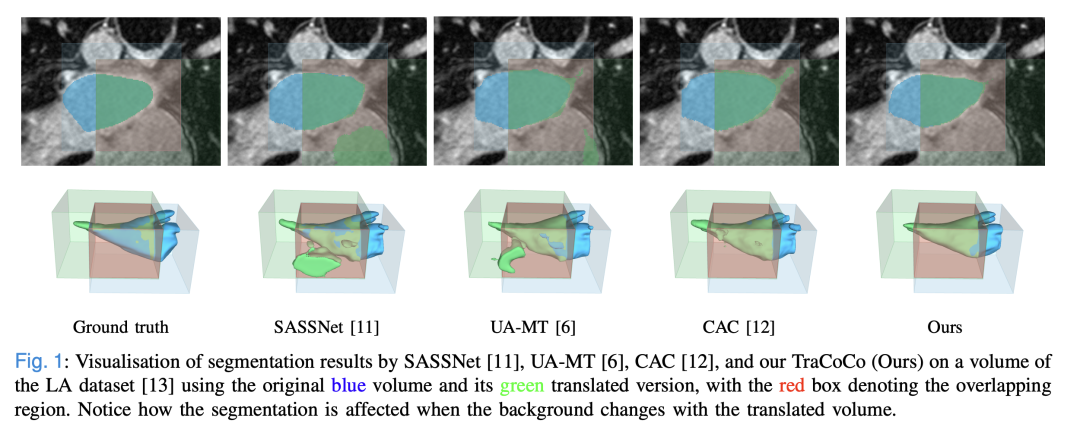
3D医学图像分割方法虽然已取得成功,但它们依赖于大量体素级别标注数据,这是一个亟需解决的问题,因为获得这些标注的成本非常高。为了克服这一挑战,半监督学习(SSL)通过结合大量未标注数据和少量标注数据来训练模型,从而减少对标注数据的需求。
最成功的SSL方法基于一致性学习,通过最小化模型在未标注数据的不同扰动视图下的响应差异来实现一致性。然而,这些扰动通常保持视图之间的空间输入上下文较为一致,这可能导致模型从空间上下文中学习分割模式,而不是从前景对象中学习。
为解决这一问题,本文提出了一种新的SSL方法,称为Translation Consistent Co-training(TraCoCo)。TraCoCo通过改变输入数据视图的空间输入上下文来扰动视图,使模型能够从前景对象中学习分割模式。同时,本文还提出了一种新的Confident Regional Cross-Entropy(CRC)损失,该损失旨在提高训练的收敛性并保持对共训练伪标签错误的鲁棒性。
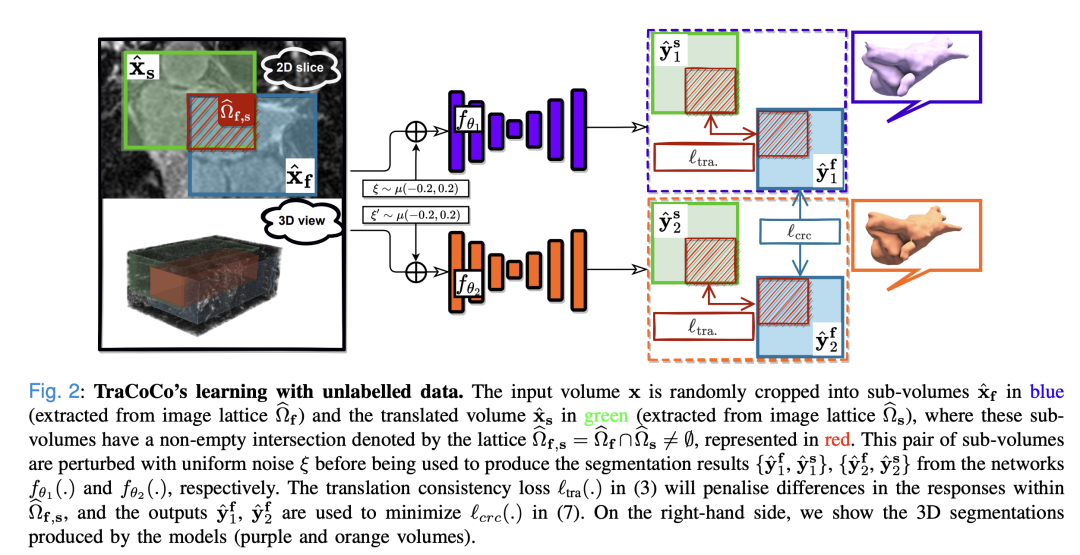
Translation Consistent Co-training (TraCoCo):
TraCoCo是一种基于互助学习(Co-training)框架的方法,该框架通过翻译一致性(Translation Consistency)来扰动输入数据的空间上下文,从而减少模型对背景模式的“记忆”,确保模型专注于前景对象的分割。
模型框架:TraCoCo包含两个初始参数不同的独立网络(通常为VNet或3D-UNet)。输入数据通过随机裁剪生成两个子体积(sub-volumes),这两个子体积的空间上下文不同,但在体素网格中存在重叠区域。
模型目标:为了确保模型在不同空间上下文下的前景分割结果一致,TraCoCo引入了多种损失函数,包括监督学习损失、半监督学习损失和翻译一致性损失。本文的方法实现通过最小化以下损失函数来进行训练:
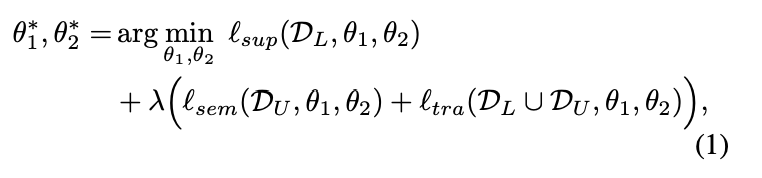
其中,分别由监督学习损失,半监督学习损失,和翻译一致性损失组成。
监督学习损失(Supervised Learning Loss):
监督学习损失通过带标注的数据集D_L进行计算,包括体素级交叉熵损失(Cross-Entropy Loss)和Dice损失,用来优化模型的分割性能。损失函数定义为:
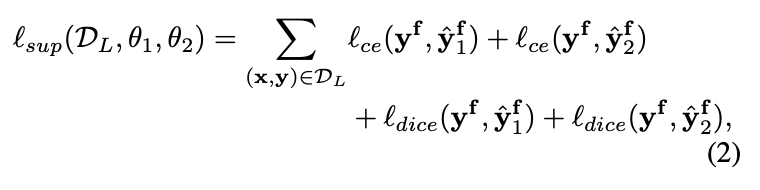
Translation Consistency Loss:
Translation Consistency Loss主要目标是确保模型在不同空间上下文下的分割结果一致。具体实现过程如下:从训练体积中随机提取两个中心不同的子体积
损失函数定义为:
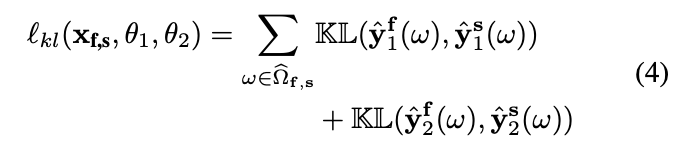
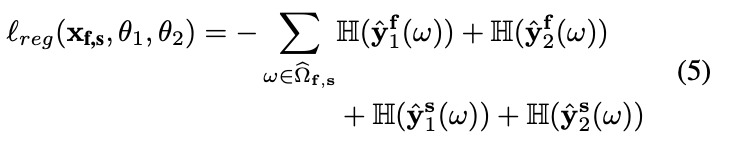
其中,公式4是Kullback-Leibler(KL)散度,用于计算两个子体积在重叠区域的分割结果之间的差异;公式5是基于负熵的正则化损失,用于平衡训练体素中的前景和背景类。
半监督学习损失(Semi-supervised Learning Loss):
半监督学习损失用于强化两个模型之间的分割一致性,Confident Regional Cross-Entropy (CRC) Loss, 其定义为:
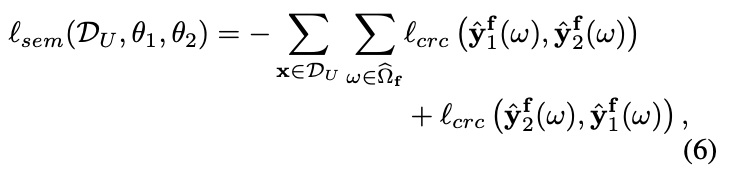
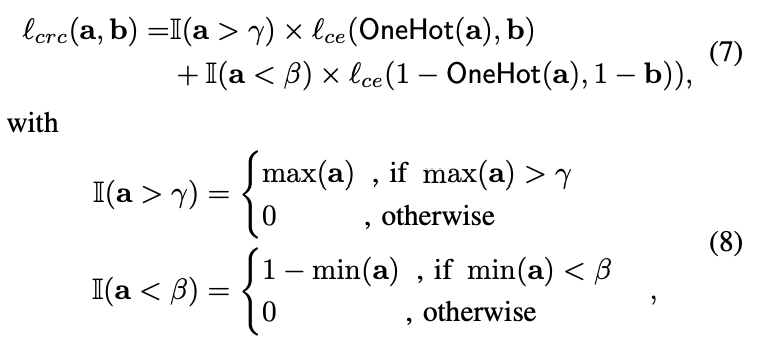
3D CutMix:
为了进一步提高训练的泛化性,本文采用了3D CutMix技术。实现为随机生成一个3D二值掩码,该掩码包含一个随机定义位置和尺寸的“1”方块。掩码应用在未标注数据和伪标签上,定义为:
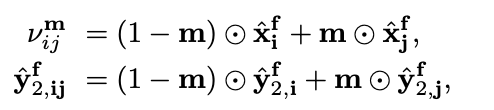
实验
本文在四个公开的3D医学图像半监督分割数据集上进行了实验,包括:
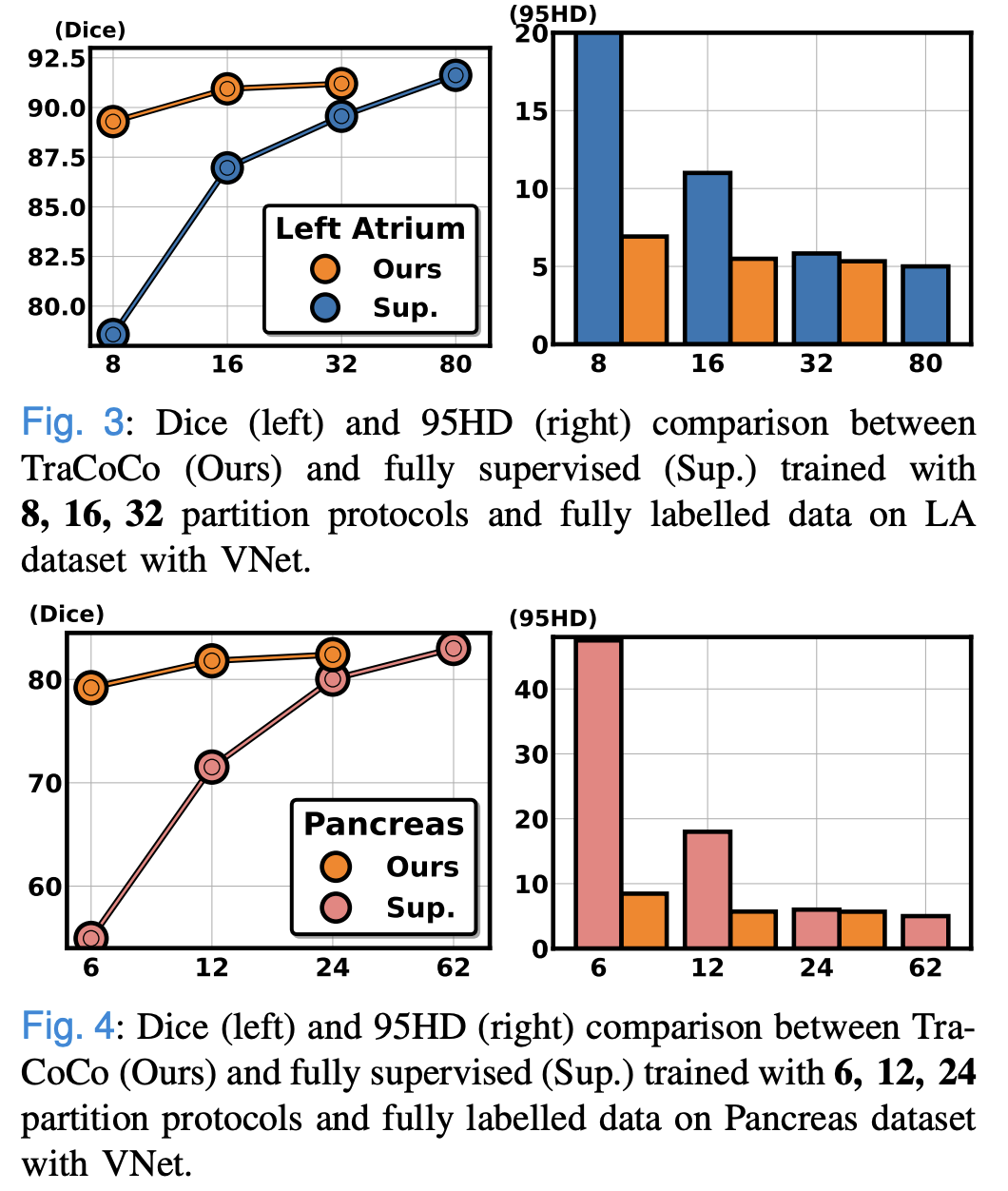
左心房(Left Atrium, LA):100个3D MRI体积,其中80个用于训练,20个用于测试。
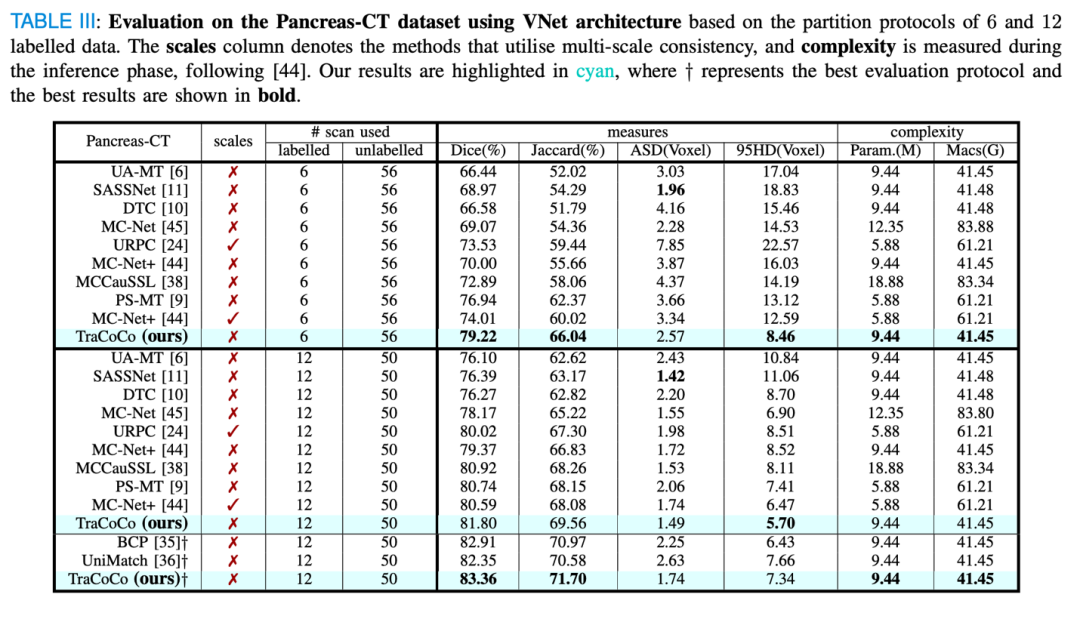
胰腺CT(Pancreas-CT):82个对比增强的CT扫描,采用特定的预处理步骤。
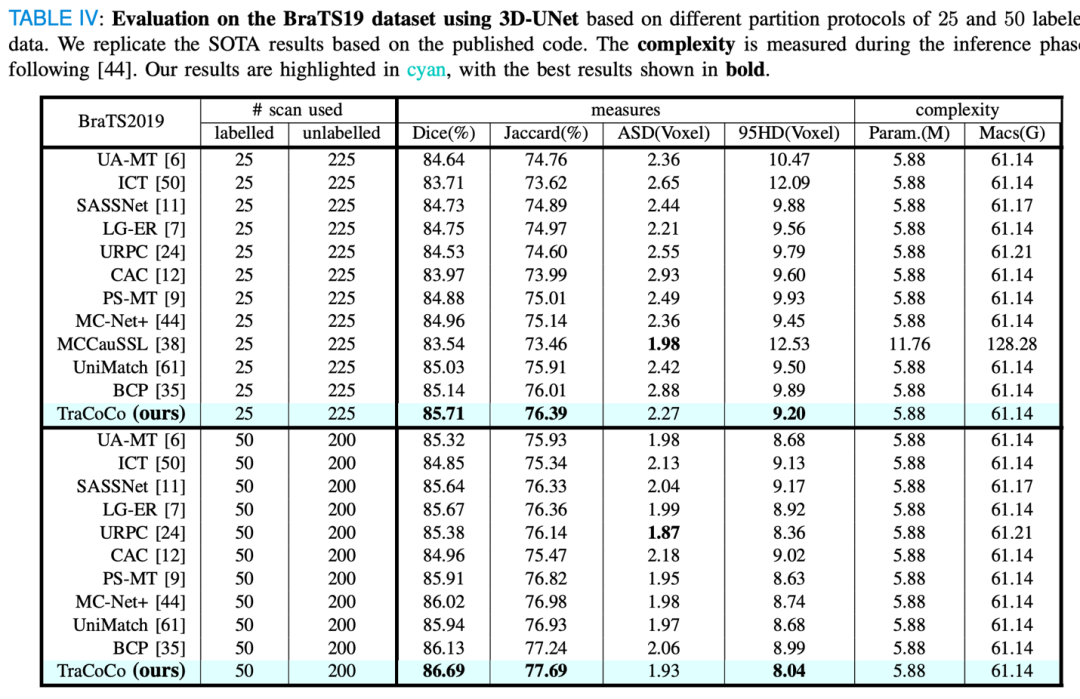
脑肿瘤分割2019(BraTS19):包含335个脑部MRI样本,每个样本包含四种扫描类型(T1, T1-ce, T2, FLAIR)。
自动心脏诊断挑战(ACDC):该数据集包含100个心脏MRI扫描,本文使用了其2D切片进行半监督学习。
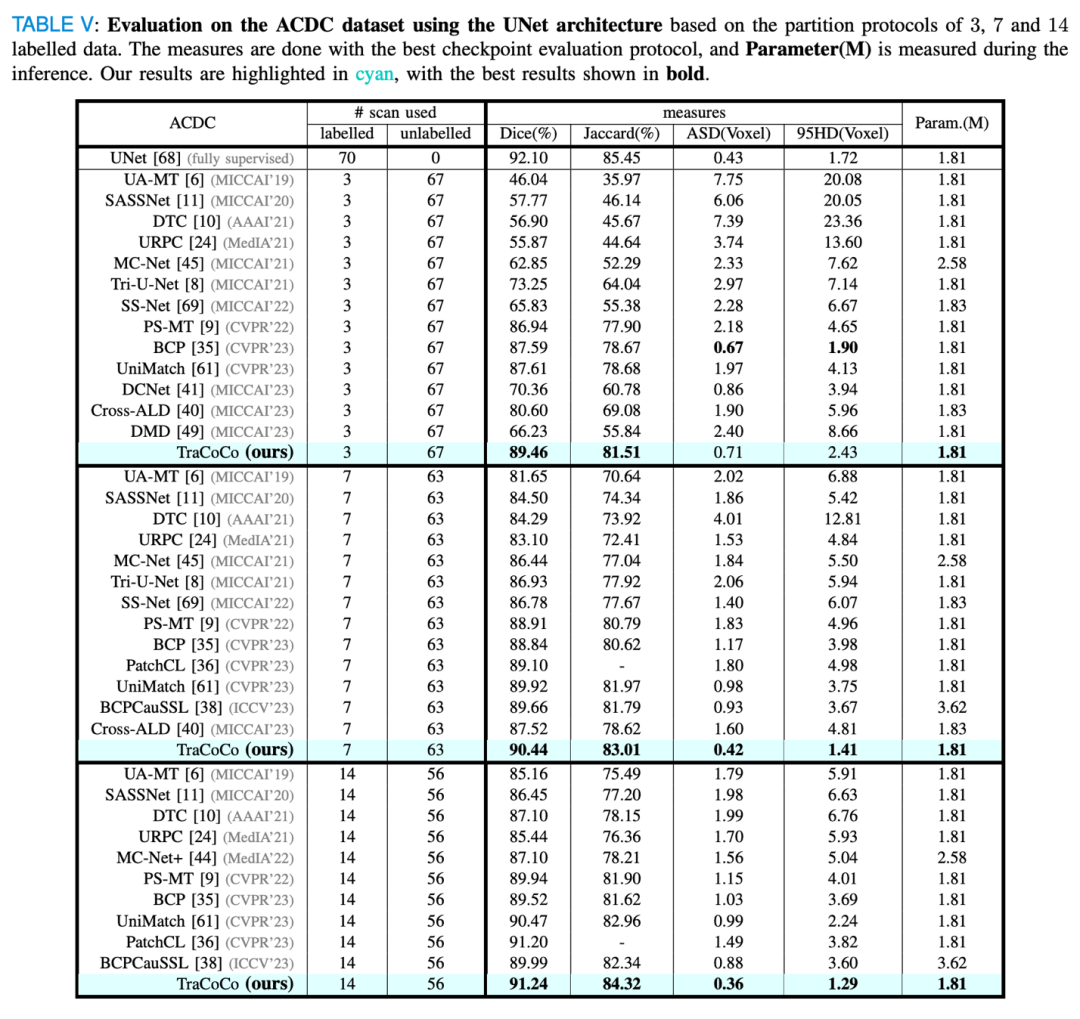
实验设置:采用了VNet和3D-UNet作为模型架构,实验评估指标包括Dice、Jaccard、平均表面距离(ASD)和95% Hausdorff距离(95HD)。实验结果显示,TraCoCo在这些基准测试上均优于现有的最先进(SOTA)方法,特别是在标注数据较少的情况下

总结
本文提出了TraCoCo和CRC损失在3D和2D医学图像分割任务中的有效性,证明了其在减少对背景模式的“记忆”以及提高前景对象分割精度方面的能力。未来的工作计划包括扩展TraCoCo到更多复杂的医学图像任务,并在多模态医学图像中探索其半监督学习能力 。
何恺明在MIT授课的课件PPT下载
在CVer公众号后台回复:何恺明,即可下载本课程的所有566页课件PPT!赶紧学起来!
ECCV 2024 论文和代码下载
在CVer公众号后台回复:ECCV2024,即可下载ECCV 2024论文和代码开源的论文合集CVPR 2024 论文和代码下载
在CVer公众号后台回复:CVPR2024,即可下载CVPR 2024论文和代码开源的论文合集Mamba和医学影像交流群成立
扫描下方二维码,或者添加微信号:CVer2233,即可添加CVer小助手微信,便可申请加入CVer-Mamba或者医学影像微信交流群。另外其他垂直方向已涵盖:目标检测、图像分割、目标跟踪、人脸检测&识别、OCR、姿态估计、超分辨率、SLAM、医疗影像、Re-ID、GAN、NAS、深度估计、自动驾驶、强化学习、车道线检测、模型剪枝&压缩、去噪、去雾、去雨、风格迁移、遥感图像、行为识别、视频理解、图像融合、图像检索、论文投稿&交流、PyTorch、TensorFlow和Transformer、NeRF、3DGS、Mamba等。
一定要备注:研究方向+地点+学校/公司+昵称(如Mamba、多模态学习或者医学影像+上海+上交+卡卡),根据格式备注,可更快被通过且邀请进群▲扫码或加微信号: CVer2233,进交流群
CVer计算机视觉(知识星球)来了!想要了解最新最快最好的CV/DL/AI论文速递、优质实战项目、AI行业前沿、从入门到精通学习教程等资料,欢迎扫描下方二维码,加入CVer计算机视觉(知识星球),已汇集上万人!
▲扫码加入星球学习▲点击上方卡片,关注CVer公众号
整理不易,请赞和在看






















 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








