在3D Slicer(4.10.1)中进行三维体数据的图像分割标注。
扩展教程:
3D Slicer - 医学图像检测标注教程(矩形框)
3D Slicer - 医学图像检测标注教程(导入查看标签与修改标签)
1. 读取数据
三维体数据一般为DICOM格式或者NIFIT格式,
DICOM:
将包含.dcm文件序列的文件夹拖入3D Slicer,或者点击左上角的dcm图标:

在DICOM Browser Import,导入.dcm序列后,examime and load:

NIFIT:
直接将.nii文件拖入3D Slicer即可。
2. 勾画标签
1. 选择Segmentations模块,点击"Create new Segmentation":

Create之后可以通过Rename修改名字。
2. 添加Segment:
点击Edit selected之后,会到Segment Editor模块。
3. 编辑Segment:
双击Segment的Color可修改颜色,双击Name可修改名字。

通过Effects下的不同工具可在体数据的三个视图上勾画label:

点击Show details查看各个工具的使用方法。点击show 3D可显示segment三维图。
3. 保存标签
勾画label完成后,切换到Data模块,可以看到刚刚所勾画的label:

注意,有多个Segment的时候,若只想保存其中一个,则需要将其他的Segment设为不可见,即关闭Segment右侧的小眼睛。每次保存的是同级目录下的所有设为可视的Segment。
选中想要保存的segment,右键,选择"Export visible segments to binary labelmap",再选中出现的labelmap:

右键,选择"Export to DIOCM…",选择路径进行保存。保存为DICOM格式后,也可以重新打开,然后将其保存为NIFIT格式。
3. 显示多个标签
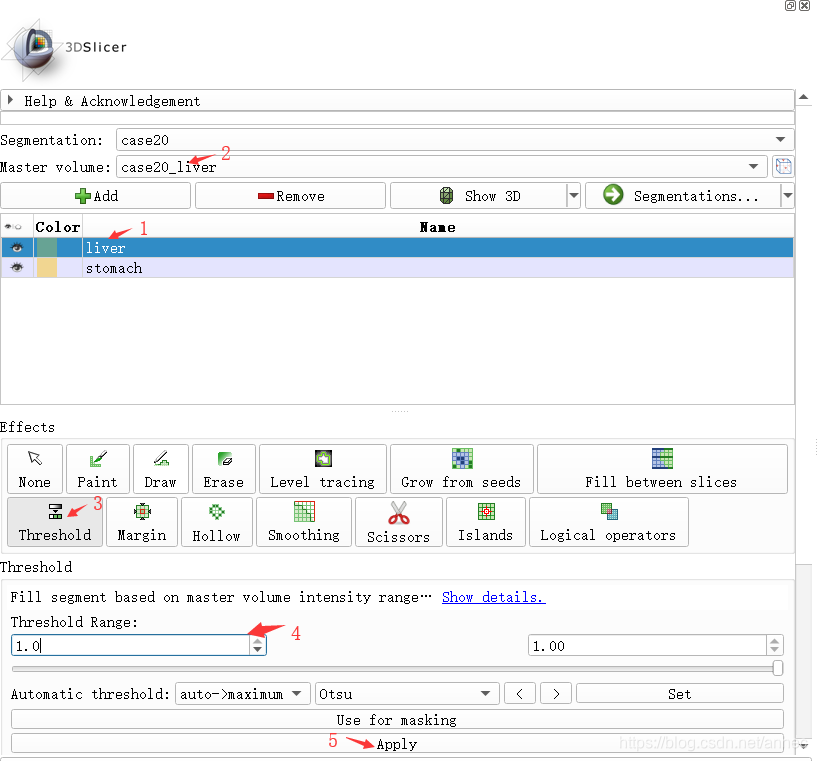
将原始体数据和多个label的.nii同时打开,然后在Segment模块中,首先新建一个Segmentation,然后为每一个label添加一个Segment,具体方法为:
- 选中一个Segment;
- 选择labeld对应的Volume;
- 点击Threshold工具;
- 设置高低阈值为label的值(可将鼠标在右侧可视化图中移动,左下角会显示当前鼠标位置的灰度值);
- 点击apply。
为每个label都添加Segment并apply之后,可以在右侧看到不同label的可视化图:

将Master Volume切换为原始体数据:
 就可以看到label在原始体数据上的叠加效果:
就可以看到label在原始体数据上的叠加效果:

























 686
686

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








