我自己在用R做各种分析时有不少需要反复用到的基础功能,比如一些简单的统计呀,画一些简单的图等等,虽说具体实现的代码也不麻烦,但还是不太想每次用的时候去找之前的代码。
索性将常用的各种函数整成了一个包:pcutils,
网址:https://github.com/Asa12138/pcutils
从CRAN安装:
install.packages("pcutils")
但目前还是建议从github安装,包含的功能会多一些:
install.packages("devtools")
devtools::install_github('Asa12138/pcutils',dependencies=T)
Introduction
层级数据展示是数据分析中常有的需求,比如物种分类等级,KEGG功能层次等。
我们以下列test数据为例,这是一个物种分类等级的表格。表格从左到右具有包含关系,最后一列是物种的丰度:
data(otutab)
cbind(taxonomy, num = rowSums(otutab))[1:10, ] -> test
| Kingdom | Phylum | Class | Order | Family | Genus | Species | num |
|---|---|---|---|---|---|---|---|
| k__Bacteria | p__Actinobacteria | c__Actinobacteria | o__Actinomycetales | f__Thermomonosporaceae | g__un_f__Thermomonosporaceae | s__un_f__Thermomonosporaceae | 26147 |
| k__Bacteria | p__Proteobacteria | c__Betaproteobacteria | o__Burkholderiales | f__Comamonadaceae | g__Pelomonas | s__Pelomonas_puraquae | 25217 |
| k__Bacteria | p__Proteobacteria | c__Gammaproteobacteria | o__Pseudomonadales | f__Pseudomonadaceae | g__Rhizobacter | s__Rhizobacter_bergeniae | 16592 |
| k__Bacteria | p__Bacteroidetes | c__Flavobacteriia | o__Flavobacteriales | f__Flavobacteriaceae | g__Flavobacterium | s__Flavobacterium_terrae | 16484 |
| k__Bacteria | p__Proteobacteria | c__Gammaproteobacteria | o__Pseudomonadales | f__Pseudomonadaceae | g__Rhizobacter | s__un_g__Rhizobacter | 13895 |
| k__Bacteria | p__Proteobacteria | c__Betaproteobacteria | o__Burkholderiales | f__un_o__Burkholderiales | g__un_o__Burkholderiales | s__un_o__Burkholderiales | 10937 |
| k__Bacteria | p__Actinobacteria | c__Actinobacteria | o__Actinomycetales | f__Streptomycetaceae | g__Streptomyces | s__un_g__Streptomyces | 10813 |
| k__Bacteria | p__Actinobacteria | c__Actinobacteria | o__Actinomycetales | f__Pseudonocardiaceae | g__Lentzea | s__Lentzea_flaviverrucosa | 9508 |
| k__Bacteria | p__Actinobacteria | c__Actinobacteria | o__Actinomycetales | f__Micromonosporaceae | g__Actinoplanes | s__un_g__Actinoplanes | 8518 |
| k__Bacteria | p__Proteobacteria | c__Alphaproteobacteria | o__Rhizobiales | f__Rhizobiaceae | g__Rhizobium | s__un_g__Rhizobium | 7789 |
我们有很多种方法来展示这种类型的层次数据,比如桑基图,旭日图,包珠图,Treemap等。
桑基图
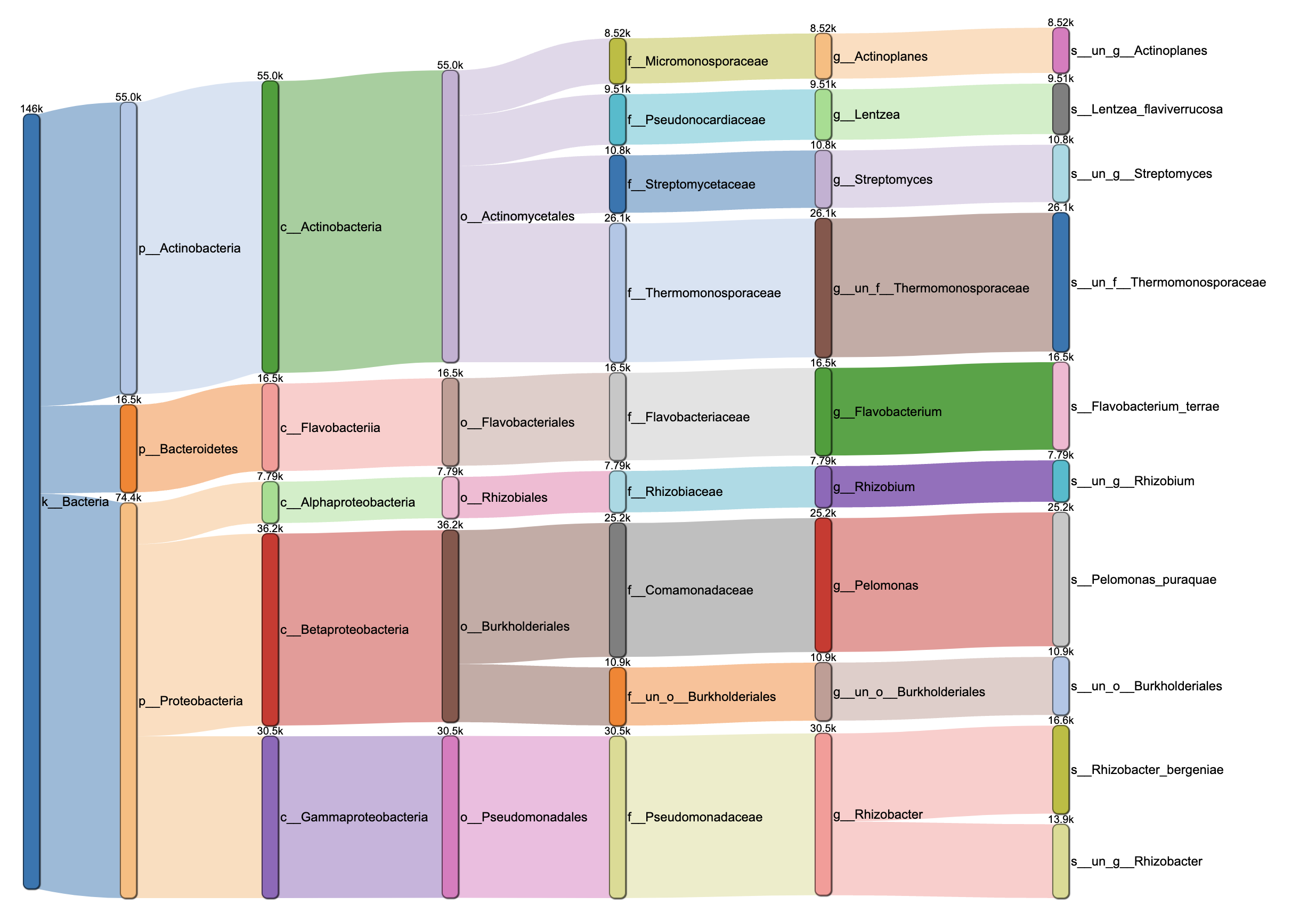
桑基图(Sankey diagram)是一种常用的层次数据展示方法,它通过使用有向连接不同的节点来显示流动的路径和量级。
- 使用
sankeyD3包绘制桑基图:
my_sankey(test,mode = "sankeyD3")
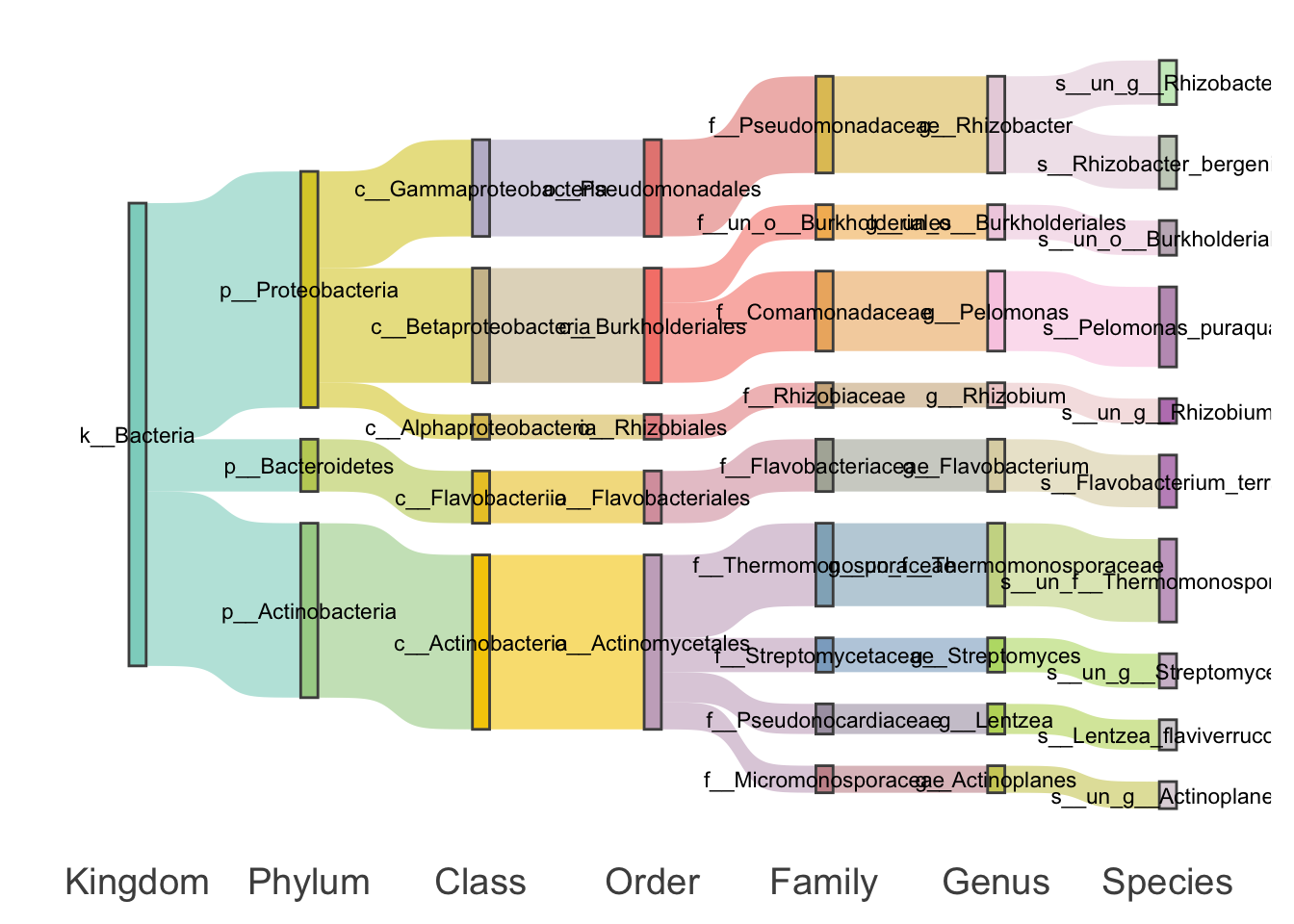
- 使用
ggsankey包绘制桑基图:
my_sankey(test, mode = "ggsankey", space = 1e4)
旭日图
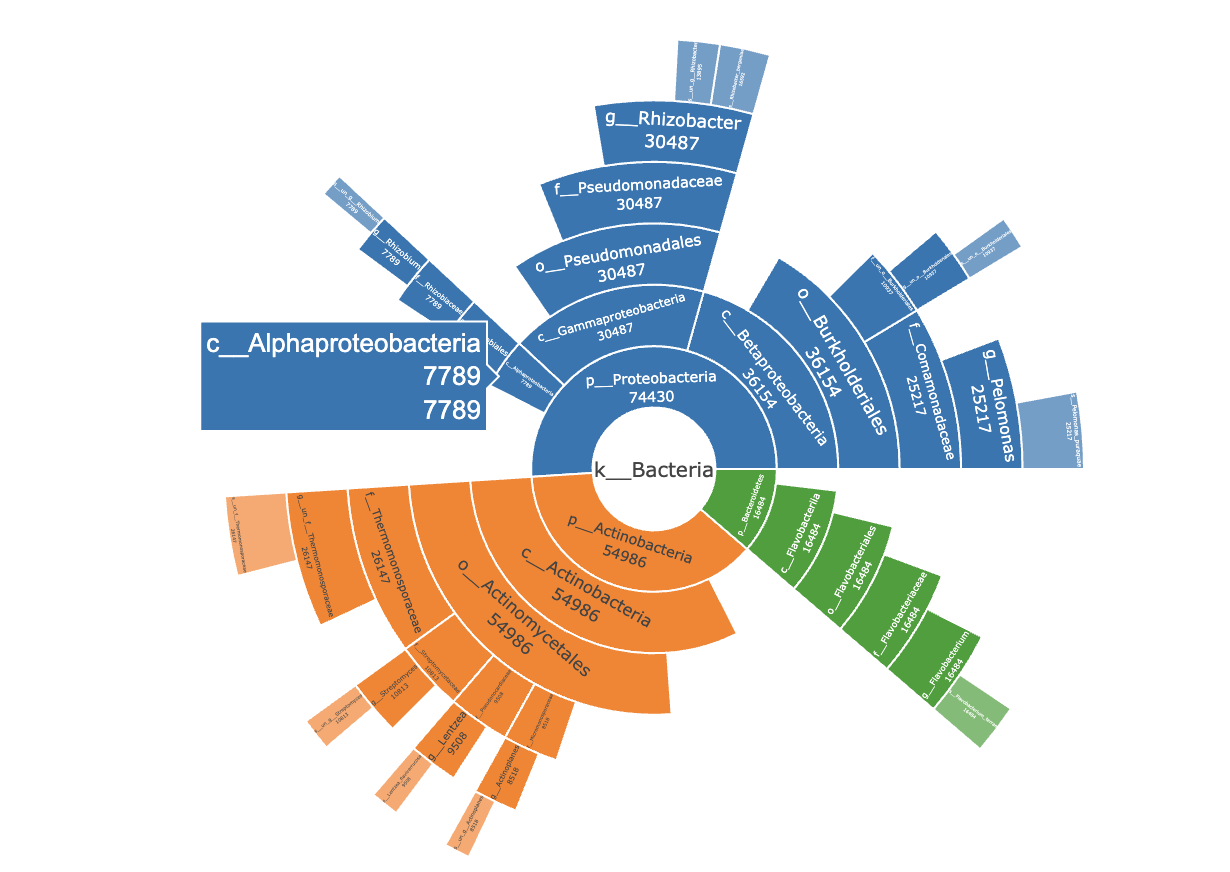
旭日图(sunburst)是饼图的变形,简单来说是多个饼图的组合升级版。饼图只能展示一层数据的占比情况,而旭日图不仅可以展示数据的占比情况,还能厘清多级数据之间的关系。
my_sunburst(test)
多层环图
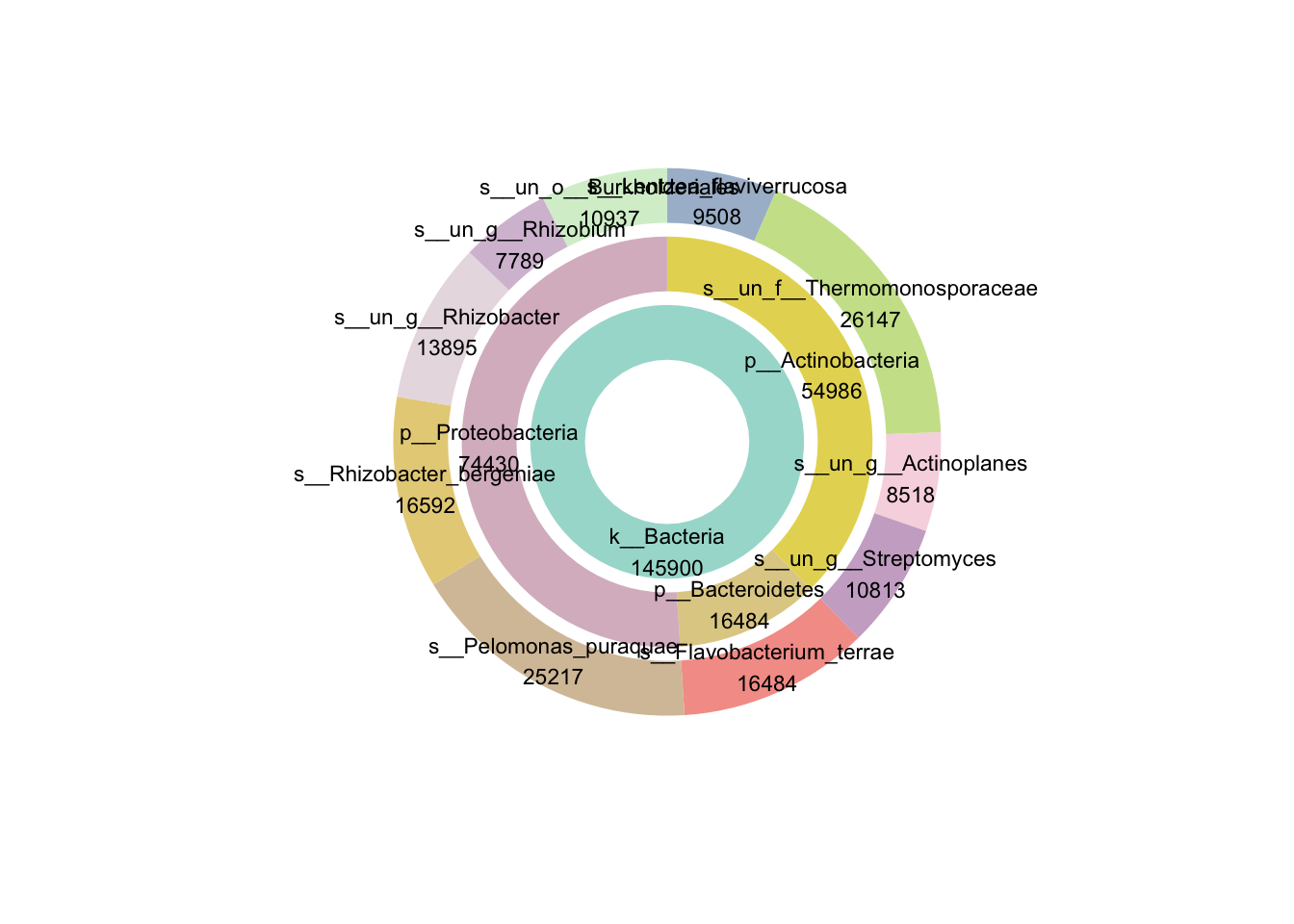
多层环图是一种多层次数据展示方法,它将数据分成多个环,每个环代表一层数据。可以使用gghuan2函数绘制:
gghuan2(arrange_all(test[,c(1,2,7,8)]))+
scale_fill_pc(n = 14)
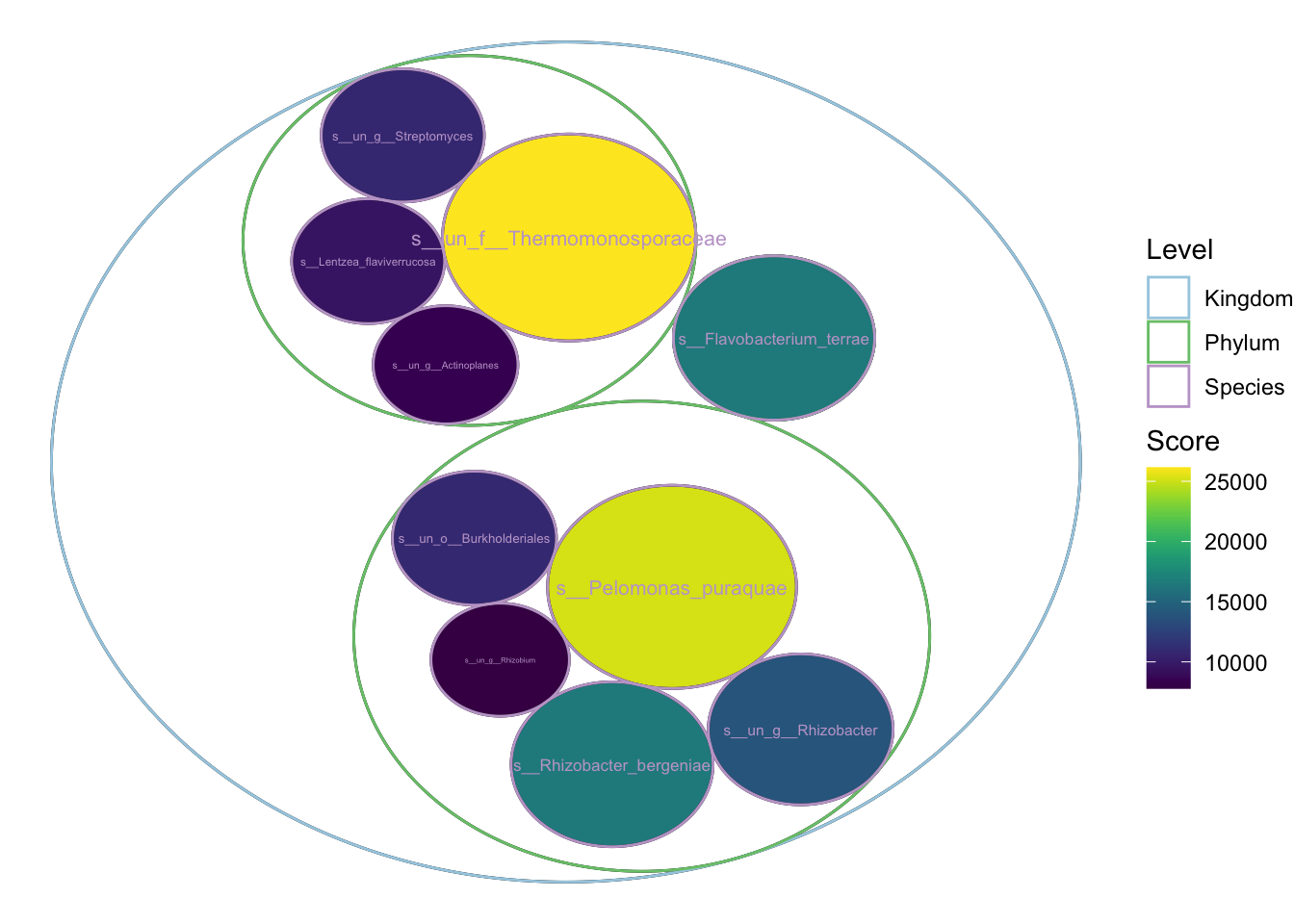
包珠图
包珠图(Circular Packing),是一种比较特殊的分类树状图,以气泡之间的包含关系展示层级关系,以气泡面积(或半径)展示数值的大小。
my_circle_packing(test[,c(1,2,7,8)],show_level_name = T)+
viridis::scale_fill_viridis(na.value = NA)+
scale_color_pc(palette = "col2")
Treemap
Treemap是一种层次数据展示方法,它将数据分成矩形,矩形的大小代表数值的大小,矩形之间的包含关系代表层级关系。
my_treemap(test[,c(2,7,8)])

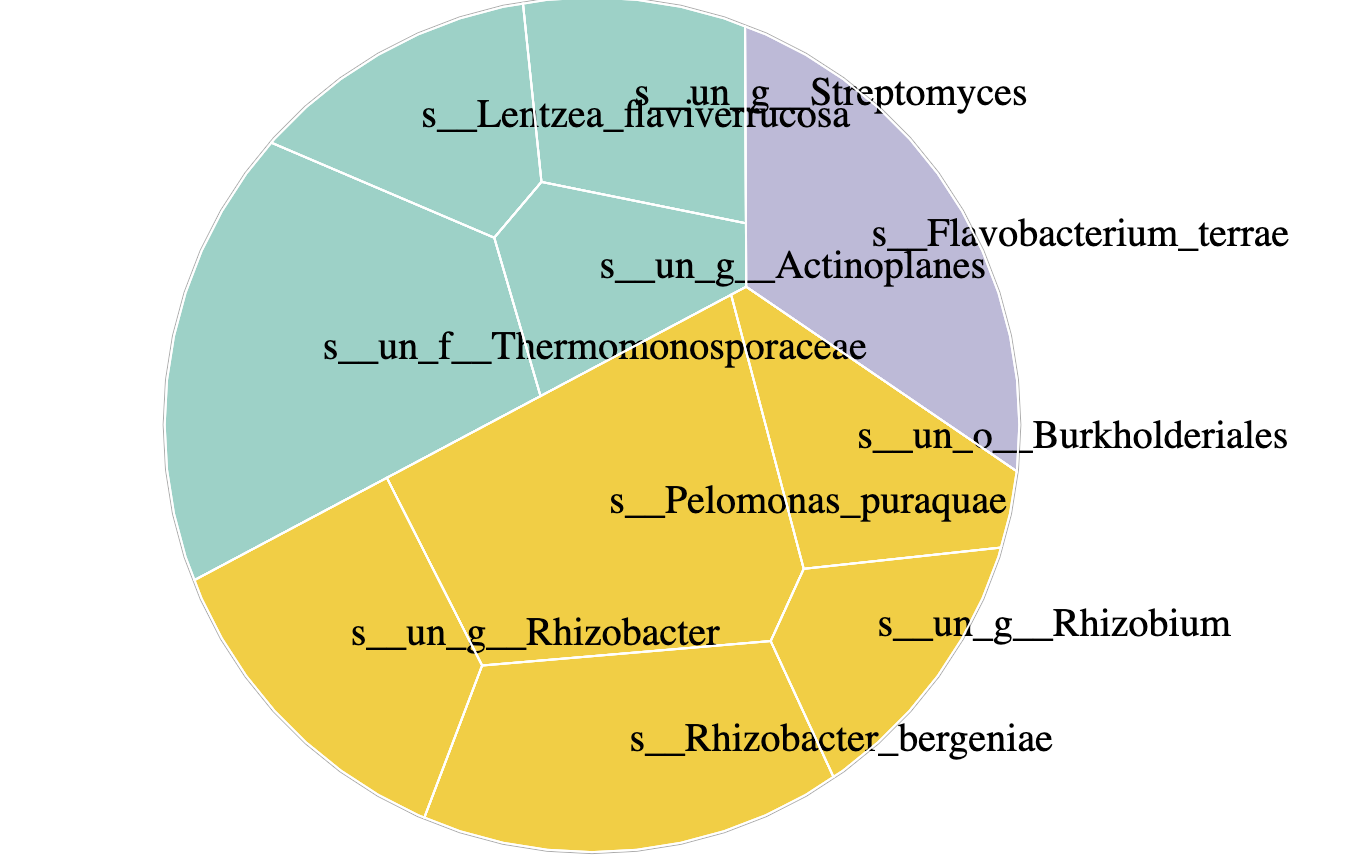
Voronoi Treemap
Voronoi Treemap是一种基于Voronoi tessellation的层次数据展示方法,它将数据分成多边形,多边形的边界代表数据之间的距离,多边形的面积代表数值的大小,多边形之间的包含关系代表层级关系。
my_voronoi_treemap(test[,c(2,7,8)])
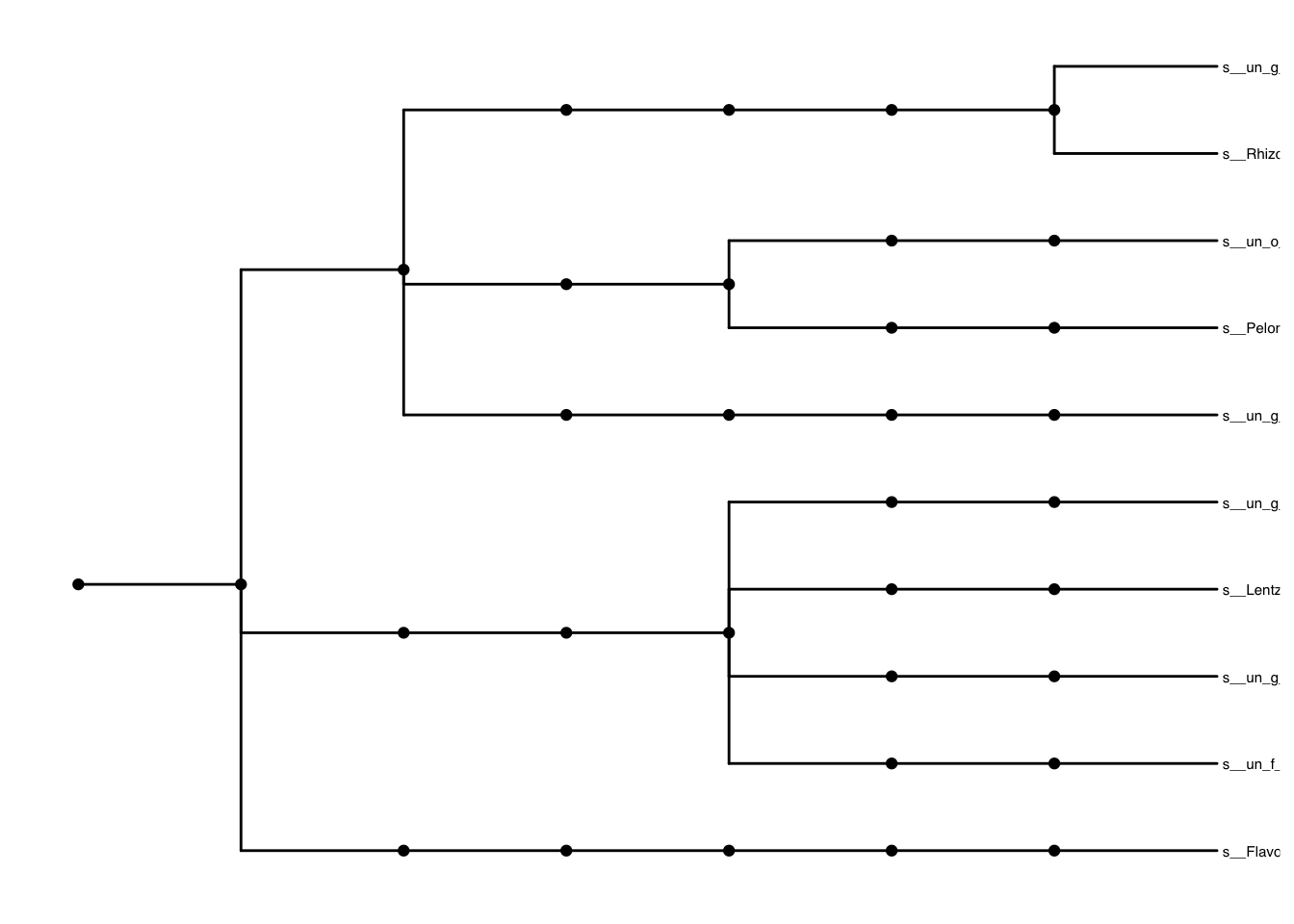
树图
我在pctax包中提供了一个函数df2tree可以将层级数据转换为树结构数据(phylo对象),树图就可以用ggtree之类的包进行绘图,可以参考我之前介绍过的R绘制系统发育树的基本用法,进阶使用。
# install.packages("pctax")
tr=pctax::df2tree(test[,1:7])
class(tr)
## [1] "phylo"
ggtree::ggtree(tr)+
ggtree::geom_tiplab(size = 2)+
ggtree::geom_nodepoint()
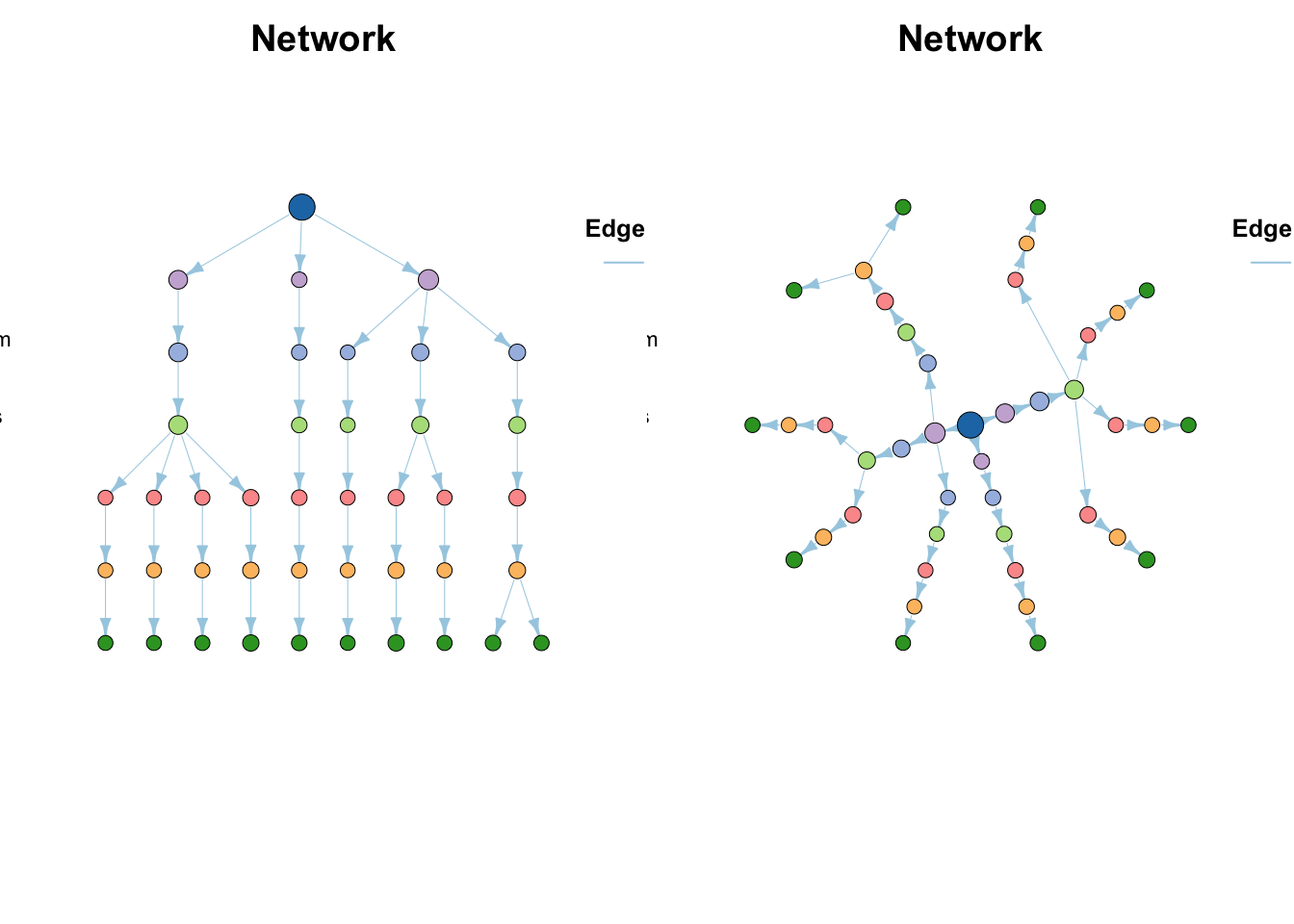
网络图
我在MetaNet包提供了将层级数据转换为网络图的功能,可以用MetaNet包进行绘图。
# install.packages("MetaNet")
go=MetaNet::df2net_tree(test)
par(mfrow=c(1,2))
plot(go,edge_width_range=c(0.5,0.5))
plot(go,MetaNet::as_circle_tree(),edge_width_range=c(0.5,0.5))
所有的可视化方法都是为展示数据服务的,
我整合这些函数也是希望可以更关注数据本身,花更少的精力在调节图形上,先快速对我们的数据有整体的把握。
pcutils的初衷还是迎合我自己的编程与数据分析习惯的,所以可能并不适合所有人,大家也可以直接fork并修改我的源码,欢迎大家提出建议与意见。

关注公众号 ‘bio llbug’,获取最新推送。






















 103
103

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








