Naopore基因组数据组装软件---NextDenovo下载试用
1. 下载解压软件
NextDenovo软件是武汉希望组公司开发的组装软件,该软件是基于“string graph-based”组装软件,可以使用多样化三代数据组装(nanopore,pacbio, HiFi),组装HiFi数据时没有矫正过程,看文档中将HiFi这个选项划掉了,目前三代数据组装还是需要矫正这个步骤的。
该软件设计类似 canu 的“校正后组装”策略(对于PacBio的HiFi reads,则不需要校正步骤,因为本身这部分数据的质量就很高了。),但需要的计算资源和存储空间要少得多。 组装完成后,per-base 精度约为 98-99.8%,若要进一步提高单 base 精度,可以使用NextPolish(希望组的另一个软件)。
NextDenovo共有两个模块组成NextCorrect 和NextGraph,显然NextCorrect 使用来矫正的工具,NextCorrect 可纠正长reads中约15% 的测序错误(这样说可能在Nanopore的组装方面更具优势),而NextGraph则用来组装。
需要注意的是,NextDenovo不可以进行大基因组组装项目(genome size > 3.5 Gb),过大的基因组需要开发公司内部版本。那对于我们普通用户来说,显然该软件不适合小麦基因组组装(~16 Gb)。
但可以看出NextDenovo同样可以作为矫正软件使用,随后使用其他软件进行组装也是可以的(wtdbg,smartdenovo,miniasm,fly)。
https://github.com/Nextomics/NextDenovo/issues/52
软件github维护者回复:
在Github找到该软件地址,下载编译好的版本:
wget -c wget https://github.com/Nextomics/NextDenovo/releases/download/v2.5.0/NextDenovo.tgz
## Note:
If you get an error like version 'GLIBC_2.14' not found or liblzma.so.0:
cannot open shared object file, Please download this version.
此外,本版本加入了Python 模块要求:
Python (Support python 2 and 3):
Paralleltask
## 下载python包
pip install paralleltask
解压NextDenovo完成:
## 解压软件
tar -vxzf NextDenovo.tgz && cd NextDenovo
## 调用软件版本信息
./nextDenovo -v
nextDenovo v2.5.0
## 调用软件参数信息
./nextDenovo -h
usage: nextDenovo [-l FILE] [-v] [-h]
nextDenovo:
Fast and accurate de novo assembler for long reads
exmples:
nextDenovo run.cfg
For more information about NextDenovo, see https://github.com/Nextomics/NextDenovo
optional arguments:
-l FILE, --log FILE log file (default: pidXXX.log.info)
-v, --version show program's version number and exit
-h, --help please use the config file to pass parameters
2. 简单试用
该软件中包含一个小的测试数据集合(1000条fasta数据,用于初步试用软件)
测试序列
zcat ./test_data/reads_test.fa.gz | wc -l
2000
zgrep ">" -c ./test_data/reads_test.fa.gz
1000 ## 1000条序列
cat input.fofn
## 待组装的fasta
./reads_test.fa.gz
cat run.cfg
## 组装配置文件内容
[General]
job_type = local
job_prefix = nextDenovo
task = all # 'all', 'correct', 'assemble'
rewrite = yes # yes/no
deltmp = yes
rerun = 3
parallel_jobs = 2
input_type = raw
read_type = clr
input_fofn = ./input.fofn
workdir = ./01_rundir
[correct_option]
read_cutoff = 1k ## 设置 read 长度阈值
genome_size = 308161 ## 设置基因组大小
pa_correction = 2
sort_options = -m 1g -t 2
minimap2_options_raw = -t 8
correction_options = -p 15
[assemble_option]
minimap2_options_cns = -t 8
nextgraph_options = -a 1
测试命令
进行组装:
./nextDenovo ./test_data/run.cfg
3. 三套测试数据组装
该软件教程中公布了三套数据的组装测试,分别包括三个不同的物种(人、拟南街和果蝇),三套数据非常具有代表性:
人类基因组
拟南芥F1基因组(杂合度 ~ 1%)
果蝇基因组
使用果蝇数据组装的时候,同时比较了不同组装软件的性能:
NextDenovo;
Canu;
Flye;
Shasta
评估过程使用Quast v5.0.2:
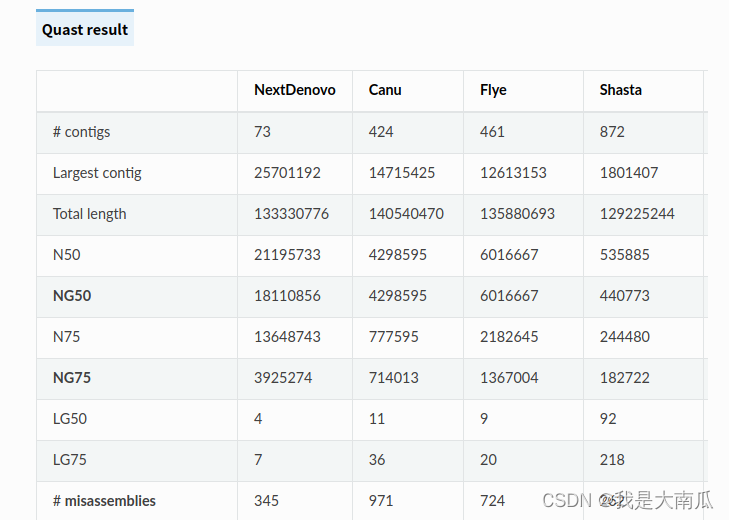
就果蝇评估结果而言,NextDenovo表现出不错的组装性能。
后面可以使用这个软件练习一下。
4. 拟南芥Nanopore数据测试
先使用NextDenovo软件中seq_stat评估软件的seed_cutoff:
之前genome survey基因组大小为138Mb,-d为预测矫正的测序深度取值30-45(默认为45):
ls *.gz > input.file ## CRR302667.fastq.gz
~/bin/nextDenovo_v2.5/NextDenovo/bin/seq_stat -g 138Mb -d 30 input.file -o seq_stat_results.txt ## -d default 45
根据软件输出结果,得到seed_cutoff=126736 bp:
[Read length histogram ('*' =~ 2517 reads)]
0 999 0
1000 1999 249221 ***************************************************************************************************
2000 2999 191207 ***************************************************************************
....
[Read length stat]
Types Count (#) Length (bp)
N10 38874 115421
N20 95226 88910
N30 166382 71560
N40 254137 58128
N50 362714 46716
N60 499462 36567
N70 677767 27407
N80 924760 18964
N90 1313680 10879
Types Count (#) Bases (bp) Depth (X)
Raw 3064191 56814196989 411.70
Filtered 546501 278888284 2.02
Clean 2517690 56535308705 409.68
*Suggested seed_cutoff (genome size: 138.00Mb, expected seed depth: 30, real seed depth: 30.00): 126736 bp
所以可以设定NextDenovo组装配置文件:
run2.cfg
[General]
job_type = local
job_prefix = nextDenovo
task = all # 'all', 'correct', 'assemble'
rewrite = yes # yes/no
deltmp = yes
rerun = 3
parallel_jobs = 5
input_type = raw
read_type = ont # ont,clr,hifi
input_fofn = ./input.file # contain nanopore files
workdir = ./02_rundir # output dir
[correct_option]
read_cutoff = 1k
seed_cutoff = 126736 ## seq_stat suggest cutoff
genome_size = 137964725 ## jellyfish estimate genome size with Illumina
pa_correction = 2
sort_options = -m 10g -t 4
minimap2_options_raw = -t 4
correction_options = -p 4
[assemble_option]
minimap2_options_cns = -x ava-ont -t 4 -k17 -w17 ## need minimap2-nd params finds
nextgraph_options = -a 1
组装结果128953860(128.95Mb),contig个数为18个:
## ll
总用量 12
drwxr-xr-x 5 debian debian 4096 9月 1 15:28 01.raw_align
drwxr-xr-x 4 debian debian 4096 9月 2 04:48 02.cns_align
drwxr-xr-x 5 debian debian 4096 9月 2 05:04 03.ctg_graph
## cd 03.ctg_graph
## cat nd.asm.fasta.stat
Type Length (bp) Count (#)
N10 16493425 1
N20 16041071 2
N30 15775569 3
N40 15152058 4
N50 15096536 5
N60 15096536 5
N70 13884570 6
N80 11369235 7
N90 4463503 9
Min. 341250 -
Max. 16493425 -
Ave. 7164103 -
Total 128953860 18
此外,也看了不设定seed_cutoff值的结果:
run.cfg
[General]
job_type = local
job_prefix = nextDenovo
task = all # 'all', 'correct', 'assemble'
rewrite = yes # yes/no
deltmp = yes
rerun = 3
parallel_jobs = 5
input_type = raw
read_type = ont # ont,clr,hifi
input_fofn = ./input.file # contain nanopore files
workdir = ./01_rundir # output dir
[correct_option]
read_cutoff = 1k
genome_size = 137964725
pa_correction = 2
sort_options = -m 10g -t 4
minimap2_options_raw = -t 4
correction_options = -p 4
[assemble_option]
minimap2_options_cns = -x ava-ont -t 4 -k17 -w17 ## need minimap2-nd params finds
nextgraph_options = -a 1
组装结果为127365625 bp,contig数目为16:
## cat nd.asm.fasta.stat
Type Length (bp) Count (#)
N10 16284040 1
N20 15773625 2
N30 15616219 3
N40 15150589 4
N50 15089107 5
N60 15089107 5
N70 14020371 6
N80 11371677 7
N90 4845570 9
Min. 339658 -
Max. 16284040 -
Ave. 7960351 -
Total 127365625 16
上面的NextDenovo:
echo first try
echo `date` start nextdenovo
~/bin/nextDenovo_v2.5/NextDenovo/nextDenovo run.cfg
echo `date` end nextdenovo
echo second try
echo `date` start nextdenovo
~/bin/nextDenovo_v2.5/NextDenovo/nextDenovo run2.cfg
echo `date` end nextdenovo
以上是拟南芥的Nanopore数据组装,当然这个组装的数据未进行过滤,两次组装只是seed_cutoff不同,contig数目和基因组大小整体较小,偏向128Mb的结果,不只N50, N90也都不错。当然这只是初步组装,可以比对到基因组看看质量。
参考:
https://github.com/Nextomics/NextDenovo (Github地址)
https://nextdenovo.readthedocs.io/en/latest/QSTART.html#quick-start (快速开始的例子)
https://nextdenovo.readthedocs.io/en/latest/downloads/3b331fa278b7706b8c6510800318fa52/TEST4.pdf (官方例子)
https://nextdenovo.readthedocs.io//downloads/en/latest/pdf/ (参数解释,2022-7-13)























 2370
2370











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








