1. 图数据集 种类
https://pytorch-geometric.readthedocs.io/en/latest/modules/datasets.html#graph-generators
按照PyG 官方的划分方式,目前存在如下几种数据集:
-
Homogeneous Datasets 同构数据集
-
Heterogeneous Datasets 异构数据集
-
Hypergraph Datasets 超图数据集
-
Synthetic Datasets 合成数据集
-
Graph Generators 图形生成器
-
Motif Generators 主题生成器
2. 图数据集的组成
在 PyG 中,数据集通常分为两个主文件夹: raw 和 processed 。
Raw Folder: 原始文件夹:此文件夹包含原始数据文件,格式通常为 .txt 或 .csv 。这些文件直接从数据源获取,无需任何特定于图神经网络模型的预处理。
Processed Folder:
已处理文件夹:此文件夹包含 .pt 文件形式的已处理数据,即 PyTorch 张量文件。这些文件中的数据已转换为适合在GNN中直接使用的格式。
2.1 raw folder
原始文件中, 常见的 .txt 文件有如下几种:
DS_A.txt:
Description: Contains the sparse (block diagonal) adjacency matrix for all graphs.
描述:包含所有图形的稀疏(块对角线)邻接矩阵。以邻接列表的形式描述节点之间的边,这对于构造图形的结构至关重要。
Function: Each line represents an edge in terms of two node identifiers (row, col), effectively encoding how nodes (proteins or parts thereof) are connected in each of the enzyme structures.
功能:每条线代表两个节点标识符(行、列)的一条边,有效地编码节点(蛋白质或其部分)在每个酶结构中的连接方式。
-
DS_graph_indicator.txt:
Description: Column vector of graph identifiers for all nodes.
说明:所有节点的图形标识符的列向量。
Function: Specifies which graph a node belongs to, allowing multiple graphs (individual enzyme structures) to be encoded within the same dataset. This is critical for distinguishing between separate enzyme samples in the dataset.
功能:指定节点所属的图,允许在同一数据集中编码多个图(单个酶结构)。这对于区分数据集中的单独酶样本至关重要。 指定每个节点所属的图,允许数据集中有多个图。 -
DS_graph_labels.txt:
Description: Contains class labels for each graph.
说明:包含每个图形的类标签。
Function: Each line assigns a class label (e.g., one of six EC top-level classes) to a graph, which is essential for supervised learning tasks where the model learns to predict the class of new, unseen graphs.
功能:每行为图形分配一个类标签(例如,六个 EC 顶级类之一),这对于监督学习任务至关重要,在监督学习任务中,模型学习预测新的、看不见的图形的类。
为整个图形分配标签,通常用于图形分类任务。
DS_node_labels.txt:
Description: Column vector of node labels.
描述:节点标签的列向量。
Function: Provides labels for individual nodes, which could represent specific types or parts of the enzyme. This is useful in tasks that require node classification or feature extraction at the node level.
功能:为单个节点提供标签,这些节点可以代表酶的特定类型或部分。这在需要节点级别节点分类或特征提取的任务中非常有用。
为每个节点提供标签,通常用于节点分类任务。
DS_edge_labels.txt (Optional):
Description: Labels for the edges.
描述:边缘的标签。
Function: Adds additional information to the edges, such as the type of interaction between protein components, which can be critical for understanding complex biological interactions.
功能:向边缘添加其他信息,例如蛋白质成分之间的相互作用类型,这对于理解复杂的生物相互作用至关重要。
如果节点之间的关系(例如,氨基酸或子结构之间的关系)具有分类差异,则这些标签为模型提供了额外的信息供学习。
DS_edge_attributes.txt (Optional):
Description: Attributes for edges.
描述:边的属性。
Function: Provides numerical or categorical attributes for each edge, enriching the edge data with extra details that can improve the performance of predictive models.
功能:为每条边提供数值或分类属性,使用可提高预测模型性能的额外详细信息丰富边数据。
此文件将包含有关节点之间连接的更多详细信息,可能包括距离测量、键合类型或交互强度,从而使用定量数据丰富图形表示。
-
DS_node_attributes.txt (Optional):
Description: Matrix of node attributes.
描述:节点属性矩阵。
Function: Each line contains a comma-separated attribute vector for a node, offering more detailed features per node that can enhance model learning capabilities, especially for more nuanced node-level predictions.
函数:每行都包含一个节点的逗号分隔属性向量,为每个节点提供更详细的特征,可以增强模型学习能力,特别是对于更细微的节点级预测。
在这里,节点被赋予属性,这些属性可能包括与蛋白质结构或功能相关的各种化学或物理特性,通过提供更丰富的特征集来增强模型预测。 -
DS_graph_attributes.txt (Optional):
Description: Regression values for all graphs.
说明:所有图形的回归值。
Function: Stores continuous or additional categorical attributes for graphs, useful in regression tasks or when additional graph-level features need to be considered for classification.
功能:存储图形的连续或附加分类属性,在回归任务或需要考虑其他图形级特征进行分类时很有用。
包含图形的回归值或其他属性,以及表征每种完整酶或蛋白质的潜在信息,例如物理特性或实验测量。
2.1.1 DS_node_labels.txt 和 DS_node_attributes.txt 之间的差别
在图形数据集中,喜欢 DS_node_labels.txt 和 DS_node_attributes.txt 的文件有不同的用途,特别关注如何存储和解释与图形节点相关的数据。以下是它们的典型用途和差异的详细细分:
- DS_node_labels.txt:
Purpose: 用途:此文件通常包含图形中每个节点的标签。标签对节点进行分类或分类,通常表示节点所属的特定类型或组。这些标签通常用于节点分类等任务,其中的目标可能是根据节点在图中的位置和连接来预测节点的标签。
Content:
内容:文件通常具有简单的结构,其中每行对应一个节点,并包含至少两个值:节点标识符及其标签。有时,标签是数字,代表不同的类别或类。
NodeID Label
1 0
2 1
3 0
- DS_node_attributes.txt:
Purpose:
用途:此文件用于存储每个节点的属性或功能。这些属性可以是节点的各种属性或特征,这些属性或特征不仅仅是分类标签,也可能是数字或基于文本的数据点。这些属性在图形分析中用于研究节点属性,在机器学习模型中用作预测的特征,或在网络可视化中用于表示其他节点信息。
Content: 内容:此文件的结构可能比标签文件更复杂。每行对应一个节点,并包含一系列属性,这些属性可能因节点而异。这些可能包括人口统计信息、度量值或与节点相关的任何其他数据。
NodeID Attribute1 Attribute2 Attribute3
1 34.5 New York 12
2 22.1 Los Angeles 9
3 28.7 Chicago 15
在实践中,主要用于基于图的学习任务, DS_node_labels.txt 其中节点分类是重点,利用标签数据作为目标结果。另一方面, DS_node_attributes.txt 提供更丰富的数据集,通常用于更详细的分析,包括基于特征的机器学习任务或更精细的网络研究,其中节点的属性可能会影响它们在图中的连通性或角色。
2.2 processed Folder
已处理文件夹:保存 .pt 文件,这些文件是图形数据的序列化版本,这些数据已预先处理并可供 GNN 模型立即使用。这些文件通常是通过读取原始数据、构建图形、节点和边的张量表示以及可能合并特征和标签来生成的。这些 .pt 文件通过减少模型训练和评估期间的加载和处理时间来简化数据的后续使用。
.pt 文件是在数据预处理步骤中生成的。这涉及读取原始图数据,可能将节点和边缘特征转换为数值张量,构造邻接矩阵或边缘索引张量,最后将这些张量保存为兼容 PyTorch 的格式 ( .pt )。此预处理阶段对于在 GNN 架构中高效加载和利用图形数据至关重要。
处理后,这些 .pt 文件可以很容易地加载到 PyG 数据集和模型中,从而大大减少了模型训练期间的开销,因为不需要动态预处理数据。这导致了更快的实验周期和更简化的GNN应用开发过程。
总之,PyG 带有一个用于处理图形数据的复杂生态系统,其中 .txt 文件和其他格式用作原始输入,这些输入被转换为可高效加载 .pt 的文件以供 GNN 开发。此设置增强了在各种应用程序中开发图神经网络解决方案的可用性和效率。
2.3 数据集读取示例
ENZYMES数据集特别有利于生物信息学的研究和应用,特别是在蛋白质结构和功能预测方面。通过利用基于图形的机器学习模型,研究人员可以开发以下算法:
根据其结构信息将酶分类为EC顶级类别。
通过分析节点和边缘属性来预测蛋白质或酶(如活性位点)的特性。
Study protein structure-function relationships by correlating graph representations with enzyme classifications and attributes.
通过将图形表示与酶分类和属性相关联来研究蛋白质结构-功能关系。
ENZYMES 数据集的详细组织有助于广泛的计算生物学任务,尤其是那些利用图神经网络 (GNN) 或其他图表示学习方法的任务。通过以图形形式捕获蛋白质的复杂、错综复杂的结构,该数据集为酶分类和蛋白质生物信息学的高级分析和预测开辟了途径。
提供的代码是一个 Python 函数,它从一系列文本文件中读取图形数据,并使用 NetworkX 库构造图形对象列表。
它是为处理格式类似于多特蒙德工业大学图核数据集页面上的基准数据集的数据而量身定制的。下面是详细的逐行说明和提到的特定数据集文件的作用:
def read_graphfile(datadir, dataname, max_nodes=None):
''' Read data from https://ls11-www.cs.tu-dortmund.de/staff/morris/graphkerneldatasets
graph index starts with 1 in file
Returns:
List of networkx objects with graph and node labels
'''
prefix = os.path.join(datadir, dataname, dataname)
filename_graph_indic = prefix + '_graph_indicator.txt'
# index of graphs that a given node belongs to
graph_indic={}
with open(filename_graph_indic) as f:
i=1
for line in f:
line=line.strip("\n")
graph_indic[i]=int(line)
i+=1
filename_nodes=prefix + '_node_labels.txt'
node_labels=[]
try:
with open(filename_nodes) as f:
for line in f:
line=line.strip("\n")
node_labels+=[int(line) - 1]
num_unique_node_labels = max(node_labels) + 1
except IOError:
print('No node labels')
filename_node_attrs=prefix + '_node_attributes.txt'
node_attrs=[]
try:
with open(filename_node_attrs) as f:
for line in f:
line = line.strip("\s\n")
attrs = [float(attr) for attr in re.split("[,\s]+", line) if not attr == '']
node_attrs.append(np.array(attrs))
except IOError:
print('No node attributes')
label_has_zero = False
filename_graphs=prefix + '_graph_labels.txt'
graph_labels=[]
# assume that all graph labels appear in the dataset
#(set of labels don't have to be consecutive)
label_vals = []
with open(filename_graphs) as f:
for line in f:
line=line.strip("\n")
val = int(line)
#if val == 0:
# label_has_zero = True
if val not in label_vals:
label_vals.append(val)
graph_labels.append(val)
#graph_labels = np.array(graph_labels)
label_map_to_int = {val: i for i, val in enumerate(label_vals)}
graph_labels = np.array([label_map_to_int[l] for l in graph_labels])
#if label_has_zero:
# graph_labels += 1
filename_adj=prefix + '_A.txt'
adj_list={i:[] for i in range(1,len(graph_labels)+1)}
index_graph={i:[] for i in range(1,len(graph_labels)+1)}
num_edges = 0
with open(filename_adj) as f:
for line in f:
line=line.strip("\n").split(",")
e0,e1=(int(line[0].strip(" ")),int(line[1].strip(" ")))
adj_list[graph_indic[e0]].append((e0,e1))
index_graph[graph_indic[e0]]+=[e0,e1]
num_edges += 1
for k in index_graph.keys():
index_graph[k]=[u-1 for u in set(index_graph[k])]
graphs=[]
for i in range(1,1+len(adj_list)):
# indexed from 1 here
G=nx.from_edgelist(adj_list[i])
if max_nodes is not None and G.number_of_nodes() > max_nodes:
continue
# add features and labels
G.graph['label'] = graph_labels[i-1]
for u in util.node_iter(G):
if len(node_labels) > 0:
node_label_one_hot = [0] * num_unique_node_labels
node_label = node_labels[u-1]
node_label_one_hot[node_label] = 1
util.node_dict(G)[u]['label'] = node_label_one_hot
if len(node_attrs) > 0:
util.node_dict(G)[u]['feat'] = node_attrs[u-1]
if len(node_attrs) > 0:
G.graph['feat_dim'] = node_attrs[0].shape[0]
# relabeling
mapping={}
it=0
for n in util.node_iter(G):
mapping[n]=it
it+=1
# indexed from 0
graphs.append(nx.relabel_nodes(G, mapping))
return graphs
2.3.1 代码分解
Function read_graphfile 功能 read_graphfile
Inputs:
输入: datadir (目录路径)、 dataname (数据集名称)、 max_nodes (可选,将图形限制为最大节点数)。
Outputs:
输出:具有节点标签和功能以及图形标签的 NetworkX 图形对象列表。
代码分解
Prefix Setup: 前缀设置:将目录和数据集名称组合在一起,为所有文件创建基本路径。
-
Graph Indicator Reading: 图表指标读数:
打开 _graph_indicator.txt ,将节点链接到特定图形(每行指示节点属于哪个图形)。
Constructs a dictionary graph_indic where keys are node indices and values are graph identifiers.
构造一个字典, graph_indic 其中键是节点索引,值是图形标识符。
Node Labels Reading: 节点标签读取: -
Opens the _node_labels.txt,
打开 _node_labels.txt ,其中每行对应一个节点的标签。
构造一个存储这些标签的列表 node_labels ,并针对零索引进行调整。
Node Attributes Reading: 节点属性读取: -
Opens the _node_attributes.txt,
打开 _node_attributes.txt ,其中每行都包含节点的属性,用逗号或空格分隔。
解析这些属性并将其转换为 numpy 数组,并将它们存储在 node_attrs .
Graph Labels Reading: 图形标签读取:
- Opens the _graph_labels.txt,
打开 _graph_labels.txt ,其中每行都包含图形的标签。
构造一个列表 graph_labels ,存储这些标签,并在必要时创建到整数索引( label_map_to_int )的映射。
-
Adjacency List Construction:
邻接列表构造: -
Opens the _A.txt,
打开 _A.txt ,其中列出了节点之间的边。每条线包含两个形成边的节点索引。
使用 将 graph_indic 每条边放置在正确图形的邻接列表 ( adj_list ) 中。 -
Graph Construction: 图形构造:
循环访问邻接列表以创建 NetworkX 图形对象。
如果 max_nodes 提供,则筛选出超过此大小的图形。
将节点标签作为单热编码要素和节点属性添加到图中的每个节点。
Optionally sets a feature dimension attribute for each graph.
(可选)为每个图形设置要素维度属性。 -
Node Relabeling: 节点重新标记:
将每个图中的节点从从 1 开始的索引重新标记到从 0 开始的索引,以确保在 Python 中处理的一致性。
2.3.2 文件小结
-
_graph.txt :通常,此文件将包含显式图形结构或与图形相关的元数据。在某些数据集中,它可能类似于枚举图形组件的文件。
-
_node_labels.txt :存储每个节点的标签,在节点级别的监督学习任务中很有用。
-
_node_attributes.txt :包含节点的连续或分类属性,丰富机器学习模型的节点特征。
-
_graph_labels.txt :为整个图形提供标签,这对于图形分类等任务至关重要。
-
_A.txt :列出数据集中的边,这对于构建图形结构至关重要。
3. 常见图数据集
-
KarateClub:数据为无向图;
源于论文 An Information Flow Model for Conflict and Fission in Small Groups -
TUDataset:数据为无向图;
包含 58 个基础的分类数据集几何,如 “IMDB-BINARY”,“PROTEINS”等;
来源于TU Dortmund University -
Plantoid:数据都为无向图;
引用网络数据集,包括“Cora”,“CiteSeer”,和 “PubMed”;
来源于论文Revisiting Semi-Supervised Learning with Graph Embeddings。
节点代表文档,边代表引用关系。 -
CoraFull:数据为无向图;
完整的“Cora”引用网络数据集;
来源于论文Deep Gaussian Embedding of Graphs: Unsupervised Inductive Learning via Ranking。
节点代表文档,边代表引用关系。【论文的引用关系】 -
Coauthor:数据都为无向图;
共同作者网络数据集,包括“CS” 和 “Physics”;
来源于论文Pitfalls of Graph Neural Network Evaluation。
节点代表作者,若是共同作者则被边连接。
学习任务是将作者映射到各自的研究领域中。 -
Amazon:数据都为无向图;
亚马逊网络数据集,包括“computers” 和 “Photo”,
来源于论文Pitfalls of Graph Neural Network Evaluation。
节点代表货物,边代表两种货物经常被同时购买。
学习任务是将货物映射到各自的种类里。 -
PPI:数据都为无向图;
蛋白质-蛋白质反应网络;
来源于论文Predicting multicellular function through multi-layer tissue networks -
Entities:数据都为无向图;
关系实体网络,包括“AIFB”,“MUTAG”,“BGS”,“AM”;
来源于论文Modeling Relational Data with Graph Convolutional Networks -
BitcoinOTC:数据都为有向图;
包括138 个“who-trusts-whom”网络;
来源于论文EvolveGCN: Evolving Graph Convolutional Networks for Dynamic Graphs;
数据链接为Bitcoin OTC trust weighted signed network
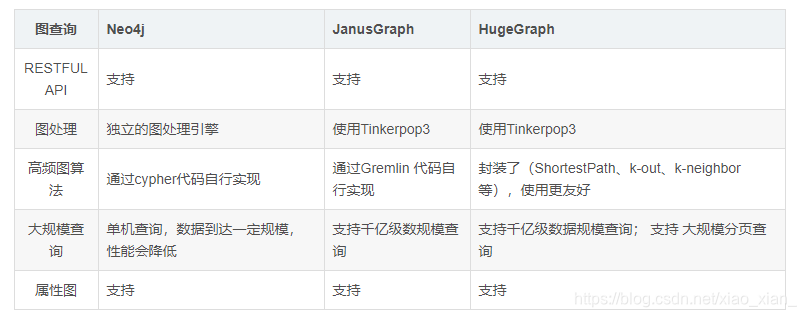
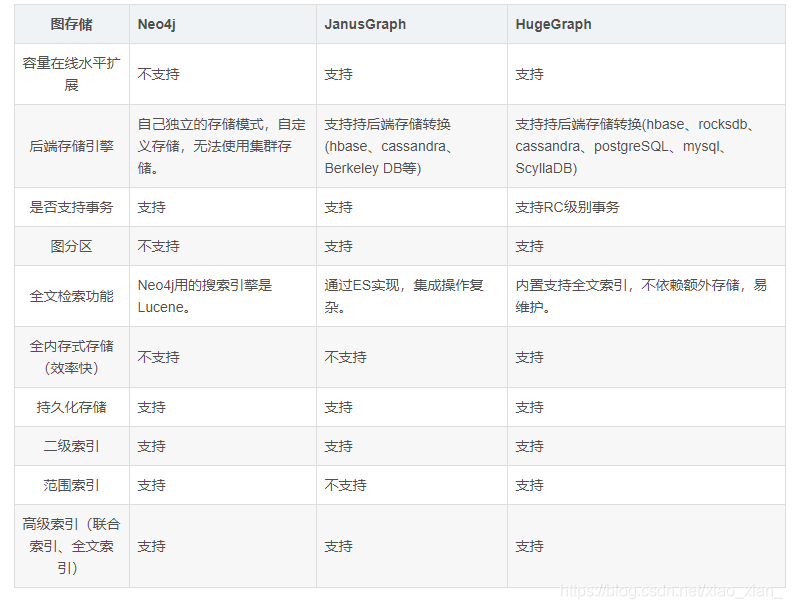
》neo4j不是开源的,社区版不能上线,也不支持分布式和多机备份
janusgraph开源,支持分布式,而且可以多机备份






















 2793
2793











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








