文章概述
- 关键词:反应数据库
- 简介:Rhea (http://www.rhea-db.org) 是一个全面且非冗余的资源,包含超过 11,000 个专家策划的生化反应,使用来自 ChEBI 本体的化学实体来表示反应参与者。Rhea 最初被设计为 UniProt 知识库 (UniProtKB) 的注释词汇表,旨在为包括 ChEBI 和 MetaboLights 在内的一系列其他核心知识库和数据存储库提供反应数据。
- 使用 Rhea 描述酶功能的资源包括 IntEnz (4)、酶门户 (5) 和机制和催化位点图谱 (M-CSA) (6),以及 MetaNetX (7) 和 BiGG (8) 等基因组尺度代谢模型的平台。Rhea 目前还通过 IUBMB 的酶分类链接到 UniProtKB (9)。使用Rhea反应数据的代谢物和代谢组学资源包括化学本体ChEBI、用于脂质生物学的瑞士Lipids知识库(10)和代谢组学存储库MetaboLights(11)。Rhea 还与其他反应资源(例如 KEGG (12)、MetaCyc (13) 和 Reactome (14))的链接,每个反应资源也提供数千个独特的反应。
- 内容:我们描述了 Rhea 的最新发展,重点关注 Rhea 反应数据和 SPARQL 端点 (https://sparql.rhea-db.org/sparql) 的新资源描述框架表示,提供对它的访问。我们演示了如何将Rhea SPARQL端点和其他SPARQL端点(如UniProt)相结合的联邦查询如何提供改进的代谢物注释,并支持将代谢组通过蛋白质组链接到转录组和基因组的综合分析。这些发展将显着提升 Rhea 作为连接化学和生物学的一种手段的效用,以便更全面地了解生物系统及其在健康和疾病中的作用。
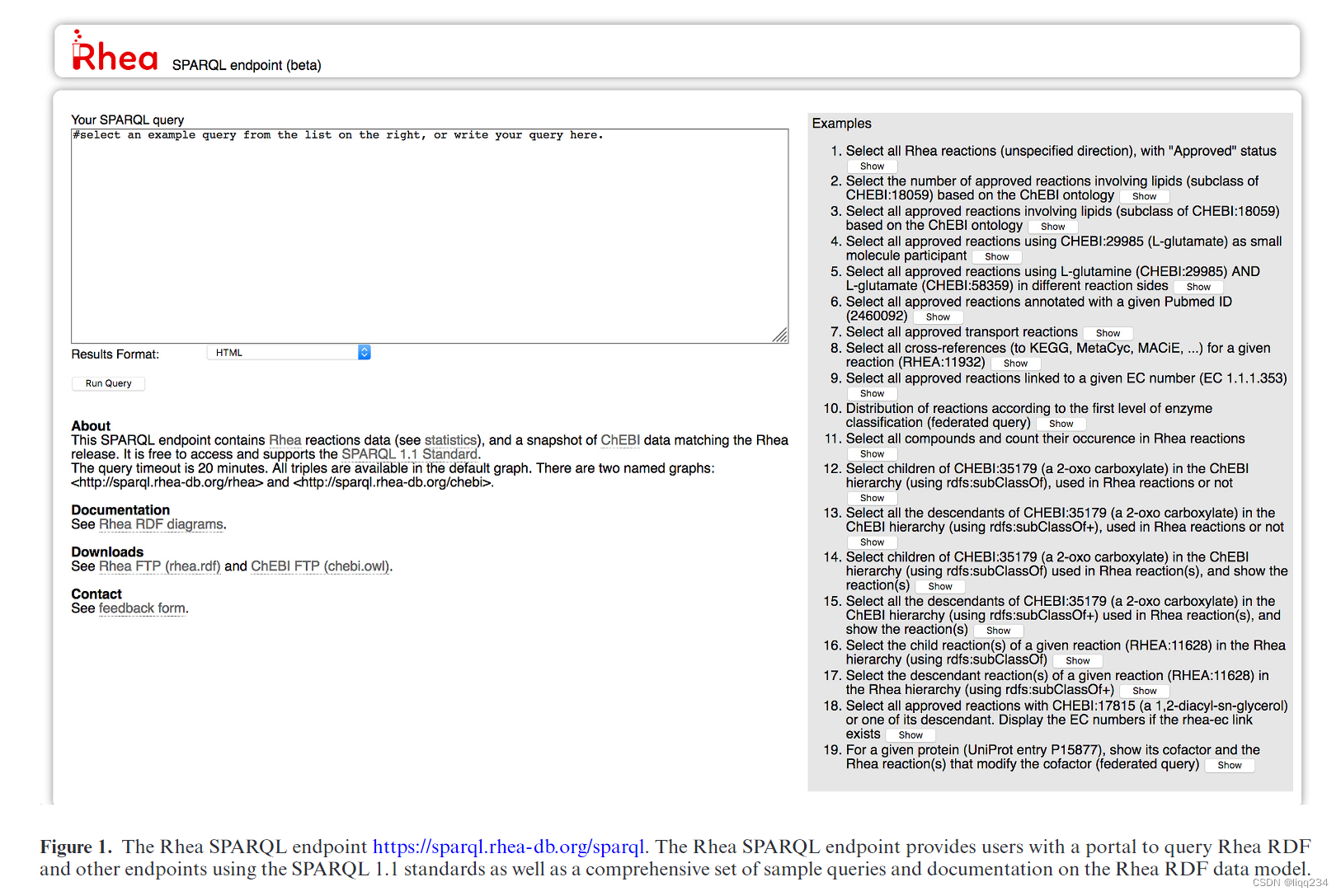
结果
用户可以在 Rhea SPARQL 端点 https://sparql.rhea-db.org/sparql(见图 1)上使用 SPARQL(SPARQL 协议和 RDF 查询语言)查询 Rhea RDF 数据,它支持一系列复杂和联合查询,这些查询合并来自其他 SPARQL 端点的数据。我们在网站 https://www.rhea-db.org/rhea rdf 文档提供了对 Rheea 数据模型的详细描述。pdf 并邀请感兴趣的读者查阅那里的文档。Rhea SPARQL 端点使用 Virtuoso 软件 (https://virtuoso.openlinksw.com/),并托管在 SIB 瑞士生物信息学研究所高性能计算 (https://www.viti-it.ch/) 的 Vital-IT 中心。Rhea RDF 数据也可用于在 ftp://ftp.ebi.ac.uk/pub/databases/rhea/rdf/ 序列化为 RDF/XML。
- 查询1演示了使用 Rhea 为特定感兴趣的生物体构建酶和反应网络(在这种情况下,大肠杆菌菌株 K12),返回 UniProtKB 蛋白列表及其催化的 Rhea 反应。

-查询2演示了使用 Rhea 来整合代谢组、蛋白质组、转录组和基因组的知识;它返回映射到智人代谢它们的酶的相关基因和转录本 (Ensembl) 和蛋白质序列 (UniProtKB/Swiss-Prot) 的代谢物 (ChEBI) 的标识符列表。这种联合查询提供了类似于 MetaBridge (22) 等专用 ID 映射工具的功能。

- 查询3展示了如何将 ChEBI 分类与来自 Rhea 和 UniProtKB 的数据相结合,以识别特定感兴趣代谢物的候选酶。所讨论的代谢物是 ṣ1 , 1993;7 -dafa慢性酸 (CHEB:83137),这是 DAF-12 的有效配体,它调节秀丽隐杆线虫衰老 (27)。ⅰ1, ṣ7 - 达法慢性酸在任何 Rhea 反应中都不具有特征,并且与任何已知的酶无关。查询使用 ChEBI 父/子本体关系来检索 Ô1 、ṣ7 -dafa慢性酸的所有父 ChEBI 类,追溯到 ChEBI 本体的根,然后搜索候选酶和对这些父类的反应。该查询有效地将UniProtKB/SwissProt中实验表征的代谢物类的注释扩展到当前相同化学类的未注释成员。


其他Rhea进入的模式
除了现在提供 Rhea SPARQL 端点外,我们还继续在 www.rhea-db.org 上以我们之前的出版物 (3) 中描述的所有访问模式(交互式搜索、编程访问和数据下载)和数据格式。
Rhea内容
Rhea 继续显着增加,因为我们的最终报告通过专家管理 ChEBI 中的新化学实体和同行评审文献中的反应(详见 http://www.rhea-db.org/statistics)。Rhea 目前(2018 年 7 月 13 日第 96 版)描述了 11173 个独特的反应,涉及 9916 个独特的反应参与者,并引用了 12 611 个独特的文献参考(PubMed 标识符)。这代表了自我们最后一次发表以来约 1900 个独特的反应、1800 个独特的反应参与者和 3700 个文献参考的增加 (3)(描述了 2016 年 7 月 30 日的释放 75 日)。
讨论
- 我们已经展示了将Rhea反应数据与其他SPARQL端点(如UniProt)的数据相结合的联邦SPARQL查询如何促进一系列数据集成和数据挖掘任务。这些包括基因组规模代谢反应网络的生成和候选酶的识别,它们是代谢建模和工程等系统生物学应用中的常见用例,以及基因组、转录组、蛋白质组和代谢组数据的整合,具有广泛的实用性,包括个性化健康和医学领域。
- 我们描述的联邦查询目前利用Rhea反应和IUBMB酶分类之间的映射来链接Rhea和UniProtKB。在不久的将来,UniProt将Rhea作为UniProtKB中酶的注释词汇,UniProt策展人从厦门大学用户于2022年5月19日下载于厦门大学用户从https://academic.oup.com/nar/article/47/D1/D596/5112989下载。47数据库问题将直接将Rhea反应链接到UniProtKB/Swiss-Prot记录作为其正常管理工作流程的一部分。这将显着增加 UniProtKB 中酶注释的覆盖和特异性,增强 UniProtKB 和 Rhea 在“组学数据集成”方面的效用,并为将蛋白质序列和功能与化学结构数据相结合的新搜索和分析能力提供支持。





















 81
81











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








