深度学习这一方法越来越多地用于医疗领域。卷积神经网络(CNN)作为其中代表。目前已有一些研究将深度学习技术用于神经胶质瘤MRI影像分析。
1.MSCNN
Akkus等人[1]使用MRI图像中T2和T1加权图像这两种常用的类型作为1p/19q联合缺失分类算法的输入。其分类算法包括多模态图像配准、数据归一化,数据增强和肿瘤分割等预处理步骤,之后利用分割后的图像训练了一个多尺度CNN来预测1p/19q的状态。最终实验结果表明,CNN预测1p/19q联合缺失有良好效果。

Figure 1.多尺度 CNN 架构的流程图。蓝色框是输入图像。黄色框是卷积层。绿色框是整流线性单元(RELU),激活。红框是最大池化层。紫色框是全连接层加上一个 softmax 二元分类器。青色圆圈显示输出标签。
MSCNN的优点:
1、 提取多尺度特征、单列网络参数少且易于训练。
MSCNN的缺点:
1、 需要大样本量,训练最好要GPU
2、 不能保证小尺度目标的高召回。
2. AlexNet
Fukuma等人[2]利用了AlexNet18卷积网络对IDH突变进行了分类,同时还评估了在模型中加入临床信息对预测的影响。最终结果为IDH突变的预测精度可达到69.6%。
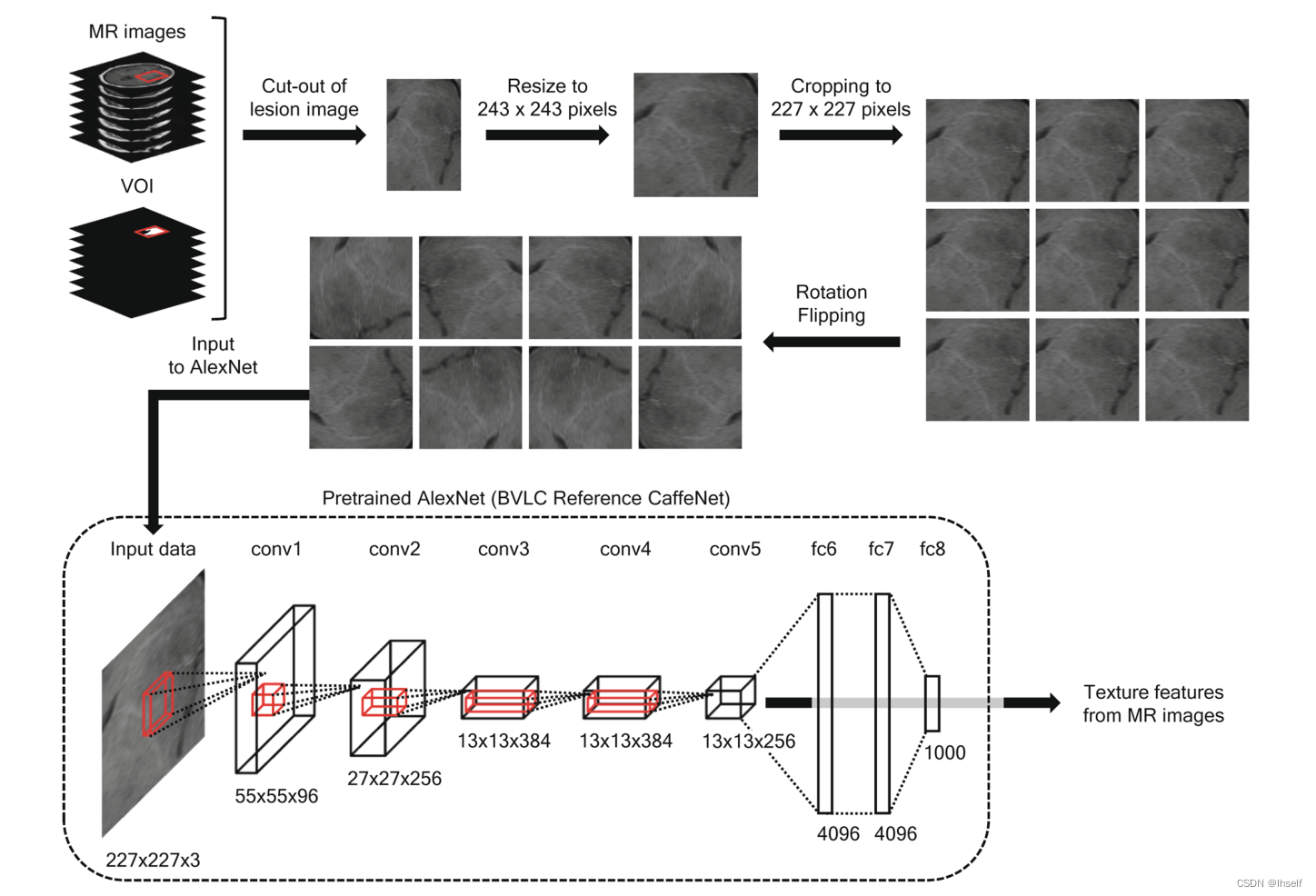
Figure 2. AlexNet 的特征提取。使用 VOI 剪切病变图像并将其大小调整为 243 × 243 像素。图像被裁剪为 227 × 227 像素,偏移为 ±8 像素,并旋转/翻转以进行数据增强。将增强后的图像输入到预训练的 AlexNet 以获取用于分类的纹理特征。
AlexNet的优点
使用sigmoid函数计算量大,改为Relu函数后整个计算量减少很多。会使一部分神经元输出为零,缓解了过拟合现象
AlexNet的缺点
会造成梯度弥散问题,无法完成深层网络训练。
3. CRNN
Lichy等人[3]使用MRI图像来预测MGMT甲基化状态。其方法使用了一个双向卷积递归神经网络(CRNN)
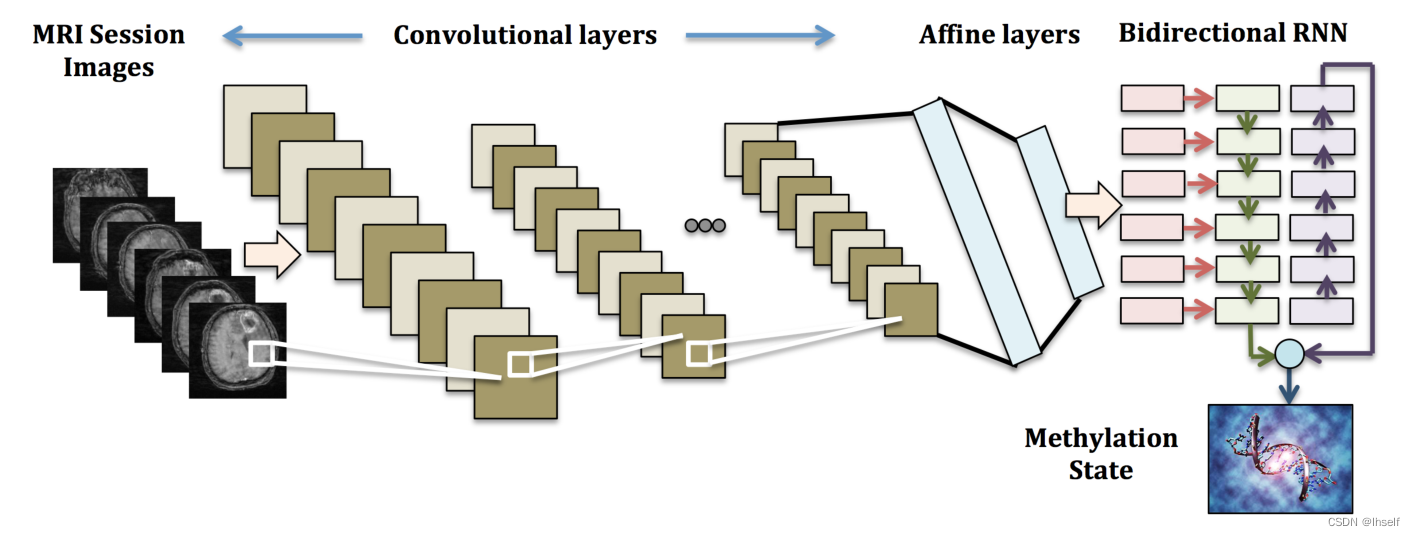
Figure 3. CRNN 架构概述。结合 CNN 和 RNN 从 MRI 扫描图像中预测甲基化状态。
CRNN的优点:
容易训练,模型容易收敛,且鲁棒性较高。
易于部署,模型结构简单
CRNN的缺点:
识别准确率相对较低。
4. ResNet
Korfiatis等人[4]比较了三种不同的残余深度神经网络(ResNet)架构,以评估它们预测MGMT甲基化状态的能力,并且无需进行肿瘤MR图像的分割。最终实验发现ResNet50层体系结构是性能最佳的模型,测试集的准确度达到94.90%。

Figure 4. ResNet50模型的视觉描述。Conv、pool、和fc分别代表卷积层、池层和全连接层。使用的池大小为2(用/2表示,例如, 256/2表示使用了256个过滤器,池层的大小为2)。所以第一个框(7×7conv,64)意味着卷积核的大小是7×7和64个滤波器。然后,我们明确地将下面的层描述为2×2池层,但在本图的其他地方,我们在显示该层的框中使用了缩写/2。实线(-)表示标识,虚线(----)表示交叉剩余加权连接
ResNet优点:
使前馈/反馈传播算法顺利进行,结构更加简单;恒等映射增加基本不会降低网络的性能。
ResNet缺点:
训练时间长,对设备性能要去较高。
5. MTCNN
文献[5]中的作者提出一个多任务深度学习框架,利用四种序列(T1,T1c,T2,Flair)的MRI胶质瘤图像,分别作为5个并行神经网络的输入数据,同时预测该患者胶质瘤的四种基因分型(MGMT,IDH,1p/19q,TERT)以及患者的生存期。

Figure 5. 用于基因型和 OS 预测的多任务 CNN 网络结构概述。有五项任务:四项用于肿瘤基因型预测,一项用于 OS 预测。在 OS 预测的帮助下,可以更好地学习与肿瘤基因型相关的特征表示,反之亦然。
MTCNN的优点:
准确率高
MTCNN缺点:
速度慢。第一,生成图片金字塔慢;第二,每种尺度的图片都需要输入进模型,相当于执行了多次的模型推断流程。
6. U-net
U-net [6] 结合低分辨率信息进行目标分类,结合高分辨率信息进行精确识别和定位,非常适合医学图像分类。所以本课题研究采用Unet网络模型进行研究。

Figure 6. U-net 架构(例如最低分辨率的 32x32 像素)。每个蓝色框对应一个多通道特征图。通道数显示在框的顶部。 x-y 尺寸位于框的左下边缘。白框代表复制的特征图。箭头表示不同的操作。

图7 Unet网络模型
我们采用的Unet网络模型如图7所示。
1)下采样:图1的前半部分(编码器层),红色部分,max-pooling实现。
2)上采样:图1的后半部分(解码器层),黄色部分,反卷积实现。
3)最后通过全连接层的分类实现。
U-Net与其他常见的分割网络(如FCN)有一点非常不同的地方:
U-net采用了完全不同的特征融合方式:拼接(Concat),U-Net采用将特征在channel维度拼接在一起,形成更厚的特征。而FCN融合时使用的对应点相加,并不形成更厚的特征。
所以语义分割网络在特征融合时有两种办法:
FCN式的对应点相加。
U-Net式的channel维度拼接融合。
除了上述新颖的特征融合方式,U-Net还有以下优点:
5个pooling layer实现了网络对图像特征的多尺度特征识别。
上采样部分会融合特征提取部分的输出,这样做实际上是将多尺度特征融合在了一起,以最后一个上采样为例,它的特征既来自第一个卷积block的输出(同尺度特征),也来自上采样的输出(大尺度特征),这样的连接是贯穿整个网络的,你可以看到上图的网络中有四次融合过程,相对应的FCN网络只在最后一层进行融合。
U-Net也有不足:
该网络运行效率很慢。对于每个邻域,网络都要运行一次,且对于邻域重叠部分,网络会进行重复运算。
Reference:
[1] Zeynettin A, Issa A, Jiri S, Predicting 1p19q Chromosomal Deletion of Low-Grade Gliomas from MR Images using Deep Learning. J Digit Imaging. 2016; 30(4):469–476.
[2] Fukuma R, Yanagisawa T., Kinoshita M, et al. Prediction Of IDH And TERT Promoter Mutations In Low-Grade Glioma From Magnetic Resonance Images Using A Convolutional Neural Network. Sci Rep 9. 2019; 20311.
[3] Lichy H, Maulik K. MRI To MGMT: Predicting Methylation Status In Glioblastoma Patients Using Convolutional Recurrent Neural Networks. Pac Symp Biocomput. 2018; 23:331-342.
[4] Korfiatis P, Kline T, Lachance D, et al. Residual Deep Convolutional Neural Network Predicts MGMT Methylation Status. J Digit Imaging. 2017; 30(5):622-628.
[5] Tang Z, Xu Y, Jin L, Aibaidula A, Lu J, Jiao Z, Wu J, Zhang H and Shen D. Deep Learning of Imaging Phenotype and Genotype for Predicting Overall Survival Time of Glioblastoma Patients. IEEE Transactions on Medical Imaging. 2020; 1-10.
[6] Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation[C]//International Conference on Medical image computing and computer-assisted intervention. Springer, Cham, 2015: 234-241.











 本文综述了深度学习技术,如多尺度CNN、AlexNet、CRNN、ResNet、MTCNN和U-Net在医疗影像分析中的应用,特别是在神经胶质瘤MRI影像上的表现。这些模型在肿瘤分类、基因型预测和生存期预测等方面显示出潜力,但也存在样本需求大、训练时间长、计算资源要求高等挑战。
本文综述了深度学习技术,如多尺度CNN、AlexNet、CRNN、ResNet、MTCNN和U-Net在医疗影像分析中的应用,特别是在神经胶质瘤MRI影像上的表现。这些模型在肿瘤分类、基因型预测和生存期预测等方面显示出潜力,但也存在样本需求大、训练时间长、计算资源要求高等挑战。
















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








