目录
quality_summary.tsv(三个模块的综合输出)
这里的virus序列可能是游离病毒的,也可能是前病毒的一部分
checkv对provirus序列切割后的产物,provirus region / contig sequence
checkv会进一步鉴定所有序列中的host污染及位置信息,关于估计污染的详细概述
1.checkv运行的的步骤
A: Remove host contamination
B:Estime genome completeness
C:Predict closed genomes
D:Summarieze quality
2.下载与安装checkv
checkv基于conda的环境
conda install -c conda-forge -c bioconda checkv
#数据库下载(自动)
checkv download_database ./
#数据库下载(手动)
wget https://portal.nersc.gov/CheckV/checkv-db-v1.0.tar.gz
tar -zxvf checkv-db-v1.0.tar.gz
export CHECKVDB=/path/to/checkv-db-v1.03.checkv的基本使用
一步法
checkv end_to_end input_file.fna output_fiel -t -16分步法
checkv contamination input_file.fna output_directory -t 16
checkv completeness input_file.fna output_directory -t 16
checkv complete_genomes input_file.fna output_directory
checkv quality_summary input_file.fna output_directory4.checkv的运行的过程
CheckV v0.8.1: contamination
[1/8] Reading database info...
[2/8] Reading genome info...
[3/8] Calling genes with Prodigal...
[4/8] Reading gene info...
[5/8] Running hmmsearch...
[6/8] Annotating genes...
[7/8] Identifying host regions...
[8/8] Writing results...
Run time: 128.65 seconds
Peak mem: 0.12 GB
CheckV v0.8.1: completeness
[1/8] Skipping gene calling...
[2/8] Initializing queries and database...
[3/8] Running DIAMOND blastp search...
[4/8] Computing AAI...
[5/8] Running AAI based completeness estimation...
[6/8] Running HMM based completeness estimation...
[7/8] Determining genome copy number...
[8/8] Writing results...
Run time: 28.43 seconds
Peak mem: 0.25 GB
CheckV v0.8.1: complete_genomes
[1/7] Reading input sequences...
[2/7] Finding complete proviruses...
[3/7] Finding direct/inverted terminal repeats...
[4/7] Filtering terminal repeats...
[5/7] Checking genome for completeness...
[6/7] Checking genome for large duplications...
[7/7] Writing results...
Run time: 0.13 seconds
Peak mem: 0.25 GB
CheckV v0.8.1: quality_summary
[1/6] Reading input sequences...
[2/6] Reading results from contamination module...
[3/6] Reading results from completeness module...
[4/6] Reading results from complete genomes module...
[5/6] Classifying contigs into quality tiers...
[6/6] Writing results...
Run time: 0.04 seconds
Peak mem: 0.25 GB
5.checkv的结果解读
![]()
quality_summary.tsv(三个模块的综合输出)
provirus/virus、质量、完整度

virus.fna
这里的virus序列可能是游离病毒的,也可能是前病毒的一部分
provirus.fna
checkv对provirus序列切割后的产物,provirus region / contig sequence
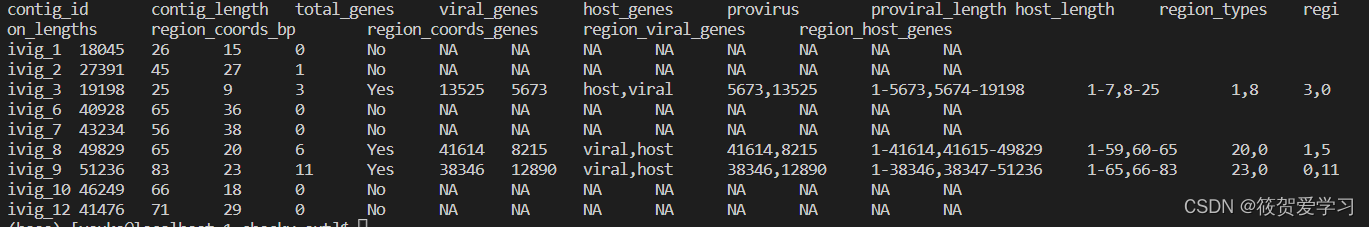
contamination.tsv
checkv会进一步鉴定所有序列中的host污染及位置信息,关于估计污染的详细概述
tmp/proteins.faa
病毒蛋白序列信息
completeness.tsv
关于估计完整性的详细概述

complete_genomes.tsv
已确定的假定完整基因组的详细概述

























 1486
1486

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








