表达差异矩阵
当产生的表达差异矩阵后,做后续的分析:
1.KEGG是通路富集分析
2.GO是功能富集分析,包括BP分析,CC分析以及MF分析。GO数据库,全称是Gene Ontology(基因本体),他们把基因的功能分成了三个部分分别是:细胞组分(cellular component, CC)、分子功能(molecular function, MF)、生物过程(biological process, BP)。
传统KEGG(通路富集分析)和GO(功能富集)分析时,如果富集到的同一通路下,既有上调差异基因,也有下调差异基因,那么这条通路总体的表现形式究竟是怎样?是被抑制还是激活?或者更直观点说,这条通路下的基因表达水平在实验处理后是上升了呢,还是下降了呢?
传统的富集分析,针对总体的差异基因,不区分哪些差异基因是上调还是下调。
GSEA
3.GSEA分析是 基因集富集分析,由Broad Institute研究所提出的一种富集方法。对应的基因集数据库MSigdb。
GSEA的输入是一个基因表达量矩阵,其中的样本分成了A和B两组,首先对所有基因进行排序,简单理解就是根据处理后的差异倍数值进行从大到小排序, 用来表示基因在两组间的表达量变化趋势。排序之后的基因列表其顶部可看做是上调的差异基因,其底部是下调的差异基因。
结果分析:
“MUT vs WT”的差异gene集(MUT为实验组,WT为对照组)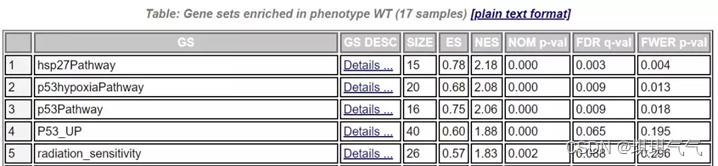
GS:基因集(通路)的名字。
SIZE:代表该基因集(通路)下的基因总数。
ES:代表Enrichment score,NES代表归一化后的Enrichment score。
NOM p-val:代表p值,表征富集结果的可信度。
FDR q-val`代表q值, 是多重假设检验矫正后的p值,注意GSEA采用pvalue < 5%, qvalue < 25% 对结果进行过滤。
对于某个基因集下(通路里)的每个基因给出了详细的统计信息,如下图

RANK IN GENE LIST:代表该基因在排序中的位置。
RANK METRIC SCORE:代表该基因排序量的值,即:处理后的foldchange值。
RUNNIG ES:代表累计的Enrichment score。
CORE ENRICHMENT:代表是否属于核心基因,即对该基因集的Enerchment score做出了主要贡献的基因。
上图表格中的数据对应下面这张图
图分为3部分,如下:
第一部分:最顶部的绿色折线为基因Enrichment Score的折线图。纵轴为对应的Running ES, 在折线图中有个峰值,该峰值就是这个基因集的Enrichemnt score,峰值之前的基因就是该基因集下的核心基因。横轴代表此基因集下的每个基因,对应第二部分类似条形码的竖线。
第二部分:类似条形码的部分,为Hits,每条竖线对应该基因集下的一个基因。
第三部分:为所有基因的rank值分布图,纵坐标为ranked list metric,即该基因排序量的值,可理解为“公式化处理后的foldchange值”。







 本文讨论了在基因表达差异研究中,KEGG通路富集分析和GO功能富集分析遇到的问题,特别是当上调和下调基因同时存在于同一通路时。GSEA基因集富集分析被引入,它考虑了基因上调/下调的详细信息,通过EnrichmentScore评估基因集在处理组中的显著性。文中还介绍了GSEA的输入、排序过程和关键指标如ES、NOMp-val和FDRq-val的解释。
本文讨论了在基因表达差异研究中,KEGG通路富集分析和GO功能富集分析遇到的问题,特别是当上调和下调基因同时存在于同一通路时。GSEA基因集富集分析被引入,它考虑了基因上调/下调的详细信息,通过EnrichmentScore评估基因集在处理组中的显著性。文中还介绍了GSEA的输入、排序过程和关键指标如ES、NOMp-val和FDRq-val的解释。
















 1981
1981

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








