文章目录
一、数据关联简介
- The MOT problem can be viewed as a
data association problemwhere the aim is to associate detections across frames in a video sequence- 为什么不使用前后两帧的检测结果直接进行匹配呢?1、速度快了可能误匹配,2、存在漏检的情况(此时 kf 还能继续预测,只用检测则丢失了 track 当前帧的信息)

import numpy as np
from scipy.optimize import linear_sum_assignment
"""在这里我们对检测框和跟踪框进行匹配,整个流程是遍历检测框和跟踪框,
并进行匹配,匹配成功的将其保留,未成功的将其删除"""
def associate_detections_to_trackers(detections, trackers, iou_threshold=0.3):
"""
将检测框bbox与卡尔曼滤波器的跟踪框进行关联匹配
:param detections:检测框
:param trackers:跟踪框,即跟踪目标
:param iou_threshold:IOU阈值
:return:跟踪成功目标的矩阵:matchs
新增目标的矩阵:unmatched_detections
跟踪失败即离开画面的目标矩阵:unmatched_trackers
"""
# 跟踪目标数量为0,直接构造结果
if (len(trackers) == 0) or (len(detections) == 0):
return np.empty((0, 2), dtype=int), np.arange(len(detections)), np.empty((0, 5), dtype=int)
# iou 不支持数组计算。逐个计算两两间的交并比,调用 linear_assignment 进行匹配
iou_matrix = np.zeros((len(detections), len(trackers)), dtype=np.float32)
# 遍历目标检测的 bbox 集合,每个检测框的标识为 d
for d, det in enumerate(detections):
# 遍历跟踪框(卡尔曼滤波器预测)bbox 集合,每个跟踪框标识为 t
for t, trk in enumerate(trackers):
iou_matrix[d, t] = iou(det, trk)
# 通过匈牙利算法将跟踪框和检测框以 [[d,t]...] 的二维矩阵的形式存储在 match_indices 中
result = linear_sum_assignment(-iou_matrix)
matched_indices = np.array(list(zip(*result)))
# 记录未匹配的检测框及跟踪框
# 未匹配的检测框放入 unmatched_detections 中,表示有新的目标进入画面,要新增跟踪器跟踪目标
unmatched_detections = []
for d, det in enumerate(detections):
if d not in matched_indices[:, 0]:
unmatched_detections.append(d)
# 未匹配的跟踪框放入 unmatched_trackers 中,表示目标离开之前的画面,应删除对应的跟踪器
unmatched_trackers = []
for t, trk in enumerate(trackers):
if t not in matched_indices[:, 1]:
unmatched_trackers.append(t)
# 将匹配成功的跟踪框放入 matches 中
matches = []
for m in matched_indices:
# 过滤掉 IOU 低的匹配,将其放入到 unmatched_detections 和 unmatched_trackers
if iou_matrix[m[0], m[1]] < iou_threshold:
unmatched_detections.append(m[0])
unmatched_trackers.append(m[1])
# 满足条件的以 [[d,t]...] 的形式放入 matches 中
else:
matches.append(m.reshape(1, 2))
# 初始化 matches,以 np.array 的形式返回
if len(matches) == 0:
matches = np.empty((0, 2), dtype=int)
else:
matches = np.concatenate(matches, axis=0)
return matches, np.array(unmatched_detections), np.array(unmatched_trackers)
二、计算目标之间的相似性:马氏距离/余弦距离/IoU
2.1、马氏距离(Mahalanobis distance)
马氏距离(Mahalanobis distance) 是一种有效的计算两个向量之间相似度的方法(越小越接近样本均值也越相似,越大越远离样本均值可能是异常点),可以看作是欧氏距离的一种修正,修正了欧式距离中 各个维度(x/y/r/h)尺度不一致 且相关的问题,使得在高维空间中更具有鲁棒性
- 两个向量间的马氏距离: D m ( x , y ) = ( x − y ) T Σ x , y − 1 ( x − y ) D_m(x,y) = \sqrt{(x-y)^T\Sigma_{x,y}^{-1}(x-y)} Dm(x,y)=(x−y)TΣx,y−1(x−y)
- 其中 Σ \Sigma Σ 是多维随机变量的 协方差矩阵,若其是单位矩阵,则马氏距离退化为欧氏距离,即各维度间独立且同分布(因为只有同分布才会方差相同,对角线是相同的方差)
- 马氏距离的平方 计算公式如下:

-
工程实践:由于协方差矩阵 S 及其逆的求解计算量比较大,我们可以将其分解为 S = L ∗ L T S = L*L^T S=L∗LT,L 为三角阵,可以使用
scipy.linalg.solve_triangular来高效计算
M 2 ( x , y , L L T ) = ( x − y ) T ( L L T ) − 1 ( x − y ) = ( x − y ) T L T L − 1 ( x − y ) = ∣ ∣ L − 1 ( x − y ) ∣ ∣ 2 M^2(x,y,LL^T) = (x-y)^T(LL^T)^{-1}(x-y) = (x-y)^TL^{T}L^{-1}(x-y) = ||L^{-1}(x-y)||^2 M2(x,y,LLT)=(x−y)T(LLT)−1(x−y)=(x−y)TLTL−1(x−y)=∣∣L−1(x−y)∣∣2 -
代码实现如下:
def gating_distance(self, mean, covariance, measurements,
only_position=False, metric='maha'):
"""Compute gating distance between state distribution and measurements.
A suitable distance threshold can be obtained from `chi2inv95`. If
`only_position` is False, the chi-square distribution has 4 degrees of
freedom, otherwise 2. 计算状态分布和测量值之间的门控距离
Parameters
----------
mean : ndarray
Mean vector over the state distribution (8 dimensional).
covariance : ndarray
Covariance of the state distribution (8x8 dimensional).
measurements : ndarray
An Nx4 dimensional matrix of N measurements, each in
format (x, y, a, h) where (x, y) is the bounding box center
position, a the aspect ratio, and h the height.
only_position : Optional[bool]
If True, distance computation is done with respect to the bounding
box center position only.
Returns
-------
ndarray
Returns an array of length N, where the i-th element contains the
squared Mahalanobis distance between (mean, covariance) and
`measurements[i]`.
"""
mean, covariance = self.project(mean, covariance)
if only_position:
mean, covariance = mean[:2], covariance[:2, :2]
measurements = measurements[:, :2]
d = measurements - mean
if metric == 'gaussian':
return np.sum(d * d, axis=1)
elif metric == 'maha':
# 计算协方差矩阵的 Cholesky 分解,该分解将协方差矩阵分解为下三角矩阵 cholesky_factor,
# 满足 covariance = cholesky_factor @ cholesky_factor.T
cholesky_factor = np.linalg.cholesky(covariance)
# scipy.linalg.solve_triangular 解决了一个线性方程组,形式为:cholesky_factor @ z = d.T,即求解 z = cholesky_factor^{-1} @ d.T
z = scipy.linalg.solve_triangular(cholesky_factor, d.T, lower=True, check_finite=False, overwrite_b=True)
# z^2 = (cholesky_factor^{-1} @ d.T)^2
squared_maha = np.sum(z * z, axis=0)
return squared_maha
else:
raise ValueError('invalid distance metric')
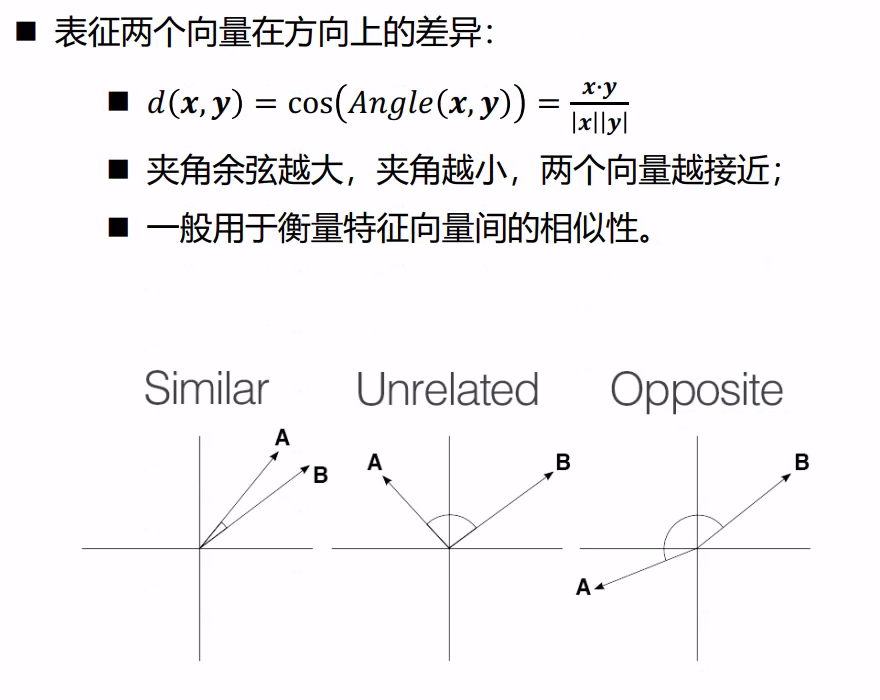
2.2、余弦距离(Cosine Distance)
- 一般用于 reid 特征间
余弦距离(1 - 余弦相似度)的计算,其中x.y是向量 x 和向量 y 的点积,|x||y|是向量 x 和 y 的二范数乘积
# 方法 1:使用 numpy 计算余弦距离
def _cosine_distance(a, b, data_is_normalized=False):
# a和b之间的余弦距离
# a : [NxM] b : [LxM]
# 余弦距离 = 1 - 余弦相似度 = 1 - (a * b) / (||a|| * ||b||)
# https://blog.csdn.net/u013749540/article/details/51813922
"""Compute pair-wise cosine distance between points in `a` and `b`.
Parameters
----------
a : array_like
An NxM matrix of N samples of dimensionality M.
b : array_like
An LxM matrix of L samples of dimensionality M.
data_is_normalized : Optional[bool]
If True, assumes rows in a and b are unit length vectors.
Otherwise, a and b are explicitly normalized to lenght 1.
Returns
-------
ndarray
Returns a matrix of size len(a), len(b) such that eleement (i, j)
contains the squared distance between `a[i]` and `b[j]`.
"""
if not data_is_normalized:
# 需要将余弦相似度转化成类似欧氏距离的余弦距离。
a = np.asarray(a) / np.linalg.norm(a, axis=1, keepdims=True)
# np.linalg.norm 操作是求向量的范式,默认是 L2 范式,等同于求向量的欧式距离。
b = np.asarray(b) / np.linalg.norm(b, axis=1, keepdims=True)
return 1. - np.dot(a, b.T)
# 方法 2
from scipy.spatial.distance import cosine
# 示例向量
vector_a = [1, 0, 1]
vector_b = [1, 1, 0]
# 计算余弦距离
distance = cosine(vector_a, vector_b)
print("Cosine Distance:", distance)
2.3、IoU 系列

DIoU:兼顾交并比 + 预测框和真实框的中心点距离

- 代码实现
def iou(bbox, candidates):
# 计算iou
"""Computer intersection over union.
Parameters
----------
bbox : ndarray(track)
A bounding box in format `(top left x, top left y, width, height)`.
candidates : ndarray(detections)
A matrix of candidate bounding boxes (one per row) in the same format
as `bbox`.
Returns
-------
ndarray
The intersection over union in [0, 1] between the `bbox` and each
candidate. A higher score means a larger fraction of the `bbox` is
occluded by the candidate.
"""
bbox_tl, bbox_br = bbox[:2], bbox[:2] + bbox[2:]
candidates_tl = candidates[:, :2]
candidates_br = candidates[:, :2] + candidates[:, 2:]
tl = np.c_[np.maximum(bbox_tl[0], candidates_tl[:, 0])[:, np.newaxis],
np.maximum(bbox_tl[1], candidates_tl[:, 1])[:, np.newaxis]]
br = np.c_[np.minimum(bbox_br[0], candidates_br[:, 0])[:, np.newaxis],
np.minimum(bbox_br[1], candidates_br[:, 1])[:, np.newaxis]]
wh = np.maximum(0., br - tl)
area_intersection = wh.prod(axis=1)
area_bbox = bbox[2:].prod()
area_candidates = candidates[:, 2:].prod(axis=1)
return area_intersection / (area_bbox + area_candidates - area_intersection)
def iou_cost(tracks, detections, track_indices=None, detection_indices=None):
# 计算 track 和 detection 之间的 iou 距离矩阵
"""An intersection over union distance metric.
Parameters
----------
tracks : List[deep_sort.track.Track]
A list of tracks.
detections : List[deep_sort.detection.Detection]
A list of detections.
track_indices : Optional[List[int]]
A list of indices to tracks that should be matched. Defaults to
all `tracks`.
detection_indices : Optional[List[int]]
A list of indices to detections that should be matched. Defaults
to all `detections`.
Returns
-------
ndarray
Returns a cost matrix of shape
len(track_indices), len(detection_indices) where entry (i, j) is
`1 - iou(tracks[track_indices[i]], detections[detection_indices[j]])`.
"""
if track_indices is None:
track_indices = np.arange(len(tracks))
if detection_indices is None:
detection_indices = np.arange(len(detections))
cost_matrix = np.zeros((len(track_indices), len(detection_indices)))
for row, track_idx in enumerate(track_indices):
# 丢失超过 1 帧的 track,直接设置为最大代价(TODO: 为什么是 INFTY_COST?为什么丢失超过 1 帧的 track 会直接设置为最大代价?可以考虑放开限制,增加被遮挡找回的可能性)
# 如果丢失超过 1 帧的 track,直接设置为最大代价,这样在后续的匹配过程中,这个 track 不会被匹配到任何 detection。
# 如果一个目标丢失太久(超过 1 帧),它可能已经不再出现在场景中,因此被认为不再是一个有效的轨迹
# 如果一个目标被短暂遮挡丢失并且之后重新出现,那么设置丢失超过 1 帧的轨迹为最大代价(INFTY_COST)的策略可能会错过这个目标。
# 这种情况下,目标可能并没有真正消失,而是因为某种原因(比如遮挡)短暂未被跟踪到
if tracks[track_idx].time_since_update > 1:
cost_matrix[row, :] = linear_assignment.INFTY_COST
continue
bbox = tracks[track_idx].to_tlwh()
candidates = np.asarray([detections[i].tlwh for i in detection_indices])
cost_matrix[row, :] = 1. - iou(bbox, candidates)
return cost_matrix
三、最优关联匹配:匈牙利匹配
- 如何定义最优的关联匹配?
- 直觉思维:让最接近/相似的目标匹配上(
最近邻匹配)- 大局观:让匹配相似度的总和最大(
匈牙利匹配)- 不做选择:每一种可能我都要(
概率数据关联/多假设跟踪)- 这里我们主要介绍匈牙利匹配
3.1、二分图的定义及其在跟踪中的理解

- 二分图的定义:二分图是能分成两组(假设是 U 和 V),其中 U 上的点不能相互连通,只能连去 V 中的点;V 中的点也不能相互连通,只能连去 U 中的点
- 二分图在跟踪中的理解:可以把二分图理解为视频中连续两帧中的所有检测框,第一帧所有检测框的集合称为 U,第二帧所有检测框的集合称为 V,同一帧的不同检测框不会为同一个目标,所以不需要互相关联,相邻两帧的检测框需要相互联通,最终将相邻两帧的检测框尽量完美地两两匹配起来
3.2、匈牙利算法、KM 算法
-
匈牙利算法定义及缺点:
- 定义: 是一种在
多项式时间内求解任务分配问题的组合优化算法,在多目标跟踪问题中可以简单理解为寻找 前后两帧的若干目标的匹配最优解 的一种算法 - 缺点: 将每个匹配对象的地位视为相同,在这个前提下求解最大匹配,这个和我们研究的多目标跟踪问题有些不合,因为每个匹配对象不可能是同等地位的,总有一个真实目标是我们要找的最佳匹配,
而这个真实目标应该拥有更高的权重,在此基础上匹配的结果才能更贴近真实情况
- 定义: 是一种在
-
-
KM 算法定义和优点:
- 定义:解决的是
带权二分图的最优匹配问题,给每条连接关系加入了权重(权重的计算可通过前后两帧状态变量的马氏距离 或者 reid 特征的余弦相似度 或者 状态变量间的IoU 等) - 优点:可以实现最优匹配,而不是简单的取相似度最大的进行匹配
- 定义:解决的是
-
KM 算法匹配原则:
- 首先,初始化各个顶点(称为顶标),将左边的顶点赋值为与其相连边的最大权重(权重的计算可通过
前后两帧状态变量的马氏距离 或者 reid 特征的余弦相似度 或者 状态变量间的IoU 等),右边的顶点赋值为 0 - 然后,只和权重与左边分数(顶标)相同的边进行匹配
- 最后,若找不到边匹配,对此条路径 所有冲突边的顶点做加减操作,令左边顶点值减 d,右边顶点值加 d(参数 d 这里取 0.1)
- 首先,初始化各个顶点(称为顶标),将左边的顶点赋值为与其相连边的最大权重(权重的计算可通过
-
KM 算法匹配示例:
- 第 1 步: 对于左 1,与顶标分值相同的边标蓝
- 第 2 步: 对于左 2,与顶标分值相同的边标蓝
- 第 3 步: 对于左 3,发现与右 1 已经与左 1 配对,而此时左 3 和 左 1 都无法更换匹配的边(找不到其它权值大于等于左顶标加右顶标的边),此时根据 KM 算法匹配原则,应对所有冲突的边的顶点做加减操作,令左边顶点值减 0.1,右边顶点值加 0.1
- 第 4 步: 进行加减操作后再进行匹配,发现左3 多了一条可匹配的边(左 3 对右 2 的匹配要求只需权重大于等于 0.8+0),所以左 3 与右 2 匹配
- 第 5 步: 对于左 4,由于左 4 唯一的匹配对象右 3 已被左 2 匹配,发生冲突,进行一轮加减操作再进行匹配,左四还是匹配失败,两轮以后左 4 期望值降为 0,放弃匹配左 4

-
3.3、匈牙利算法的实现
linear_sum_assignment求的是最大匹配,使得代价最小,传入的是 cost 距离矩阵(iou 距离或 cos 距离+马氏距离限制)
import numpy as np
from scipy.optimize import linear_sum_assignment
# 代价矩阵 4*4, 前后帧都有四个目标:权重可通过马氏距离或者余弦相似度计算
cost = np.array([[0.8, 0.6, 0, 0],
[0, 0.3, 0.9, 0],
[0.9, 0.8, 0, 0],
[0, 0, 0.2, 0]])
# 匹配结果:该方法的目的是代价最小,这里是求最大匹配,所以将 cost 取负数
row_ind, col_ind = linear_sum_assignment(-cost)
# 对应的行索引
print("行索引:\n{}".format(row_ind))
# 对应行索引的最优指派的列索引
print("列索引:\n{}".format(col_ind))
# 提取每个行索引的最优指派列索引所在的元素,形成数组
print("匹配度:\n{}".format(cost[row_ind, col_ind]))
# 输出结果如下
行索引:
[0 1 2 3]
列索引:
[0 2 1 3]
匹配度:
[0.8 0.9 0.8 0. ] # 匹配度为 0,说明没有匹配上








 本文介绍了在视频监控中,如何利用马氏距离、余弦距离和IoU计算目标间的相似性,以及如何通过匈牙利算法进行最优关联匹配,如卡尔曼滤波后的跟踪框与检测框的匹配,重点讲解了二分图的概念和KM算法在多目标跟踪中的应用。
本文介绍了在视频监控中,如何利用马氏距离、余弦距离和IoU计算目标间的相似性,以及如何通过匈牙利算法进行最优关联匹配,如卡尔曼滤波后的跟踪框与检测框的匹配,重点讲解了二分图的概念和KM算法在多目标跟踪中的应用。
















 2981
2981

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








