写在前面
本文介绍了 Bayes' theorem(贝叶斯定理),Bayesian inference(贝叶斯推理),Bayesian regression Model (贝叶斯回归模型) 等。
贝叶斯定理大家都耳熟能详,高中数学甚至都有涉及,即:用先验概率和条件概率求出另外的条件概率。
但贝叶斯推理和回归模型我认为是一个比较trick 的内容。
用一张图解释本文的内容:

一些术语
prior : 1 happening or existing before sth else or before a particular time , 2 already existing and therefore more important;
likelihood : 参考我之前的博客;
posterior : located behind sth or at the back of sth
Bayes' theorem 贝叶斯定理
简单来说就是用先验概率和条件概率求出另外的条件概率。

Bayesian inference
将贝叶斯定理应用在统计分析中, 即模型参数是未知量, 而且它们的先验分布需要被(手动/经验地)指定 — this is Bayesian inference.
(在贝叶斯框架中,我们讨论的是模型参数的概率分布 ,注意,在那些常见的框架中,如以MLE为基础的GLM,模型参数是 fixed non-random quantities ,数据才是遵循某种概率分布 ,这是两者显著的不同。
一个粗鲁的解释就是:我们有一堆观测数据,我们揣测这个数据可能符合某种模型分布,如正态。泊松等,然后模型的参数也被我们 揣测地 定了下来, 然后我们用观测数据和这个我们揣测的模型 去收敛训练, 最后我们得到了 posterior ,这个 posterior,是 我们根据观测数据收敛训练,得到的模型的参数 ,注意我们得到的是参数的分布,而不是点估计。
更多的可以参考这里。)
假设:
1 观测量,即数据 ,是 x;
2 位置量是θ,这个 θ 可以代表模型参数,数据中地缺失值,误测值等;
基于我们上面的假设,
首先我们提出 likelihood :p(x | θ)
然后我们从贝叶斯定理的角度观察 : θ 是未知的,所以它需要有(被手动/经验地指定)一个概率分布,用来表示这个 不确定性; x 是已知的,所以它是条件(condition on it),我们得到 posterior distribution :
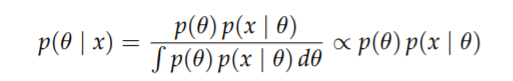
所以总的来说,我们首先指定了一个 prior distribution p(θ), 表达了我们在看到数据之前对θ不确定性的预估. The posterior distribution p(θ | x), 表达了我们在看到数据之后对θ不确定性的预估。
很明显:1 我们的 posterior 是prior 和 data (likelihood) 的居中选择,2 如果我们的数据越多,我们的 data (likelihood )的 影响力就越大,prior 影响力就越小 (比如你认为 它们都是好的,但搞了一堆观测数据发现它们基本都是坏的,那推论 必然更容易是 它们是坏的)








 本文深入浅出地介绍了贝叶斯定理、贝叶斯推理和贝叶斯回归模型,并结合R语言进行实战演示。通过马尔可夫链蒙特卡洛(MCMC)方法求解后验分布,揭示了贝叶斯方法在统计分析中的独特之处。
本文深入浅出地介绍了贝叶斯定理、贝叶斯推理和贝叶斯回归模型,并结合R语言进行实战演示。通过马尔可夫链蒙特卡洛(MCMC)方法求解后验分布,揭示了贝叶斯方法在统计分析中的独特之处。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章


















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










