我也只是个学生物的小垃圾,这个文章是写给看不懂图的那些人的,大佬勿入。
文章纯手打,可能存在错别字;我尽可能用最简单易懂的语言来解释这些图。如果文章出现漏误,请各位批评指正。
目录看我
VENN图
venn 图可用于统计多个样品中所共有和独有的OTU数目,可以比较直观的表现环境样品的OTU 数目组成相似性及重叠情况。
单张分析图,样本分组至少两个,最多 5 个。
-
韦恩图,虽然下图看着很复杂,但是我们我们其实都接触过,回想一下高中数学。下图是不是就想起来了。图a代表A∪B,图b代表A∩B,这种表示集合关系的图就是韦恩图。
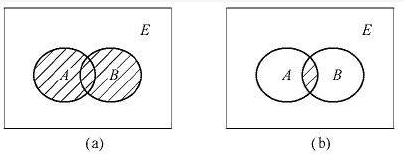
-
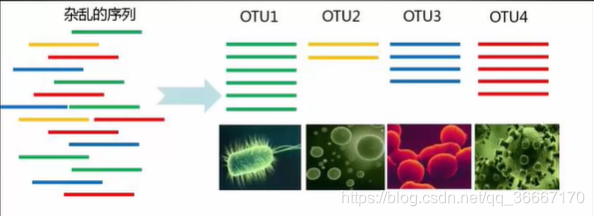
OTU(operational taxonomic unit ),就是操作分类单元。简单理解一下,你进行16S测序之后会得到各种序列。对于这些序列我们要对比数据库来看一下它是什么物种。
- 我们可以挨个序列对比,但是这样对比起来工作量实在是太大了。
- 也可以选择聚类之后再对比:一般来说,序列达到97%相似就可以认为是同一个物种,因此聚类就是把相似度为97%的序列放到一起形成一个OTU,然后在每个OTU中选取一个代表性进行数据库比对,比对结果就是整个OTU的结果。
比如下图中相似度为97%的一部分序列为红色,把这部分聚在一起形成OTU4,从中选一个序列比对,得到这个序列是XXX冠状病毒,然后整个OTU4的所有序列我们都认为是XXX冠状病毒的序列。
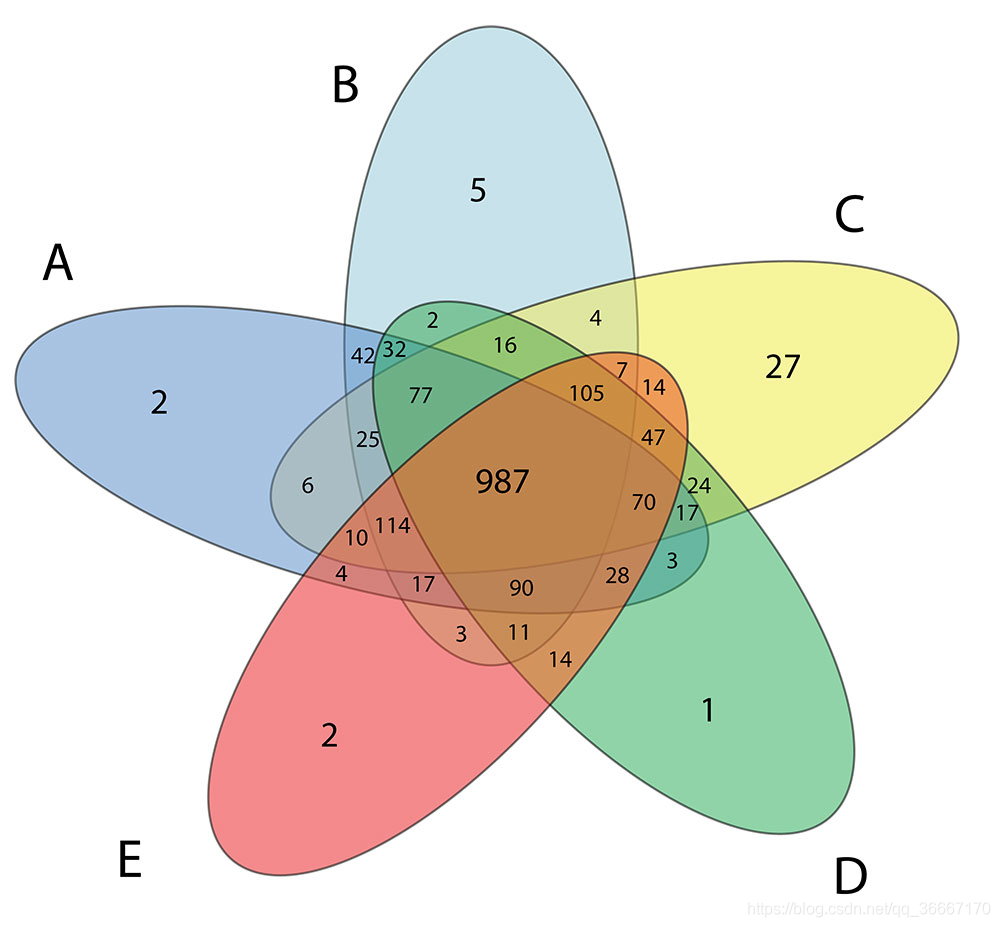
下图中A-E样品中,所有样品都含有的OTUs的数量为987,各自独有的数量为2,5,27,1,2,共有的比例较大,则证明五个样品的相似度较高。
例图:使用97%相似度的OTU,R 语言工具统计和作图。
参考文献:Fouts DE, Szpakowski S, Purushe J, Torralba M, Waterman RC, et al. (2012) Next GenerationSequencing to Define Prokaryotic and Fungal Diversity in the Bovine Rumen. PLoS ONE 7(11): e48289. doi:10.1371/journal.pone.0048289.
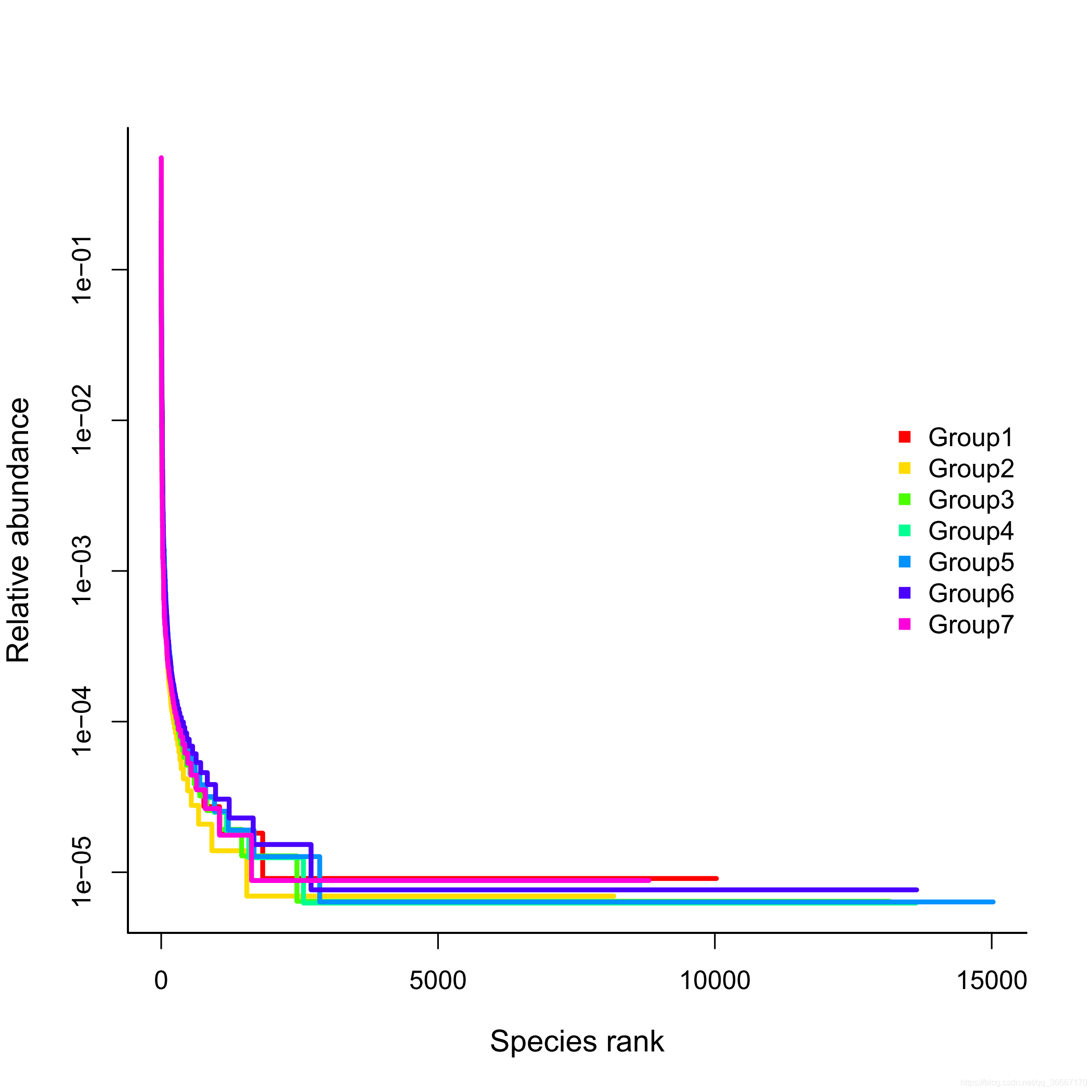
rank abundance
在16S rRNA扩增子分析中,rank abundance可以从OTU的层面总体的反映出物种的分布情况(丰度和均匀度)。
曲线中,曲线在横轴上的跨度越长,表明样品的物种含量越丰富;纵轴上看曲线越平坦,表示样品的物种组成越均匀。
- 横坐标:物种的丰度
- 有的表示OTU 等级,“500”代表样本中按照丰度排列第500 位的OTU;
- 或者可以理解为OTU的数量,横坐标跨度多大就表示多少OTU数量。
- 纵坐标:物种的均匀度,某一等级OTU中序列数的相对百分含量,即属于该OTU 的序列数除以总序列数,纵坐标轴上数字,例如“100”代表相对丰度为100%,“10”代表相对丰度为10%,依次类推。
- 相对丰度:就是该OTU所包含的序列数除以总的序列数,就是占比。
图中Group5中有15000个OTU,Group1中有10000个OTU,因此Group5的物种丰度大于Group1。
Rank abundance计算
- 获取每个样本中OTU的丰度值,即每个OTU中有多少条序列。
- 将每个样本中OTU的丰度值按照从大到小顺序进行排序,并计算总丰度
- 计算获取每个样本OTU的相对丰度。
举个栗子:
- 样本1中有5个OTU,丰度分别为
5,4,3,3,5 - 排序后为
5,5,4,3,3 - 总丰度
5 + 5 + 4 + 3 + 3 = 20 - 计算相对丰度
5 ÷ 20 = 0.25
5 ÷ 20 = 0.25
4 ÷ 20 = 0.2
3 ÷ 20 = 0.15
3 ÷ 20 = 0.15
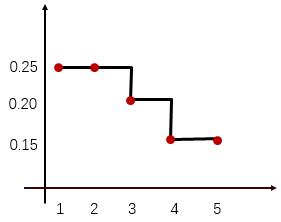
再看看下图,蓝色有6个OTU,绿色五个,所以蓝色丰度更大。绿色比蓝色更平缓,因此绿色均匀度更高。
多样性指数
- alpha多样性
- beta多样性
- gamma多样性
alpha多样性指数
(样本内多样性)
用于测量群落内生物种类数量以及生物种类间相对多度的一种测量。它反映了群落内物种间通过竞争资源或利用同种生境而产生的共存结果。是相对样本本身来说的,也就是说一个样本就可以做alpha多样性分析。
- 物种丰富度指数:样品中所含物种的多少,反应一定空间范围内生物的丰富程度。
例如:Margalef丰富度指数、 Menhnick丰富度指数等。 - 物种均匀度指数:刻画群落中各个种的相对密度。
例如:Pielou均匀度指数, Sheldon均匀度指数,Hill均匀度指数、Heip均匀度指数、 Alatalo均匀度指数等 - 物种多样性指数:将物种多样性和种的丰富度结合起来。
例如:Shannon-Wienner多样性指数, Simpson多样性指数,Hill多样性指数以及种间相遇概率(PIE)等
Chao1丰富度估计量(Chao1 richness estimator)
Chao多样性是用chao1算法估计群落中含OTU数目的指数,Chao1在生态学中常用来估计物种总数,由Chao(1984)最早提出。Chao1值越大代表物种总数越多。
香农-威纳指数(Shannon Wiener index)
Shannon值越大,说明群落多样性越高。
香农-威纳指数借用了信息论方法,你不用管什么是信息论,你看了下边的例子就知道了。
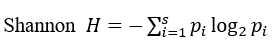
注: 也有人把log2换成ln
上边是计算公式,借助公式做个题:
假设现在有三个群落。有两个物种在群落中分布如下。
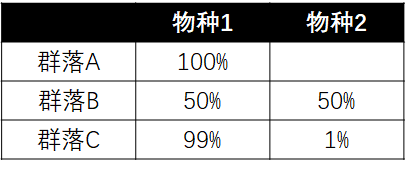
根据公式我们来计算
A群落:
H
A
=
−
(
1
∗
l
o
g
2
1
+
0
)
=
0
H_A=-(1*log_2 1 +0) =0
HA=−(1∗log21+0)=0
B群落:
H
B
=
−
(
0.5
∗
l
o
g
2
0.5
+
0.5
∗
l
o
g
2
0.5
)
=
1
H_B =-(0.5*log_2 0.5 +0.5*log_2 0.5) =1
HB=−(0.5∗log20.5+0.5∗log20.5)=1
C群落:
H
C
=
−
(
0.99
∗
l
o
g
2
0.99
+
0.01
∗
l
o
g
2
0.01
)
=
0.081
H_C=-(0.99*log_2 0.99+0.01*log_2 0.01)=0.081
HC=−(0.99∗log20.99+0.01∗log20.01)=0.081
结合数据和图我们可以看出,群落A只有一个物种,显然群落结构单一,多样性小,我们的计算公式也印证了,香农指数是0,群落多样性小;群落B和C中均有两个物种,B中分布均匀,C中分布极不均匀,从计算公式中我们得出群落B比群落C多样性更高。
辛普森多样性指数(Simpson diversity index)
Simpson指数值越大,说明群落多样性越高。
辛
普
森
多
样
性
指
数
=
随
机
取
样
的
两
个
个
体
属
于
不
同
种
的
概
率
=
1
−
随
机
取
样
的
两
个
个
体
属
于
同
种
的
概
率
辛普森多样性指数 = 随机取样的两个个体属于不同种的概率=1-随机取样的两个个体属于同种的概率
辛普森多样性指数=随机取样的两个个体属于不同种的概率=1−随机取样的两个个体属于同种的概率
辛普森指数是借助了熵的原理。也不用懂什么是熵。你可以理解为辛普森指数是从群落里随机抓两只动物,看看他们是不是一个物种。下图中表示三个群落,从每个群落中都抓两只动物,两只动物属于不同物种的概率谁大谁小一目了然吧。
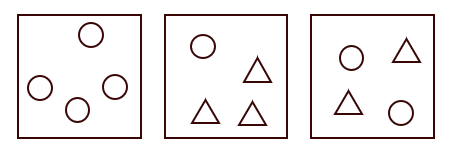
这个看下边公式感觉好麻烦,不用看了。

beta多样性指数
生态系之间的种多样性,它包含分类单位的比较。即衡量群落之间的差别。Beta多样性不仅描述生境內生物种类的数量,同时也考虑到这些种类的相同性及其彼此之间的位置。用于不同样品以及同一样品不同条件下的比较。
- Whittaker指数、Cody指数、 Wilson和 Shmida指数等
beta多样性意义
- 它可以指示生境被物种隔离的程度
- β多样性的测定值可以用来比较不同地段的生境多样性
- β多样性与α多样性一起构成了总体多样性或一定地段的生物异质性
PCoA分析
PCoA分析 principal co- ordinates analysis)是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,PCoA可以找到距离矩阵中最主要的坐标结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。通过PCoA可以观察个体或群体间的差异。
PCA分析
PCA( Principal component analysis),叫做主成分分析,是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要的前几位特征值,采取降维的思想,PCA可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。
其优点是简单且无参数限制。通过分析不同样品OTU(97%相似性)组成可以反映样品间的差异和距离,PCA 运用方差分解,将多组数据的差异反映在二维坐标图上,坐标轴取能够最大反映方差值的两个特征值。**如样品组成越相似,反映在PCA 图中的距离越近。**不同环境间的样品可能表现出分散和聚集的分布情况,PCA 结果中对样品差异性解释度最高的两个或三个成分可以用于对假设因素进行验证。
例图:使用97%相似度的OTU,PC-ORD或是CANOCO作图。
参考文献:Yu Wang, Hua-Fang Sheng, et al. Comparison of the Levels of Bacterial Diversity in Freshwater, Intertidal Wetland, and Marine Sediments by Using Millions of Illumina Tags. Appl. Environ. Microbiol. 2012, 78(23):8264. DOI: 10.1128/AEM.01821-12
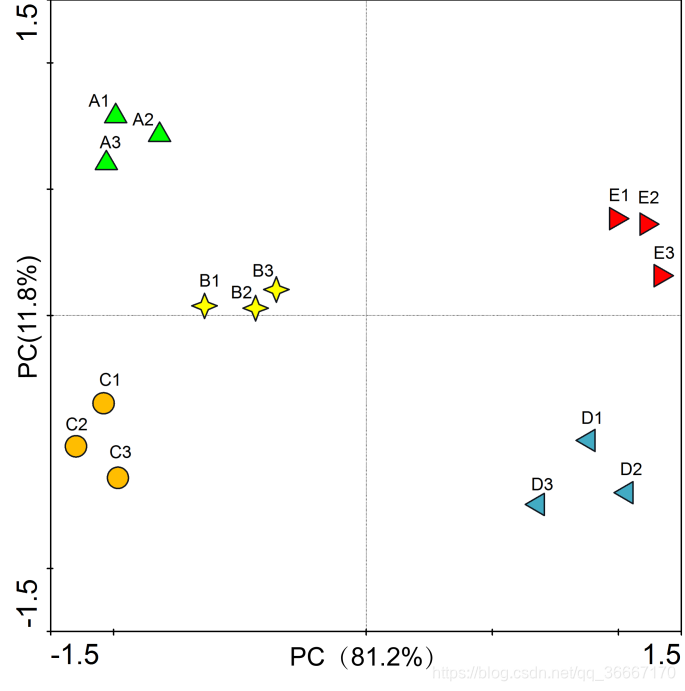
- 十字交叉线:作为 0 点基线存在,起到辅助分析的作用,本身没有意义
- 每个点代表了一个样本;颜色则代表不同的样品分组
- 两点之间在横、纵坐标上的距离,代表了样品受主成分(PC1 或 PC2)影响下的相似性距离
- 样本数量越多,该分析意义越大;反之样本数量过少,会产生个体差异,导致 PCA 分析成图后形成较大距离的分开,建议多组样品时,每组不少于 5 个,不分组时样品不少于 10 个
NMDS分析
非度量多维尺度法是一种将多维空间的研究对象(样本或变量)简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法。适用于无法获得研究对象间精确的相似性或相异性数据,仅能得到他们之间等级关系数据的情形。其基本特征是将对象间的相似性或相异性数据看成点间距离的单调函数,在保持原始数据次序关系的基础上,用新的相同次序的数据列替换原始数据进行度量型多维尺度分析。换句话说,当资料不适合直接进行变量型多维尺度分析时,对其进行变量变换,再采用变量型多维尺度分析,对原始资料而言,就称之为非度量型多维尺度分析。其特点是根据样品中包含的物种信息,以点的形式反映在多维空间上,而对不同样品间的差异程度,则是通过点与点间的距离体现的,最终获得样品的空间定位点图。
没空,以后有空我再零零碎碎补充。






















 7444
7444











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










