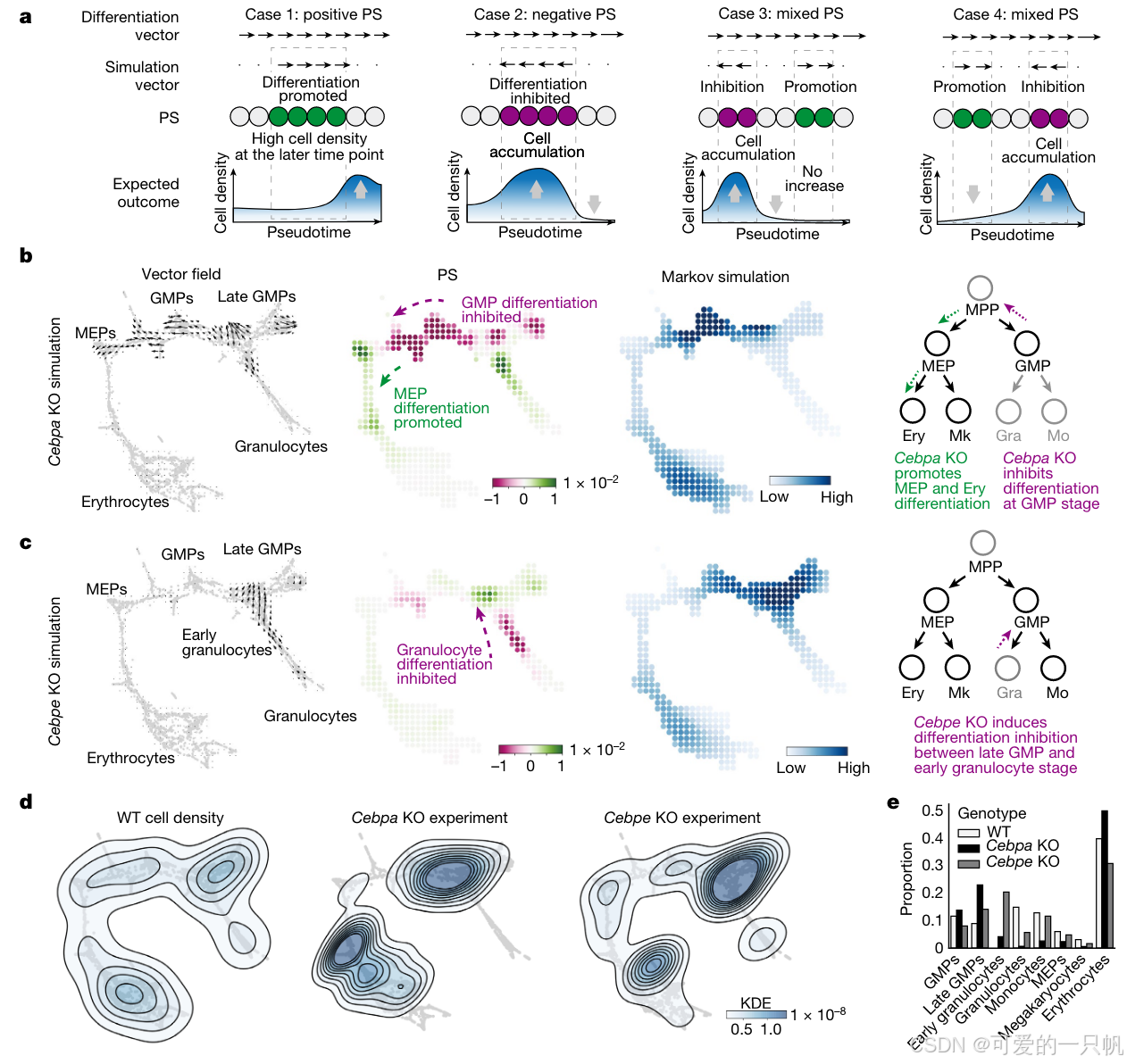
CellOracle是一种结合计算扰动与基因调控网络(GRN)建模的策略,用于模拟转录因子(TF)扰动后细胞身份的变化,并为细胞身份的调控提供系统性和直观的解释。

🔬算法流程:
1.构建细胞类型/状态特异的GRN配置:通过多组学数据和集群正则化线性回归模型,构建细胞类型或状态特异的GRN配置。
2. 计算转录因子扰动对目标基因表达的影响:利用GRN模型,模拟转录因子KO或过表达时基因表达的变化,进而推算出基因表达相对趋势变化。
3. 估算细胞身份转变的概率:通过比较基因表达的变化与局部邻域基因表达,估算细胞身份转变的概率。
4. 二维向量化转变概率:将转变概率转化为加权的局部平均向量,并通过降维得到二维向量,直观地展现细胞身份的变化方向。


🚀核心优势:无需湿实验的计算扰动
CellOracle通过计算模拟转录因子的扰动效果,无需实际实验操作即可预测基因调控机制和细胞身份变化。
参考文献:Nature volume 614, pages742–751 (2023)























 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








