场景1:
A同学想要从对蛋白结构影响的角度说明 自己发现 的 患者(病人)携带的DNAH1 突变(如 p.Val131Ala)对蛋白结构的影响,通过SWISS-Model自己构建了 正常情况下的蛋白结构,又同时构建了突变情况下的蛋白结构,如何对它们进行展示 以用于文章中的图呢?

文章中关于这个结果的描述:in silico analysis The location of the two variants is shown in Figure 2A.The affected AA residues, E446 and R971 of the DNAH1peptide, **are evolutionarily conserved with high consensus across eight species (**Fig. 2B). Only the domain containingthe p.R971H variant was credibly modeled using the PDB:6sc2.1 file as its wild-type template (Seq identity: 25.11%;Qualitative Model Energy Analysis: -4.63). The R971Hvariant, which replaced the arginine with a histidine, changed the number of hydrogen bonds from two to one at theside chain with E974, thus reducing the stability of the protein (Fig. 2C).
场景2
B同学想要在文章发表的时候提供如下的一条证据:
突变导致 患者 蛋白整体结构发生改变,从而为其致病性提供证据,如何通过蛋白结构的角度可视化展示这个结构呢?
常见的4种结构构建方法
要实现上边的可视化方法,我们首先要构建蛋白三维结构,拿到蛋白质的PDB文件,以下是常见的方法,根据需求选择其中之一进行构建即可:
- SWISS-Model:同源建模法

1.点击网站:https://swissmodel.expasy.org/interactive

(标准:氨基酸序列与选择的模板(模型)序列的一致度 >30% 则效果良好,可以进一步构建)
2.按照以下构建:
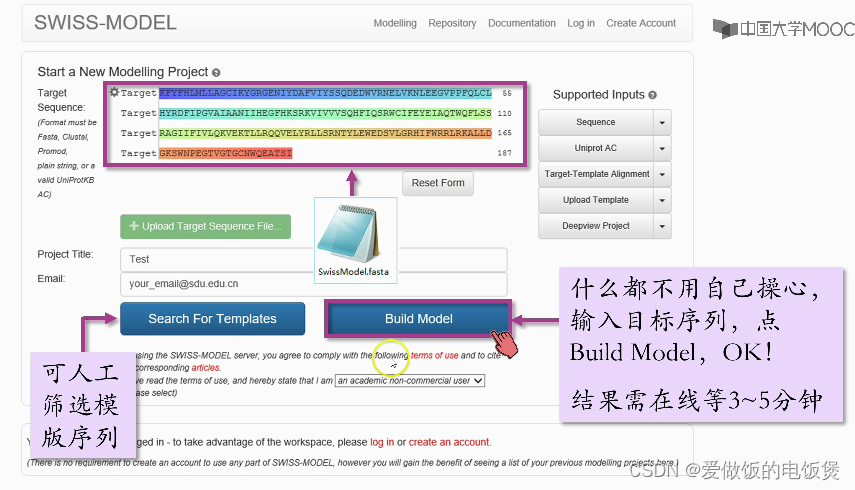
3.获得结果及分析

下载PDB文件,即完成这一部分
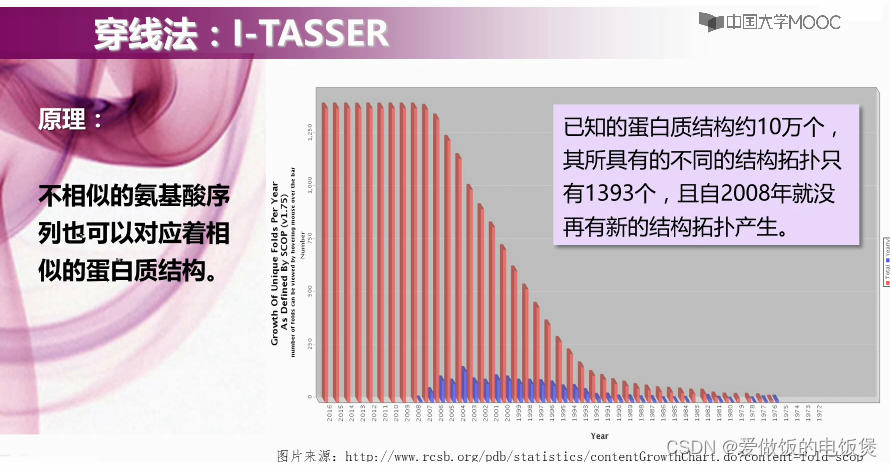
I-TASSER穿线法: 原理:不相似的氨基酸序列也可以对应着相似的蛋白质结构,对应能量最低的预测模型 在 CASP蛋白质预测信息蛋白中 表现优异
1.点击网站:I-TASSER 点击跳转 https://zhanggroup.org/

2.输入序列:

3.获得结果:

从头计算法 的原理 自由能最低的状态即是蛋白质所处的结构

构建方法:
1.点击进入QUARK 点击跳转https://zhanggroup.org/QUARK/ 还是 Zhang Lab的一款产品,需要注册账号
2.输入氨基酸序列 注意:<200个氨基酸序列的才适用

3.结果及分析
判断标准:根据TM-SCORE,查看预测的模型是否可用

综合法:
综合法原理:能够找到结果的区域用同源建模法,不能找到的采用从头计算法
1.ROBETTA:点击跳转 http://robetta.bakerlab.org/
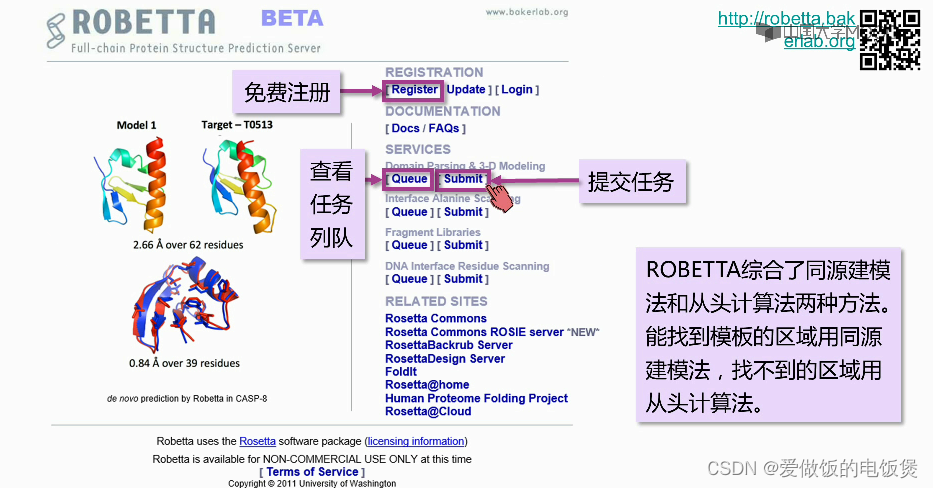
2.输入序列:

3.获得结果及分析

选择标准
蛋白质结构获得的方法:

获得模型后的最重要一步:模型质量评估:

不同软件对应不同的评估标准
统一的评估:Verify_3D
参考文献:
1.DNA Cell Biol. 2021 Jun;40(6):833-840. doi: 10.1089/dna.2021.0097.
Novel Biallelic DNAH1 Variations Cause Multiple Morphological Abnormalities of the Sperm Flagella








 本文介绍了如何通过SWISS-Model和I-TASSER等方法构建蛋白质结构,并分析DNAH1基因突变(p.Val131Ala和p.R971H)对蛋白结构的影响。突变导致的氢键变化降低了蛋白稳定性,为疾病关联性提供了结构依据。此外,还讨论了多种结构建模方法的使用和模型质量评估的重要性。
本文介绍了如何通过SWISS-Model和I-TASSER等方法构建蛋白质结构,并分析DNAH1基因突变(p.Val131Ala和p.R971H)对蛋白结构的影响。突变导致的氢键变化降低了蛋白稳定性,为疾病关联性提供了结构依据。此外,还讨论了多种结构建模方法的使用和模型质量评估的重要性。

















 1440
1440

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










