1、MedSAM 模型介绍
MedSAM全称为:Segment Anything in Medical Images
MedSAM 大模型利用了prompt engineering(提示工程)对指定区域进行分割
常见的分割提示有提示语、点、边界框等等,任务要求是收到提示符时至少输出一个有效的mask,哪怕提示是有歧义的
SAM通用分割大模型结构分为三个:图像编码器、提示器(prompt)和轻量级的解码器

这里的image encoder 输入size是1024*1024

关于SAM介绍可以自己百度或者阅读论文,这里只对代码简单介绍和复现
测试项目可以免费在这里下载,包含权重文件的:
2、环境配置
对应的readme文件写的挺详细的,这里做些补充
项目如下,github下载可能是MedSAM-main,重新命名即可

1、虚拟环境配置python的版本官方是3.10,命令如下
conda create -n medsam python=3.10 -y2、激活虚拟环境后(conda activate medsam),安装gpu版本的torch,官方的建议torch版本需要高于 2.0,应该会用到2.x版本的库文件
可以参考文章:Pytorch 配置 GPU 环境_pytorch gpu-CSDN博客
3、cd MedSAM目录后,运行下面代码即可
pip install -e .这个意思是,pip会安装setup.py脚本里面编写的库文件

3、利用官方权重推理图像
官方权重在这里:MedSAM - Google 云端硬盘
存放位置:work_dir/MedSAM/medsam_vit_b

运行 MedSAM_Inference.py 脚本即可:

这里会自动推理assets下的demo图像,然后保存在这里

不过这里推理后的图像是黑的,可以根据下面代码显示:
import matplotlib.pyplot as plt
img_path = 'assets/seg_img_demo.png'
img =plt.imread(img_path)
plt.imshow(img)
plt.show()
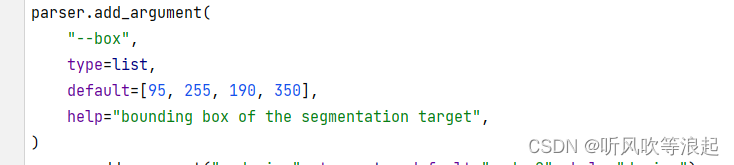
参数如下:
-i input_img
-o output path
--box bounding box of the segmentation target
因为SAM模型需要提示工程,所以传参的时候,也需要指定。这里是边界框,默认给定了。如果想要检测其他区域,需要更改这个bbox到自己想要推理的区域
官方给了简单的教程,在tutorial_quickstart.ipynb 中
4、利用 GUI 交互界面推理数据
安装库文件:
pip install PyQt5因为bbox很难指定,官方给出了GUI界面。类似于labelimg那种鼠标绘制边界框,即可推理指定的区域,这样方便推理
直接运行gui.py 脚本即可

要是出现上述错误,因为toch版本不对,这里直接将代码修改即可。红色框就是修改后的代码,源代码是被注释的四行

展示如下:

控制台输出:这里太卡了,就是做多余展示了

5、 项目复现
本章主要介绍一些代码的训练、复现等等
5.1 下载预训练权重
这里官方提供了SAM的预训练权重:https://dl.fbaipublicfiles.com/segment_anything/sam_vit_b_01ec64.pth
下载后保存在:work_dir/SAM/sam_vit_b_01ec64.pth

5.2 下载指定数据集
官方提供训练的数据集是:MICCAI FLARE22 挑战数据集,好像是腹部13类器官分割?
下载连接,也不是很大,就1.5 G多一点

数据集保存在:data/FLARE22Train/

5.3 数据预处理
预处理代码是 pre_CT_MR.py,需要先下载库文件
pip install connected-components-3d主要完成下面几个作用,划分数据集、进行窗口w和窗口h的灰度增强、归一化、resize以及保存到npy的2d格式

这里的npy其实就是2d数据,而非3D分割,后面会介绍。其实和我们平时习惯的mask用png表示一样的
这里的预处理脚本完全根据MICCAI FLARE22 挑战数据集数据集格式编写的,如果换成自己数据集的话,需要更改。其实就是目录啊、文件名之类的参数

因为这个代码还挺复杂的,这里分开介绍
5.3.1 切片成2D数据,并且剔除小部分的数据
关于切片介绍参考:
基于Unet的BraTS 3d 脑肿瘤医学图像分割,从nii.gz文件中切分出2D图片数据_brats2020unet分割-CSDN博客
或者这里更详细:nii 文件的相关操作(SimpleITK)_mimics输出nii文件-CSDN博客
MedSAM项目如下,这里明显只是对3D数据的横断面进行切分,最后保存成2D数据
3D数据的格式是【a,b,c】 这里的abc都很大
而非[a,b,3]这种颜色堆叠的rgb图像
而nii.gz 数据往往是医学的3D数据,换句话说,每个通道都是灰度的
而因为人体瘦长的缘故,沿着横断面切,效果是最好的,要不然数据会出现窄长的不均衡比例(瞎猜的)

之前本人写切片的时候,也会去除前景区域不足的数据,这里给了解释
换而言之,小区域是目标检测的任务,而非分割的重点!!
而这里设定的100,和我们之前设定的比例0.01之类的效果差不多

5.3.2 窗口宽度、窗口高度
学术叫法是 windowing 方法,专门处理医学图像这种灰度范围大的预处理方法
之前本人也写过,参考:医学图像处理的windowing 方法_医学图像常用windowing和histogram equalization-CSDN博客
其实说白了就是数字图像处理中的灰度拉伸,例如直方图均衡化啊、grammar变换啊、log变换啊等等都可以,只不过窗口化方法最好而已

就如下面图像,原始的医学图像可能是最右面的图像,经过windowing方法,变成最左边的,当然分割变得容易喽

Tips:
这个给了本人一些启发,对比其他unet之类的分割网络,也可以加入灰度拉伸增强对比度的方式对数据进行增强,以后可以尝试尝试
5.3.3 保存为npy格式
这里可以删除,不影响

保存好的数据如下:

5.3.4 读取npy图像
测试代码如下:
import numpy as np
import matplotlib.pyplot as plt
from PIL import Image
# 就是2d数据,只不过npy里面归一化了
img_path = 'data/npy/CT_Abd/imgs/CT_Abd_FLARE22_Tr_0001-000.npy'
img = np.load(img_path)
print(img.shape)
plt.imshow(img)
plt.savefig('demo.png')
打印:
![]()
如下:不仅是2d,经过windowing方法对比度也好了很多

5.4 训练
单 gpu 训练代码:
python train_one_gpu.py多 gpu 训练:
sbatch train_multi_gpus.shpython utils/ckpt_convert.py # Please set the corresponding checkpoint path first官方给的其他教程:GitHub - bowang-lab/MedSAM at 0.1
本章只介绍单 GPU 的代码
5.4.1 数据加载 dataset
这里很重要!!!!
这里很重要!!!!
这里很重要!!!!
这里很重要!!!!
本人之前一直在想,MedSAM有了bbox提示框后,怎么确认输入和输出的,其实通过dataset就可以知道
对于多类别的mask,这里每次只是选择一个。例如4分类的mask像素值有 0 1 2 3,那么这里随机挑选一个类型,例如1,那么gt2D中只有0 1这两个类别!!
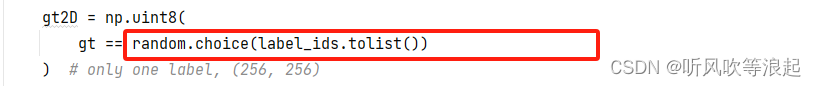
这就代表了MedSAM输出是2类别的!!因为每次都是随机挑选一个类别(加背景为2),当然,因为每次都是随机挑选某个label,那么epoch一定要多。否则的话,如果类别很多,倒霉的话,某个类别没有被挑到,那么网络对这个区域学不到东西!!
这里可以在deeplabv3代码体现:

因此对于MedSAM损失是针对2类别的 BCEWithLogitsLoss
对于bbox提示边界框,也很简单,对前景取值即可,取x,y最小、最大,加个修正偏移即可

bbox在数据上的表现就类似于画框,将图像指定区域框出来,所以输入的维度就多加了1
这里有个疑问,为什么对于医学的灰度图像,要repeat成3通道呢?
要不然输入应该是2,而非4了
5.4.2 训练
效果如下:

这里epoch改成了100

可视化结果:

权重:

5.5 推理
这里还没调试好,等后续补充
# TODO
6、其他
抛开MedSAM项目,只看代码的话,还是有很多地方值得学习
首先,对于医学图像,因为灰度的原因,导致对比度太差。对于简单的模型如unet这种,预处理也可以增加windowing方法,或者直方图之类的对比度增强的方法
其次,prompt 提示分割也可以借鉴,讲白了,就是把原始的rgb 3通道在加上一个通道,就是bbox的通道,这样每次unet的输入改成4就行了,也可以达到提示分割的目的。
后续的话,看有时间可以把SAM的bbox提示分割增加到unet里面
关于MedSAM 项目总结:
切片其实就是3d切分为2d,然后repeat成rgb 3通道,经过windowing对比度增强,最后保存成npy格式(和png格式一样其实)
dataset中对mask前景随机挑选一个,这样每次其实就是二分割任务,二分类任务简单不说,损失函数还可以实验BCE 逻辑损失。通过二分类的前景,取到bbox,增加到rgb图像中,这样输入就变成了4通道(这个思想可以添加到任何的分割网络中)
昨晚搞完的时候,好多想法, 结果写的时候全忘了。
写的地方可能还有很多不清晰的,大家有问题在评论区提问就行,看到就会回的。
算了,就先这样吧,下次想起来在更新.....

























 2964
2964

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










