还想纯生信发文?这样做就对了!
肿瘤与非肿瘤方向皆可开展的共病分析课题!
共病是指个体同时患有两种或更多的慢性疾病或医疗状况,既可以是一种疾病诱发另一种(如非肿瘤疾病导致肿瘤发生),也可以是两种疾病独立存在但共存的情况。选择创新性强的疾病组合,不仅能减少繁琐的分析与实验铺垫,还能大幅提高文章的发表潜力!
今天,我们来分享一篇创新的经典案例:整合了转录组学+生信分析,研究质量颇高,亮点突出。
这篇文章的选题方向是幽门螺杆菌感染相关胃癌的关键基因与机制。来自四川大学华西医院团队的文章,整合scRNA - seq明确基因表达模式、免疫基因挖掘+验证,随机森林算法筛选核心基因、免疫细胞浸润分析、构建调控网络揭示潜在机制。

题目:通过转录组学分析鉴定与幽门螺杆菌感染相关胃癌诊断的核心免疫相关基因CTSK、C3和IFITM1
杂志:Int J Biol Macromol
影响因子:IF=7.7
中科院分区:化学一区
发表时间:2024年12月
PART·1 研究背景
胃癌是常见的消化道恶性肿瘤,幽门螺杆菌(H. pylori)感染是其主要致病因素,约90%的病例与其相关,被列为Ⅰ类致癌物。H. pylori引发慢性炎症,最终导致胃黏膜病变进展为癌。
尽管H. pylori感染机制已有研究,其与胃癌的分子联系仍不明确。H. pylori感染通过免疫反应改变细胞稳态,促进炎症及肿瘤微环境形成,影响癌症进展。
近年来,转录组学技术揭示了H. pylori感染对宿主免疫的影响,为发现相关生物标志物提供了工具。本研究通过bulk RNA及单细胞RNA测序,分析免疫相关基因与免疫细胞浸润的关系,探索其在H. pylori相关胃癌中的机制,助力诊断和治疗策略优化。
PART·2 方法学
数据收集和处理
胃癌(GSE54129, GSE26899)及幽门螺杆菌(GSE60427, GSE5081, GSE27411, GSE60662)的基因表达谱数据来自基因表达数据库。
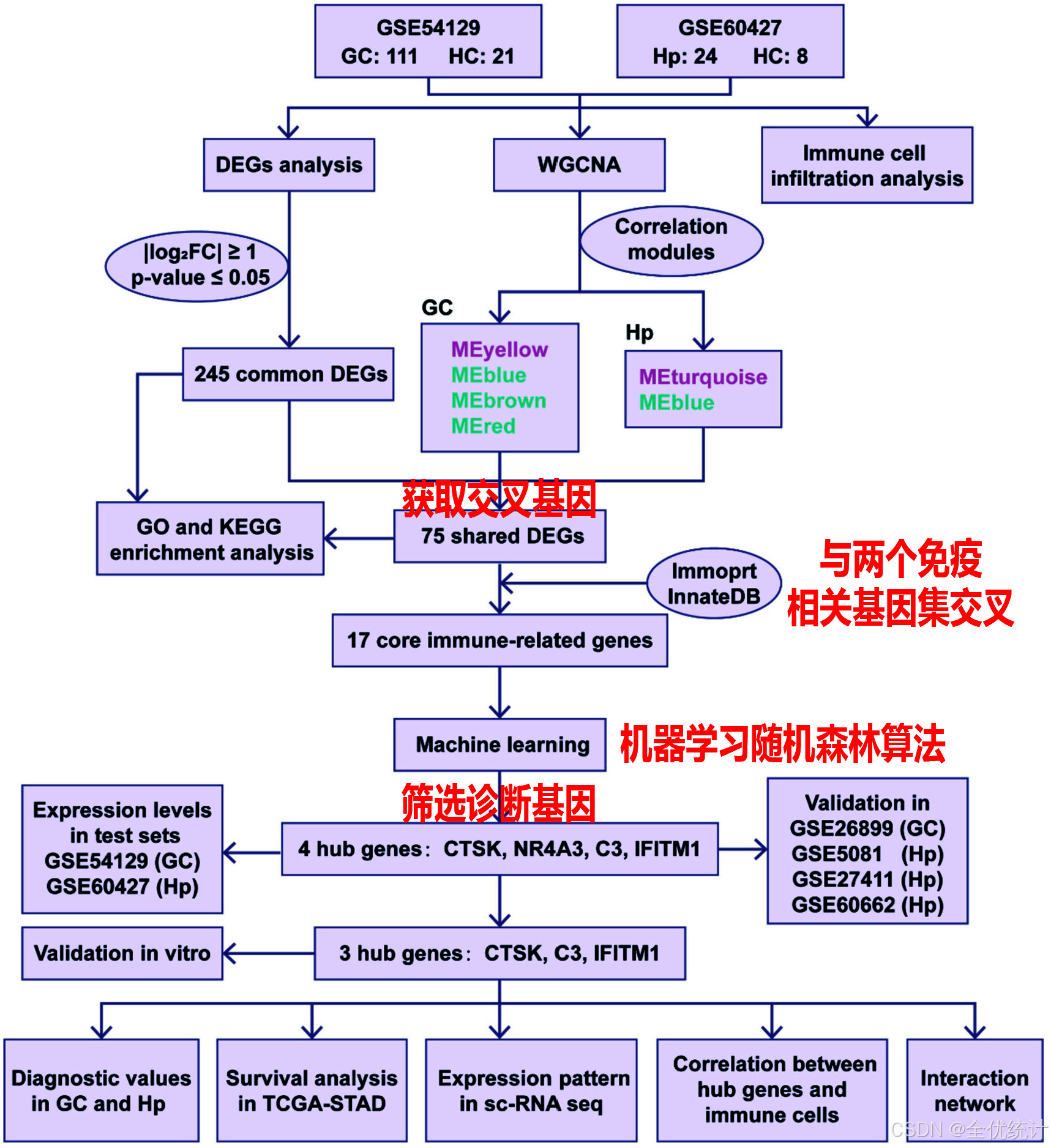
差异表达基因(DEGs)的鉴定和功能富集分析
R包“limma”识别差异表达基因,fold change为筛选标准,“gplot2”和“pheatmap”生成火山图和热图,“Venn”包描绘基因表达趋势。
加权基因共表达网络分析(WGCNA)分析
R包“WGCNA”构建基因共表达网络,选择合适值(GSE54129中,GSE60427中),应用动态树切割算法和动态混合合并方法确定基因模块,计算模块特征基因与样本特征的相关性。
免疫相关枢纽基因的鉴定
免疫相关基因列表提取自ImmPort数据库和InnateDB数据库。通过WGCNA筛选出的胃癌及幽门螺杆菌的差异表达基因(DEGs)和关键模块基因,与这些免疫相关基因使用韦恩图进行交集分析,最终确定与胃癌及幽门螺杆菌相关的核心免疫基因。
随机森林模型构建
利用“random Forest”包计算基因相对重要性得分,筛选候选诊断基因(得分>1),并通过交集确定最终候选基因。
诊断评价
用“ggplot2”包评估核心免疫相关基因在不同数据集的表达水平,ROC曲线计算AUC评估诊断准确性,采用逻辑回归建立多基因诊断模型并比较单基因和多基因模型诊断效能。
qPCR检测感染模型中候选基因表达
建立体外幽门螺杆菌(H. pylori)感染模型。用H. pylori ATCC 26695菌株以MOI 100:1感染GES-1和AGS细胞系24小时,检测mRNA水平。使用TRIzol试剂提取总RNA,HiScript III RT SuperMix反转录,Taq Pro Universal SYBR qPCR Master Mix和qTOWER3/G系统进行qPCR。基因表达水平用2^-ΔΔCt方法计算,GAPDH为内参。
候选生物标志物的生存分析
从TCGA数据库下载TCGA-STAD基因表达和生存数据,使用“survival”包分组核心免疫基因,采用Cox回归计算HR并绘制Kaplan-Meier生存曲线,P < 0.05为显著。
单细胞数据处理与分析
运用Python库“Scanpy”对单细胞数据进行质量控制、计数归一化、样本整合和批次效应校正,基于标记基因聚类细胞,用“DotPlot”“FeaturePlot”“VlnPlot”等函数可视化基因表达模式。
免疫细胞浸润分析
使用Immune Cell Abundance Identifier(ImmuCellAI)评估组织中免疫细胞浸润情况,Spearman相关分析探索免疫细胞与核心免疫相关基因的关系。
调控网络的构建
在NetworkAnalyst 3.0平台获取miRNA-mRNA和TF-mRNA相互作用信息,导入数据后用Cytoscape可视化miRNA-mRNA-TF调控网络。
PART·3 分析结果
鉴定胃癌患者和幽门螺杆菌感染者的共同差异表达基因(DEGs)
从胃癌的GSE54129数据集中识别了1793个差异表达基因(DEGs),从幽门螺杆菌感染的GSE60427数据集中识别了1468个DEGs。通过Venn图交集,找到了245个共同DEGs(188个上调,57个下调)。GO和KEGG分析显示,这些基因主要涉及中性粒细胞趋化、细胞因子、免疫和炎症通路,揭示了胃癌与幽门螺杆菌感染的关系。
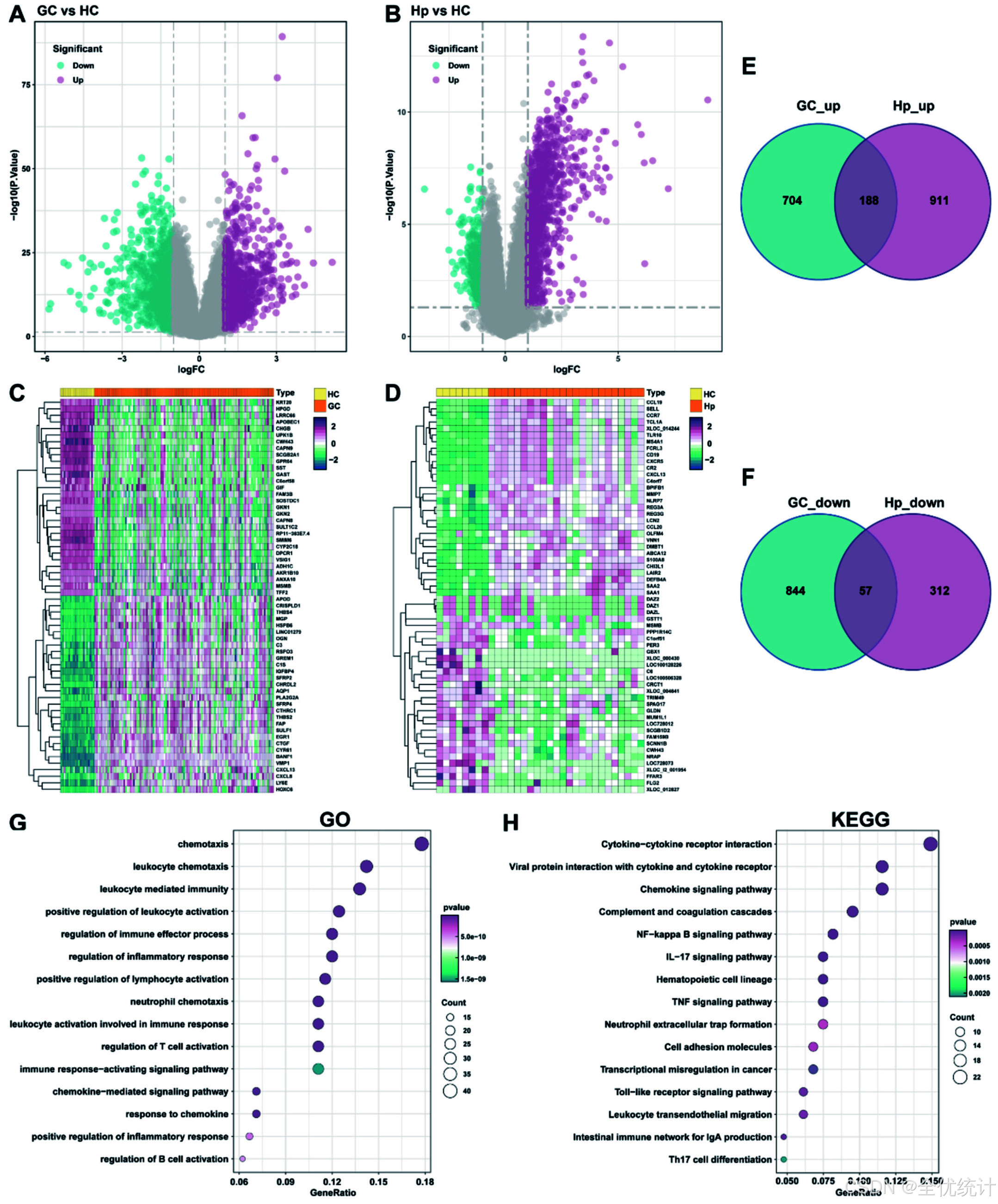
图2. 鉴定胃癌(GSE54129)和幽门螺杆菌感染(GSE60427)患者的差异表达基因(DEGs)。(A, B) GSE54129和GSE60427中所有DEGs的火山图,绿色表示下调的DEGs,紫色表示上调的DEGs。(C, D) GSE54129和GSE60427中前60个DEGs的热图。(E, F) Venn图鉴定了共同上调和共同下调的DEGs。(G, H) GO和KEGG通路富集
通过WGCNA鉴定胃癌与幽门螺杆菌感染相关的免疫核心基因
对GSE54129和GSE60427数据集进行了WGCNA分析,识别出与胃癌和幽门螺杆菌感染相关的关键模块,并计算了模块特征值与样本特征的相关性。通过与差异表达基因(DEGs)交集,发现75个共同的DEGs。GO和KEGG富集分析显示,这些DEGs主要涉及免疫反应、炎症和细胞因子通路。通过交集Immprot和InnateDB数据库中的免疫基因,识别出17个免疫相关基因。
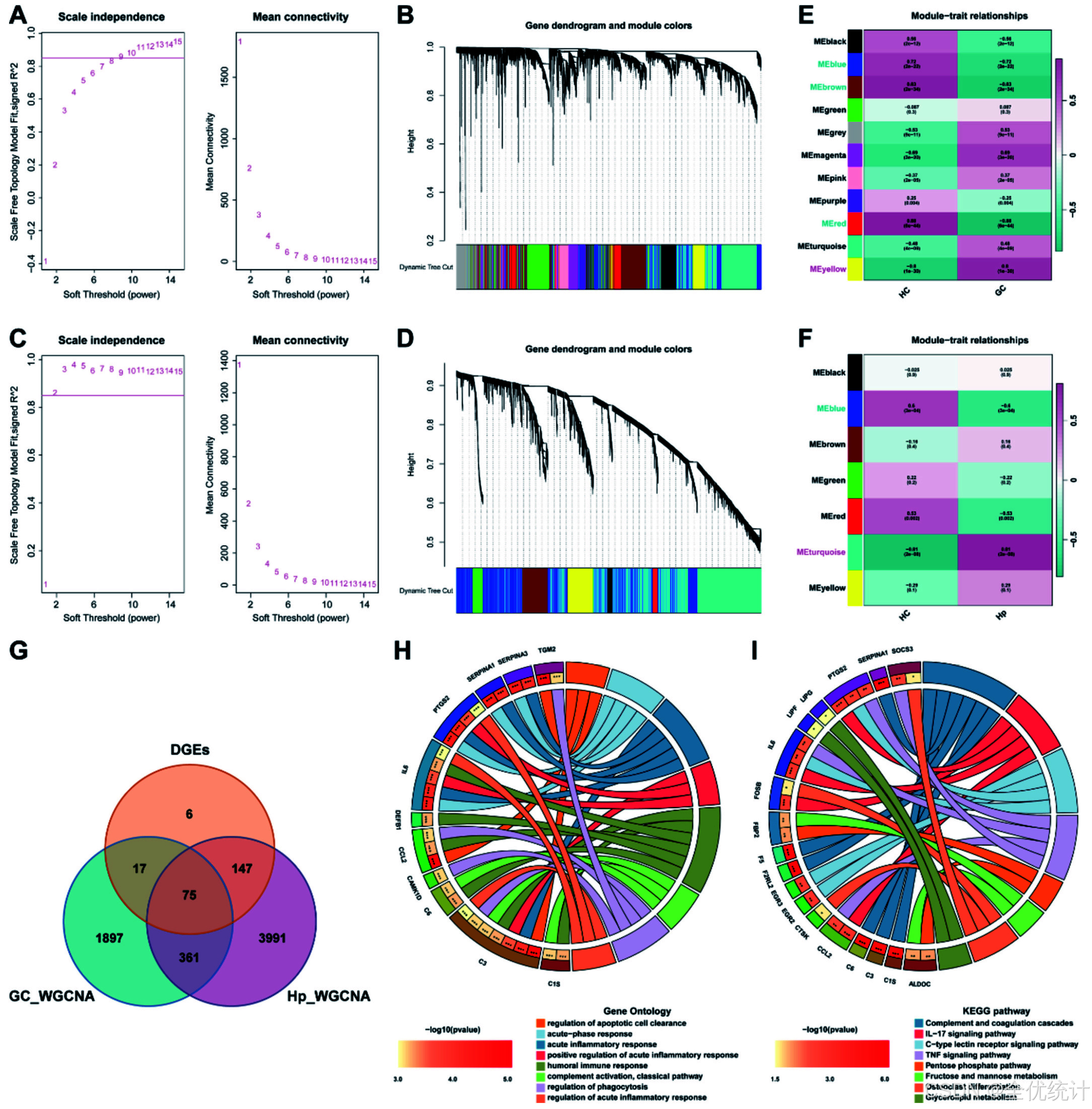
图3. 胃癌和幽门螺杆菌的WGCNA分析。(A, C) 胃癌(GSE54129)和幽门螺杆菌(GSE60427)中软阈值的选择。 (B, D) 通过动态树切割算法绘制的胃癌和幽门螺杆菌基因聚类树状图。 (E, F) 胃癌和幽门螺杆菌中模块与临床特征的关联热图。 (G) Venn图展示了通过WGCNA和DEGs获得的共同基因的交集。
使用随机森林模型识别候选生物标志物
为了鉴定潜在的胃癌和幽门螺杆菌感染诊断基因,使用随机森林算法分析了17个免疫相关基因。胃癌数据集识别了CTSK、NR4A3、SOCS3、C3、C1S、IL6、IFITM1和F2RL2等八个基因,幽门螺杆菌数据集识别了C3、IFITM1、CTSK、NR4A3和IFI16等五个基因。交集基因CTSK、NR4A3、C3和IFITM1作为候选诊断基因进一步分析。结果显示,四个基因在疾病组中的表达显著上调。通过qPCR实验,发现CTSK、C3和IFITM1在AGS细胞中表达更高,并且幽门螺杆菌感染后表达显著增加(P < 0.05)。这些结果表明,CTSK、C3和IFITM1可能是幽门螺杆菌相关胃癌的潜在诊断标志物。
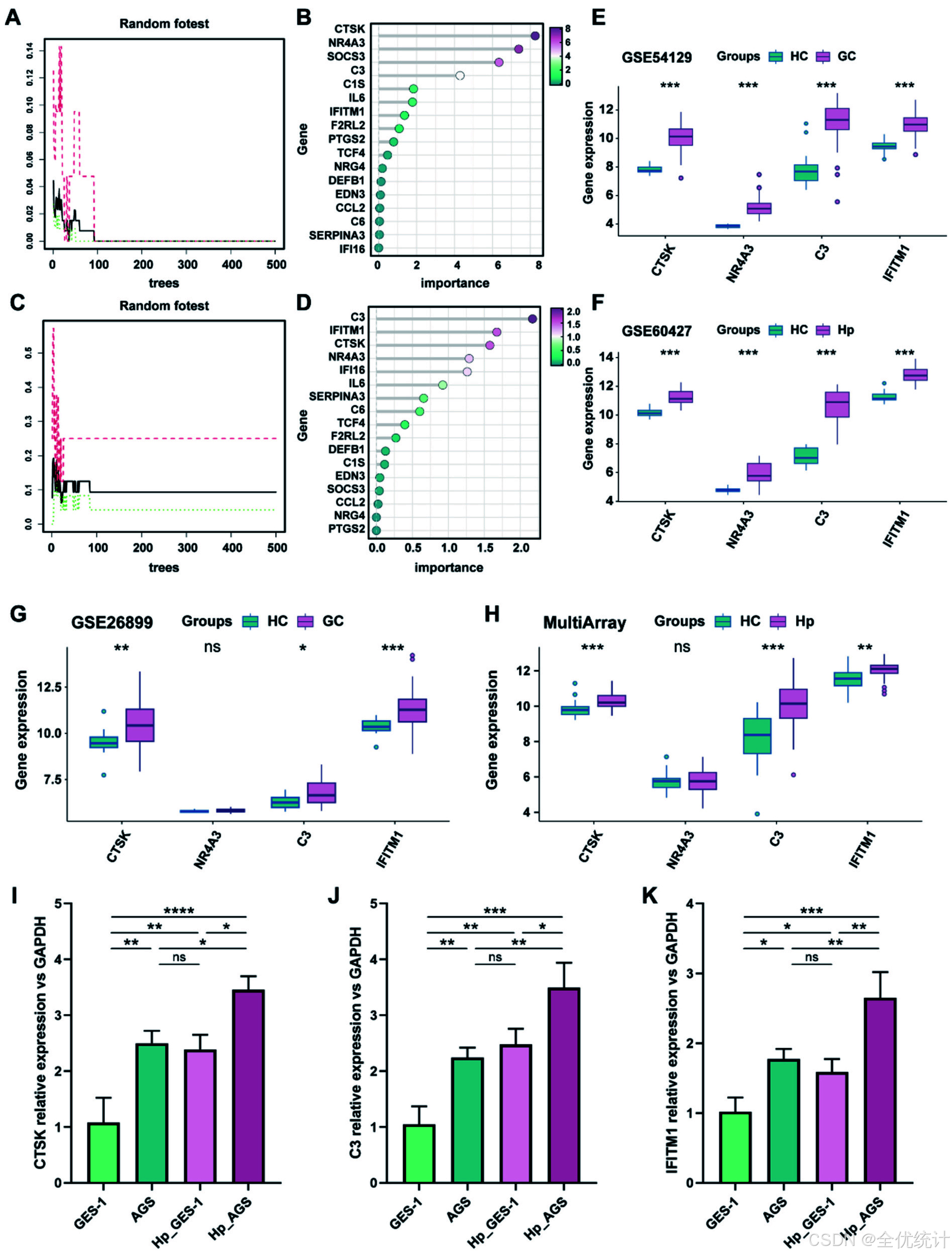
图4. 通过机器学习鉴定候选生物标志物(A, B) 基于随机森林算法,胃癌数据集中树的总数与误差率之间的关系,以及基因相对重要性评分的排名。(C, D) 基于随机森林算法,幽门螺杆菌数据集中树的总数与误差率之间的关系,以及基因相对重要性评分的排名。(E-H) CTSK、NR4A3、C3和IFITM1在GSE54129、GSE60427
胃癌和幽门螺杆菌感染患者候选生物标志物的诊断潜力与临床价值
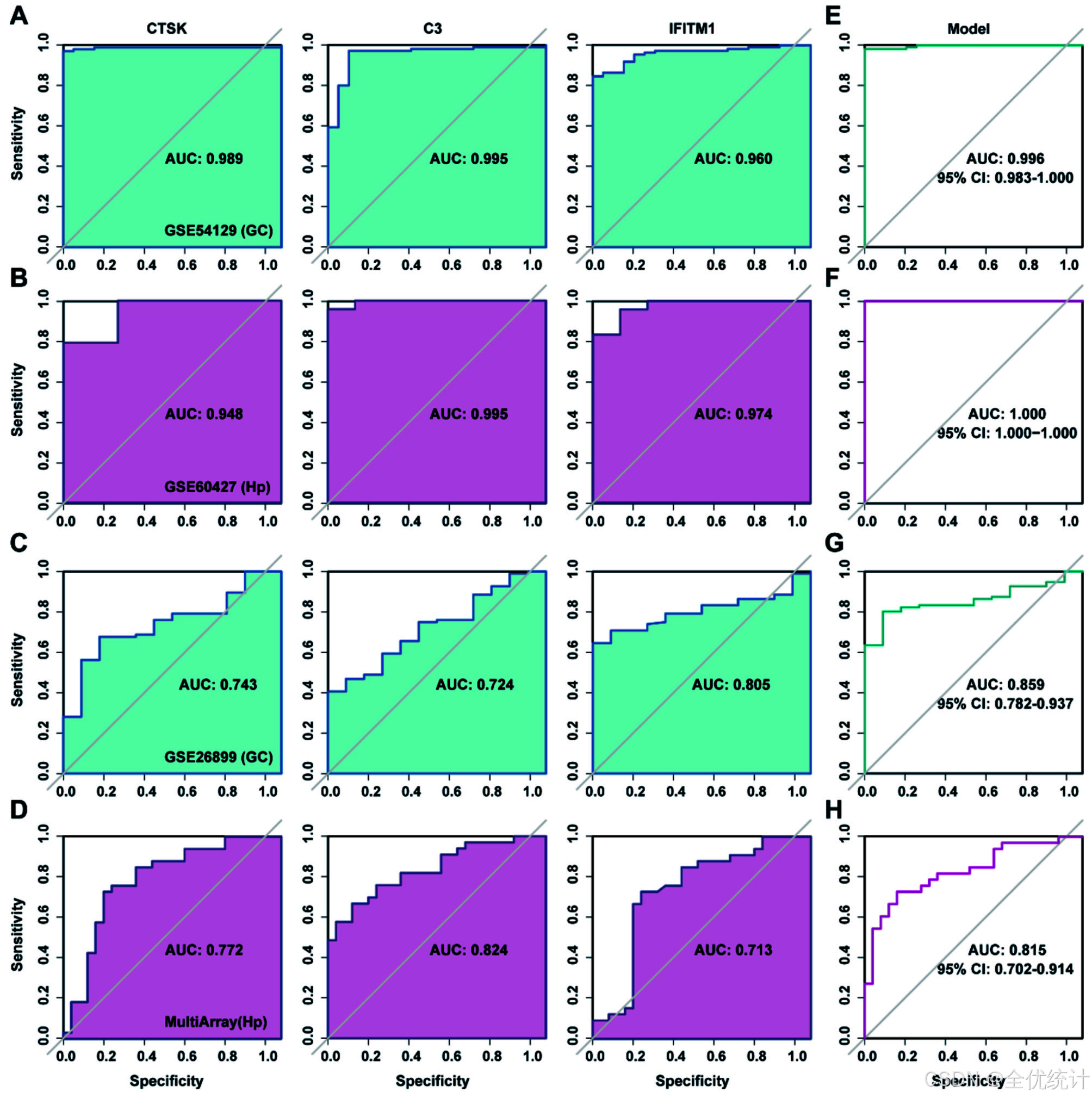
图5. CTSK、C3和IFITM1在胃癌和幽门螺杆菌感染患者中的诊断潜力。 (A-D) 在胃癌和幽门螺杆菌感染的测试和验证数据集中,三种共享基因的ROC曲线。 (E-H) 在胃癌和幽门螺杆菌感染的测试和验证数据集中,多标志物诊断模型的ROC曲线。
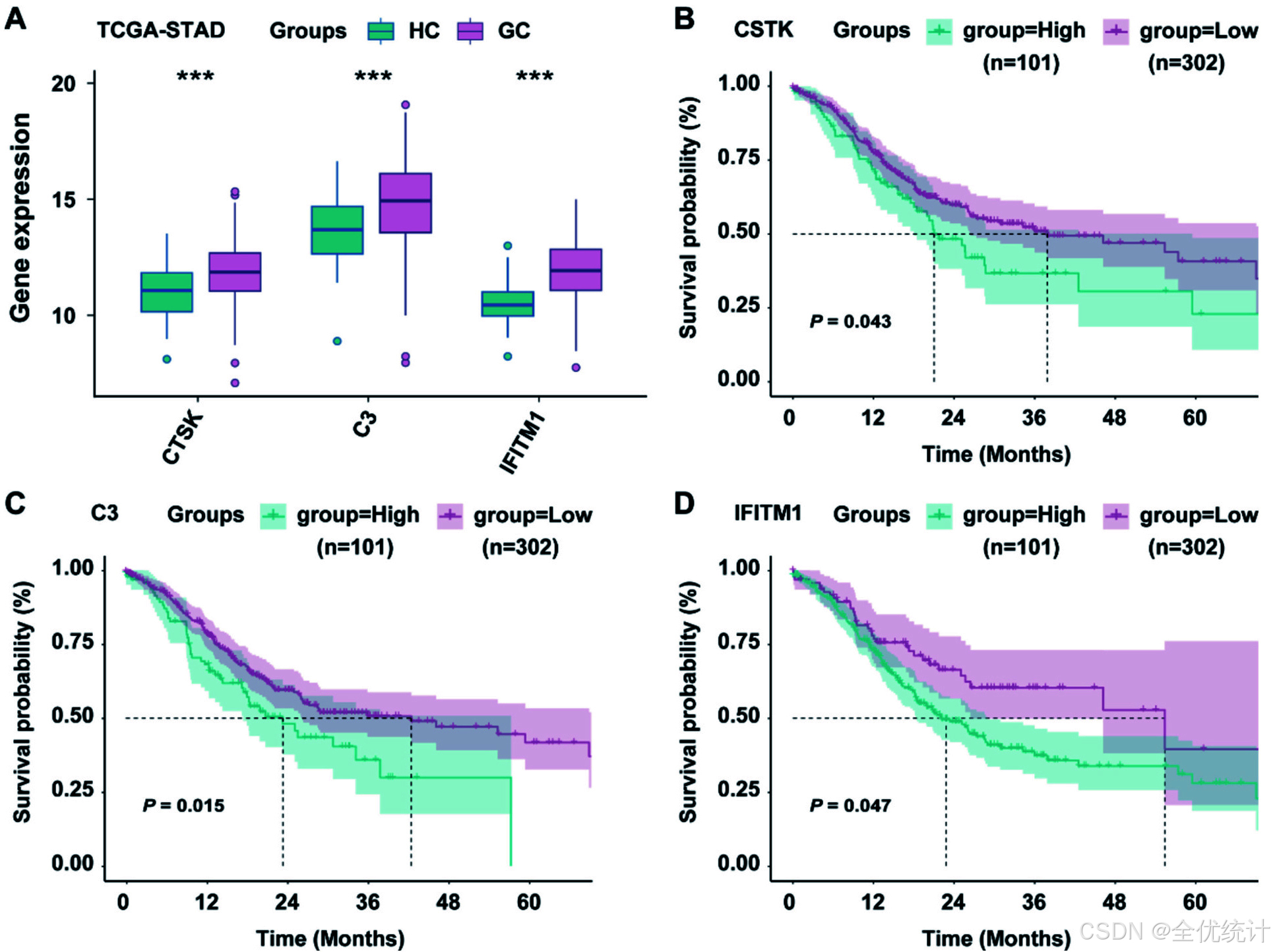
图6. CTSK、C3和IFITM1在胃癌患者中的临床价值(A) TCGA-STAD数据集中CTSK、C3和IFITM1的表达水平。*** P < 0.001(B) 高表达组与低表达组CTSK在胃癌患者中的生存分析。(C) 高表达组与低表达组C3在胃癌患者中的生存分析。(D) 高表达组与低表达组IFITM1在胃癌患者中的生存分析。
关键免疫相关基因在单细胞RNA测序中的表达模式
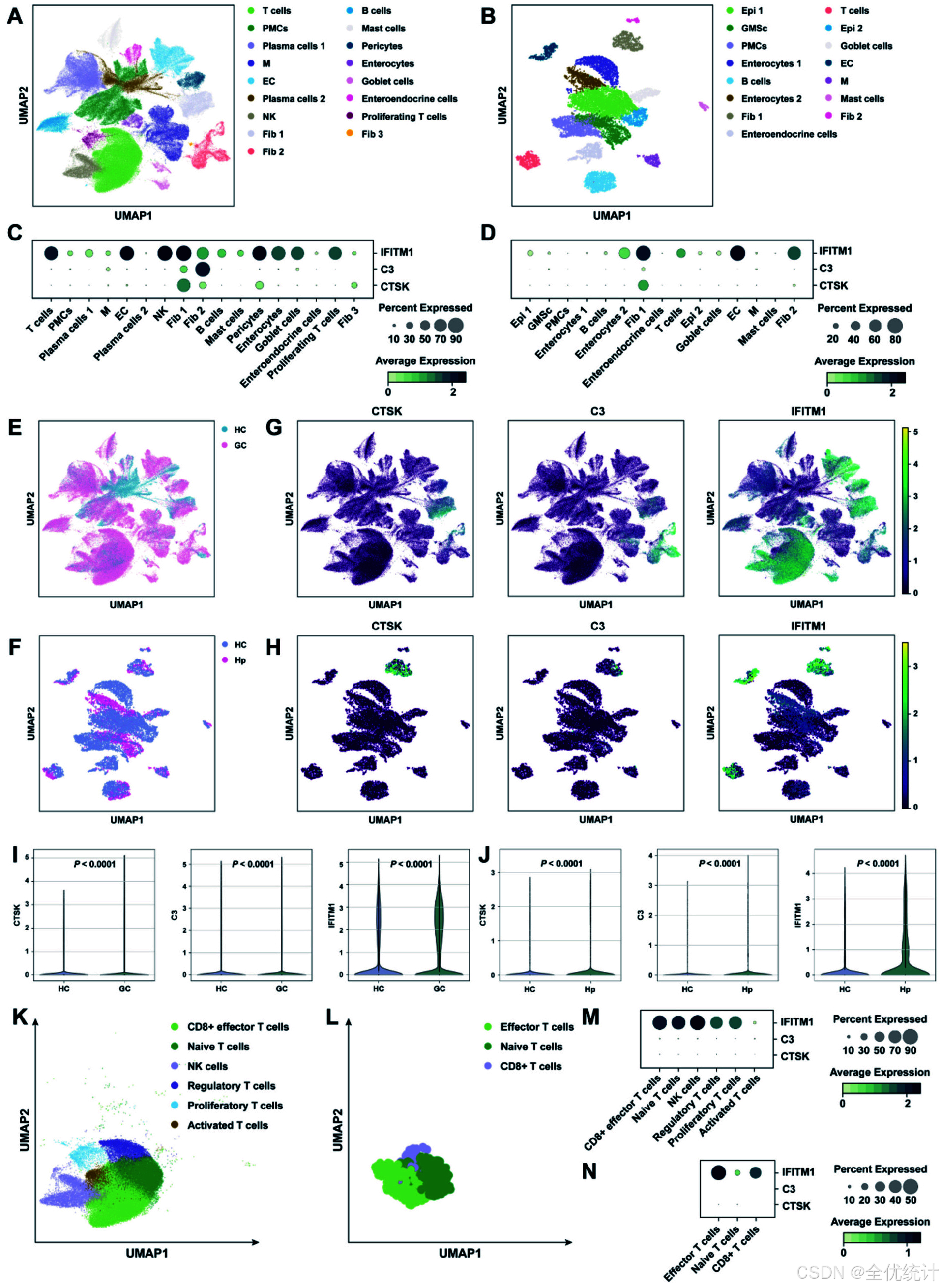
图7. 胃癌和幽门螺杆菌感染患者胃组织的单细胞转录组特征(A) 胃癌scRNA-seq数据集(GSE183904)中158,641个细胞的UMAP可视化。(B) 幽门螺杆菌scRNA-seq数据集(GSE134520)中29,241个细胞的UMAP可视化。(C, D) 点图展示了胃癌和幽门螺杆菌感染患者中三种核心基因在不同细胞群体中的表达水平。(E, F) 胃
PART·4 讨论
H. pylori感染在胃癌的发生发展中起着重要作用,其机制涉及细菌毒力、免疫反应和环境因素的相互作用。通过生物信息学分析转录组数据,本研究揭示了胃癌与H. pylori感染的转录特征,发现免疫相关基因如CTSK、C3和IFITM1可能作为潜在的诊断标志物。通过随机森林算法筛选出了四个核心免疫基因,进一步验证了它们在胃癌和H. pylori感染患者中的表达,并通过scRNA-seq分析确认它们在纤维母细胞相关细胞中主要表达。研究表明,这三种基因可能作为潜在的治疗靶点,用于H. pylori相关胃癌的早期诊断和治疗。
PART·5 小优结语
这篇文章真是开创性地把多组学数据(转录组学、临床数据)和单细胞分析相结合,再加上机器学习,选定了幽门螺杆菌-胃癌共病这一主题,简直就是创新满满!对那些准备毕业的小伙伴来说,这个方法简直是首选,做起来悄无声息,效果还杠杠的!























 5705
5705

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








