2024-09-01 ,在蛋白质结构研究领域,复旦大学的研究团队创建了Dynamic PDB数据集,这是一种新的数据集 Dynamic PDB,目的在捕获蛋白质的动态行为,以及一套全面的物理特性,例如原子速度和力、势能和动能以及模拟环境的温度。弥补了传统静态蛋白质数据库的不足,为深入理解蛋白质的动态行为和相关模型设计提供了宝贵的资源。
一、研究背景:
蛋白质作为生物体内的重要分子,不仅其静态结构对科学研究至关重要,其动态行为同样是理解其功能的关键。然而,动态蛋白质数据集的有限可用性、多样性和异质性限制了对蛋白质动态行为的深入研究。
目前遇到的困难和挑战:
1、动态蛋白质数据集的规模和多样性不足,难以全面捕捉蛋白质的动态变化。
2、缺乏包含详尽物理属性信息的数据集,如原子速度、力、能量等,这些信息对于理解蛋白质动态行为至关重要。
3、现有数据库主要关注蛋白质的静态结构,对动态行为的分析和预测能力有限。
数据集地址:Dynamic PDB|蛋白质结构数据集|分子动力学数据集
二、让我们一起来看一下Dynamic PDB数据集
Dynamic PDB是一个大规模的蛋白质动态行为数据集,包含了大约12.6K个蛋白质,每个蛋白质都经过了长达1微秒的全原子分子动力学(MD)模拟,以捕捉其构象变化。此外,数据集还提供了包括原子速度和力、蛋白质的势能和动能以及模拟环境的温度等物理属性。
数据集构建 :
数据集的构建过程包括从蛋白质数据银行(PDB)中选择蛋白质结构、预处理(包括选择、清洗和补全)、进行分子动力学模拟以及分析动态行为。
数据集特点:
1、精细的时间采样间隔(1皮秒),能够捕捉更详细的蛋白质动态路径。
2、扩展的时间采样时长(长达1微秒),有助于更全面地理解蛋白质的重要构象变化。
3、丰富的物理属性数据,包括原子速度、力、能量和模拟环境的温度等。
数据集使用方法 :
利用Dynamic PDB数据集进行蛋白质动态行为的分析,通过基准测试评估现有方法在轨迹预测任务上的性能,并开发新的模型来提高对蛋白质动态的预测能力。
基准测试 :
研究团队使用Dynamic PDB数据集对现有的最先进方法进行了评估,包括轨迹预测任务。通过与现有数据集的比较,展示了Dynamic PDB数据集在捕捉蛋白质动态行为方面的优势。
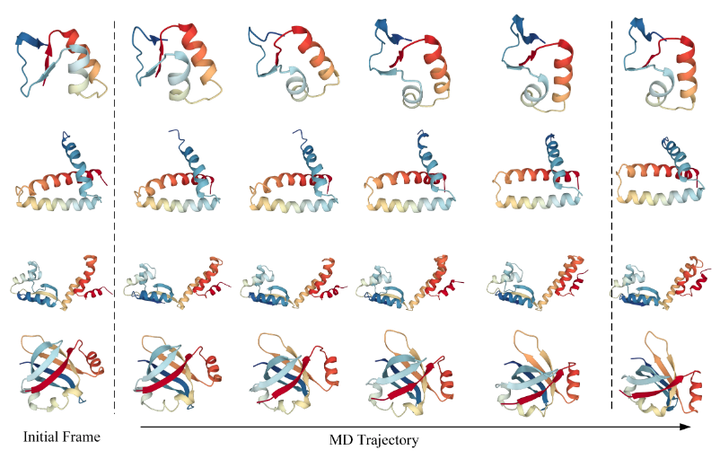
蛋白质轨迹可视化,以更高的时间分辨率存储,提供了蛋白质轨迹的更详细描述。
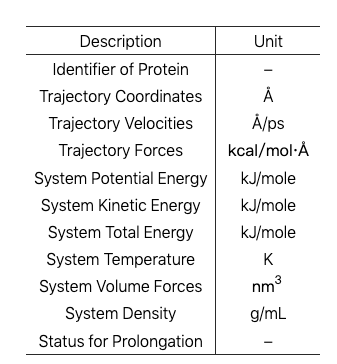
建议数据集的属性
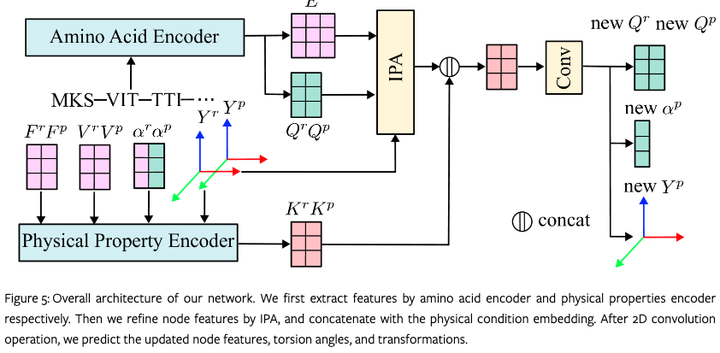
我们网络的整体架构。首先,我们分别通过氨基酸编码器和物理性质编码器提取特征。然后我们通过 IPA 细化节点特征,并与物理条件嵌入进行拼接。在 2D 卷积操作之后,我们预测更新的节点特征、扭转角和变换。
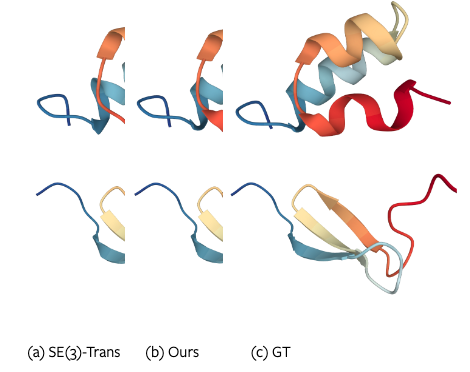
2ERL_A(上)和 3TVJ_I(下)的定性结果。我们的预测更接近 GT。
三、让我们展望数据集的应用场景:
比如,你是一位计算生物学家,对蛋白质工程充满热情。目前正在尝试设计一种全新的蛋白质,这种蛋白质能够作为一种生物催化剂,专门分解某种工业废水中的有毒化学物质。这个目标听起来是不是很有挑战性?
首先,我们得了解这种有毒化学物质的结构,以及它如何与蛋白质相互作用。这时,Dynamic PDB数据集就派上了大用场。我们利用这个数据集,通过分子动力学模拟,观察了成千上万种蛋白质与这种化学物质的潜在结合方式。
我们就像是在看一场分子层面的舞蹈,蛋白质和化学物质相互靠近,然后“拥抱”在一起。但是,我们不仅仅是在看热闹,我们是在寻找那个完美的“拥抱”,那个能让化学物质被分解的结合方式。
通过Dynamic PDB数据集,我们能够看到蛋白质的每一个微小动作,每一个可能的变形。这就像是我们有了一台超级慢动作摄像机,能捕捉到蛋白质在遇到化学物质时的每一个变化。
我们发现,有一种蛋白质在与化学物质结合时,它的几个关键残基会发生有趣的变化,这些变化使得蛋白质能够更紧密地抓住化学物质,并且引导它走向一个能够被分解的构象。
接下来,我们利用这些信息,通过计算方法对蛋白质进行了一些改造,增强了那些关键残基的灵活性,让它们能够更有效地与化学物质结合。我们还在蛋白质的其他部位做了一些调整,以确保蛋白质在催化反应中能够保持稳定。
最后,我们将这个经过计算设计改造的蛋白质送到实验室进行实际测试。结果令人兴奋,这种改造后的蛋白质确实能够高效地分解那种有毒化学物质,而且它的活性和选择性都比自然界中现有的任何蛋白质都要好。
通过Dynamic PDB数据集,它让我们能够以前所未有的细节和精度来模拟和预测蛋白质的结构变化。这不仅仅是科学上的突破,更是对环境和工业产生积极影响的创新。这就是为什么我对计算生物学如此充满热情,Dynamic PDB数据集让我们能够将想象中的蛋白质变为现实,解决实际问题。























 1453
1453

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








