基本概念
提取连通分量的过程实际上也是标注连通分量的过程,通常的做法是给原图像中的每个连通区分配一个唯一代表该区域的编号,在输出图像中该连通区内的所有的像素值就赋值为该区域的编号,我们将这样的输出图像称为标注图像。这里介绍一种基于形态学的膨胀操作的提取连通分量的方法。
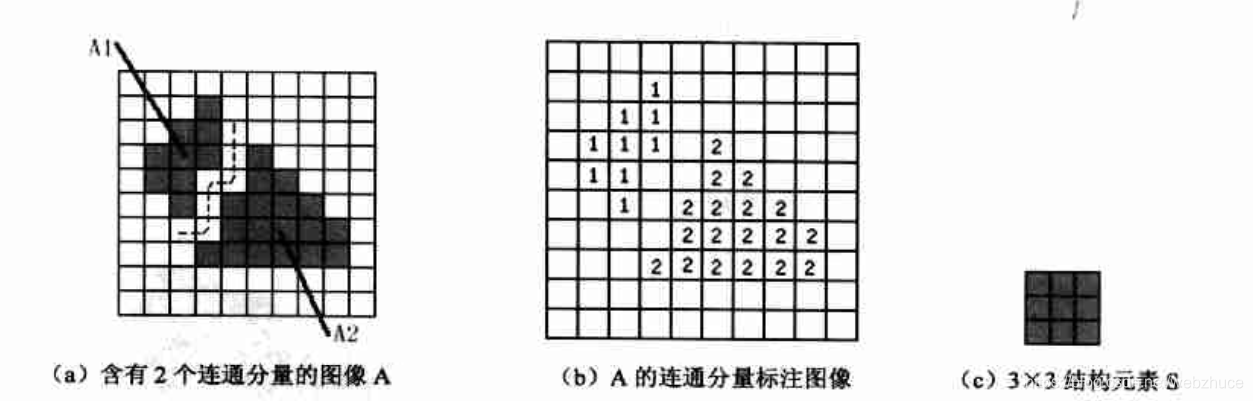
以8连通的情况为例,对于图(a)的内含多个连通分量的图像A,从仅为连通分量A1内部某个的图像B开始,不断采用如图©所示的结构S进行膨胀。由于其他连通分量与A1之间至少有一条一像素宽的空白缝隙(如图(a)中的虚线),3*3的结构元素保证了只要B在区域A1内部,则每次膨胀都不会产生位于图像A中其他连通区域之内的点,这样只需用每次膨胀后的结构图像和原始图像A相交,就能把膨胀限制在A1内部。随着对B的不断膨胀,B的区域不断生长,但每次膨胀后与图像A的相交又将B限制在连通分量A1地内部,直到最终B充满整个连通分量A1,对连通分量A1地提取完毕。
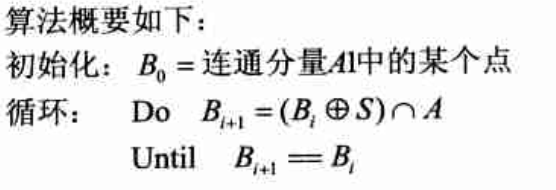
示例演示
我们用OpenCV实现连通分量提取函数,然后进行细菌计数。完整工程代码。
/*
*only process binary image
*iseight = true means eight connected, otherwise four connected
*/
int LabelConnectedComponent(const cv::Mat &src, cv::Mat &dst, bool iseight = true)
{
Mat structelement;
if(iseight)
structelement = getStructuringElement(MORPH_RECT, Size(3, 3));
else
structelement = getStructuringElement(MORPH_CROSS, Size(3, 3));
dst = Mat::ones(src.size(), src.type());
Mat tmp = Mat::ones(src.size(), src.type()); // save last reuslt image
Mat img = Mat::ones(src.size(), src.type()); //image B
int labelnum = 0; //label of connected component
Mat backupsrc;
src.copyTo(backupsrc);
for(int i = 0; i < backupsrc.rows; i++)
{
for(int j = 0; j < backupsrc.cols; j++)
{
if(backupsrc.at<uchar>(i, j) == 255)
{
Mat img = Mat::ones(src.size(), src.type());
img.at<uchar>(i, j) = 255;
img.copyTo(tmp); //Temporary save
labelnum++;
while(true) // until not change
{
cv::dilate(img, img, structelement);
bitwise_and(img, src, img);
//if img do not change, this connected component is finished
if (cv::countNonZero(img - tmp) == 0)
break;
img.copyTo(tmp);
}
//label the connected component
for(int r = 0; r < img.rows; r++)
{
for(int c = 0; c < img.cols; c++)
{
if(img.at<uchar>(r, c) == 255)
{
backupsrc.at<uchar>(r, c) = 0;
dst.at<uchar>(r, c) = labelnum;
}
}
}
}
}
}
return labelnum;
}
先对原始图像进行膨胀

然后进行细菌统计,实际的细菌个数为21。






















 2452
2452











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








