

小伙伴们,大家好。因研究方向的调整,最近笔者开始进行单个核苷酸多态性(Single Nucleotide Polymorphism, SNP)及长链非编码RNA(long non-coding RNA, lncRNA)的研究。前段时间给大家推荐过一本风湿免疫领域Top期刊 Arthritis & Rheumatolog(详情请戳这个杂志IF平均一年涨一分,今年9.586,明年将破10,不要错过!)。在检索相关文章的时候,发现中国科学院院士、中国科学院生物物理研究所研究员陈润生课题组于今年六月份在该杂志发表了题目为《Association of the polymorphism rs13259960 in SLEAR with predisposition to Systemic Lupus Erythematosus》的研究论文,细细品读,如醍醐灌顶!

该研究首次报道系统性红斑狼疮(systemic lupus erythematosus, SLE)易感长链非编码基因并命名为SLEAR,SLEAR在SLE患者外周血单个核细胞(Peripheral blood mononuclear cells, PBMC)中的表达水平显著低于正常对照人群,且在rs13259960(A>G)位点发生突变的SLE患者中的表达水平更低。rs13259960位于SLEAR的增强子位点,该位点的突变会减弱STAT1与该增强子的结合,从而影响SLEAR的表达水平。SLEAR能够与ILF2、hnRNPF和TAF15结合形成复合物来调控抗细胞凋亡基因的表达。体外实验表明SLEAR能够调控细胞的凋亡,并且在SLE病人体内SLEAR的表达水平也与凋亡显著相关。
具体易感位点rs13259960与SLEAR的调控关系及SLEAR在SLE疾病发展中的作用机制, 让我们一起品读文章。

 研究内容
研究内容
第一部分:鉴定中国汉族人口中SLE患者lncRNA相关的易感SNP位点
首先作者结合前期的汉族受试者的SLE全基因组关联研究(genome-wide association study,GWAS)的SNP信息及3个数据库(GENCODE, lincRNA, H-InvDB)中的lncRNA信息,筛选出了70个位于lncRNA基因区域上的SNP位点。该团队进一步通过对SLE患者与对照组(3,509名SLE患者和8,246名健康对照样本)的重复验证研究,最终确定了位于SLEAR基因上的易感位点rs13259960。接下来分析SLEAR的特性:长度(约为2000个核苷酸),表达(相对于正常人表达降低),细胞分布(约98%分布在核内)。

第二部分:rs13259960[G]降低SLEAR增强子活性
接下来作者分析rs13259960与SLEAR的调控关系。首先作者试图评估rs13259960不同等位基因对SLE患者PBMC中SLEAR的表达影响。但由于A/G基因型之间的样本量不匹配:A/G 基因型(n = 22)、G/G基因型(n = 3)和A/A基因型基因型(n = 103),作者采用置换检验。结果证实在A/A基因型,SLEAR是表达显著上调。rs13259960位于染色质开放区域并具有组蛋白H3K4me1和H3K27ac修饰特性,因此作者推测该位点可能为增强子区域。为验证该假设,作者采用荧光素酶报告基因检测技术,发现A/A基因型可产生较高的荧光素酶活性。

接着作者通过应用染色体构象捕获(Chromosome conformation capture,3C)技术,证明了rs13259960所在的增强子位点可与SLEAR启动子位点形成环状结构发挥作用。
Motif序列分析发现rs13259960与转录因子STAT1结合位点存在重叠。通过桑格测序及ChIP-PCR实验表明,STAT1与rs13259960中A基因型具有更高的亲和力。此外,沉默STAT1表达后,SLEAR水平明显降低。到这里,rs13259960与SLEAR的调控关系问题得以解决。

第三部分:SLEAR调控细胞凋亡
接下来,作者分析SLEAR的功能及下游机制。作者采用shRNA沉默技术,先通过RNA-seq寻找差异基因,enriched gene ontology(GO)分析显示细胞凋亡基因被最显著富集,并采用gene set enrichment分析进行验证,最终确定了3个凋亡相关基因:BCL2, BIRC2和XIAP。此外,沉默SLEAR表达,Jurkat细胞凋亡比例增加,增殖能力较低;SLEAR水平与SLE患者中CD3细胞凋亡比例呈负相关。
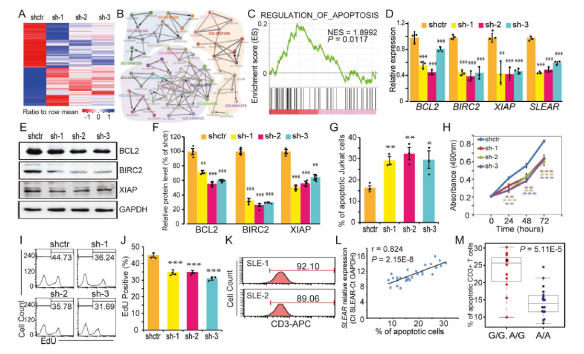
第四部分:SLEAR与 ILF2,hnRNP F和TAF15相结合
接下来,作者寻找SLEAR调控细胞凋亡的直接作用靶点。通过SLEAR pull-down+质谱/ Western blotting检测和RNA immunoprecipitation分析验证,发现SLEAR可与ILF2, hnRNP F和TAF15蛋白直接结合,并且四者形成RNA和蛋白的复合物来调控抗细胞凋亡基因的表达。

第五部分:CHIRP-seq揭示全基因组SLEAR结合位点
最后,作者采用CHIRP-seq(chromatin isolation by RNA purification sequencing)实验技术对全基因组范围内SLEAR的结合位点进行定位分析:大于4%的SLEAR结合在UTR3/5区域且富集倍数较高;SLEAR倾向于在启动子区域富集;SLEAR结合位点分布在除Y染色体的所有染色体上;SLEAR参与P53与其靶基因的结合。

 总结
总结
整篇文章作用机制示意图如下:

到这里,文章介绍完毕,小伙伴们有什么启发或者收获嘛?对于笔者而言,文中涉及到了多种高大上的研究方法,如ChIP-seq, CHIRP-seq。对于一般实验室,估计开展此类实验较为困难。不过,此文中SNP与lncRNA调控关系的研究方法,如3C,Sanger sequencing,allele-specific ChIP-qPCR,值得借鉴与学习。
有讲解不当的地方,欢迎小伙伴们在留言批评指正;有正在进行相关研究的小伙伴也可留言,大家伙集思广益,共同进步!
原文链接:
https://onlinelibrary.wiley.com/doi/abs/10.1002/art.41200

注:此推文未经许可禁止转载!
阅读推荐:- 【课题套路】入门级蛋白磷酸化研究
- 【课题套路】进阶级蛋白磷酸化研究——掐架的蛋白磷酸化和乙酰化
- 【课题套路】这样研究肿瘤干细胞你也能发9分文章
- 【课题套路】如何套牢ceRNA研究套路
- 动物模型系列之常见骨科疾病动物模型






















 1392
1392











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








