Falcon是PacBio公司开发的用于自家SMRT产出数据的基因组组装工具。Falcon分为三个部分:
- HGAP:PacBio最先开发的工具,用于组装细菌基因组,名字缩写自Hierarchical genome-assembly process(层次基因组组装进程)。 适用于已知复杂度的基因组,且基因组大小不能超过3Gb. 由于是图形界面,所以用起来会非常方便。
- Falcon:和HGAP工作流程相似,可认为是命令行版本的HGAP,能与Falcon-Unzip无缝衔接。
- Falcon-Unzip: 适用于杂合度较高或者远亲繁殖或者是多倍体的物种
层次基因组组装过程(HGAP)分为两轮.
第一轮是选择种子序列或者是数据集中最长的序列(通过length_cufoff设置),比较短的序列比对到长序列上用于产生高可信度的一致性序列。PacBio称其为预组装(pre-asembled), 其实和纠错等价。这一步可能会将种子序列在低覆盖度的区域进行分割(split)或者修整(trim),由falcon_sense_options参数控制,最后得到preads(pre-assembled reads)。
第二轮是将preads相互比对,从而组装成contigs(contig指的是连续的不间断的基因组序列, contiguous sequence)
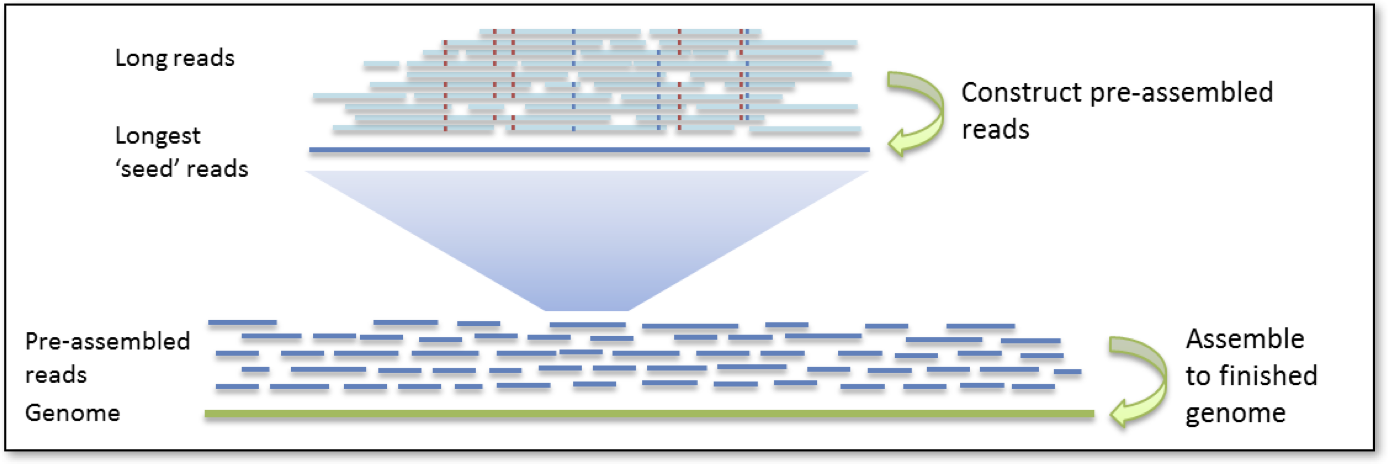
HGAP
基因组最后组装结果是单倍体,但实际上人类、动物和植物大部分的基因组都是二倍体,两套染色体之间或多或少存在的差异。这种差异在组装时就是“图”里的气泡(bubble)。PacBio开发的Falcon-Unzip就是用来处理“气泡”,把不同单倍型的contig分开。
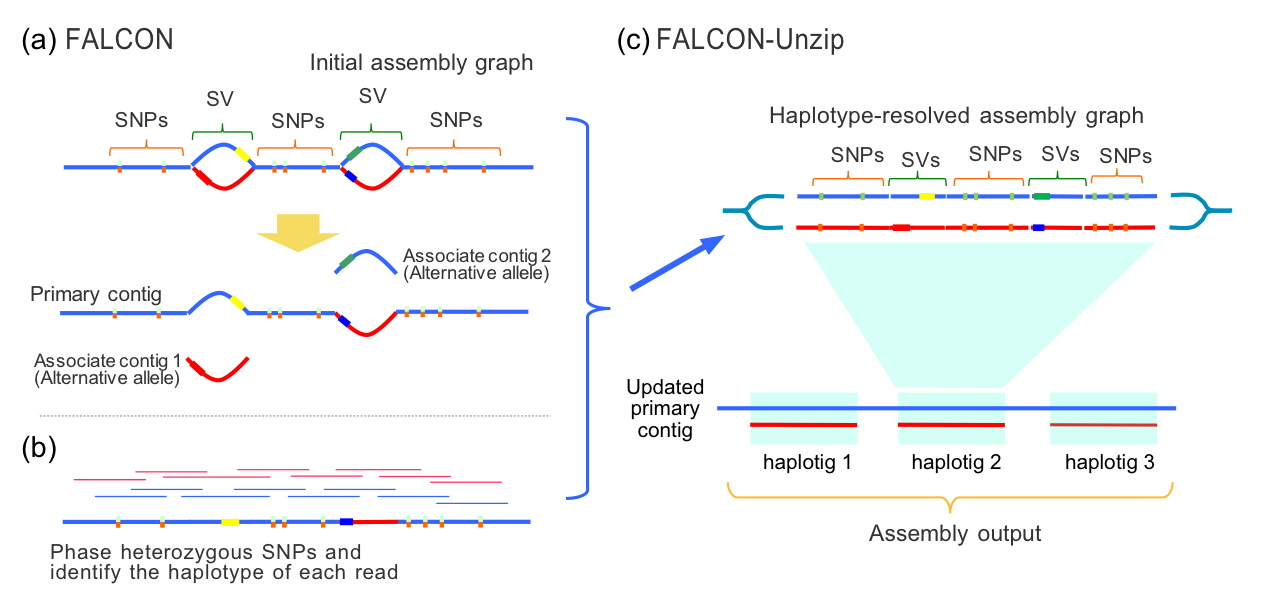
Falcon-Unzip
运行参数分类
Falcon的运行非常简单,就是准备好配置文件传给fc_run.py,然后让fc_run.py调度所有需要的软件完成基因组组装即可。只不过初学者一开始可能会迷失在茫茫的参数中,所以我们要对参数进行划分,分层理解。
参数从是否直接参与基因组组装分为任务投递管理系统相关和实际








 本文详细介绍了使用Falcon进行三代测序数据的基因组组装过程,包括准备数据、创建FOFN、配置文件的设定、运行程序以及评估组装结果。Falcon分为HGAP、Falcon和Falcon-Unzip三个部分,适用于不同类型的基因组组装。在实践中,通过调整参数和学习官方文档,可以优化组装效果。文章以E. coli为例,展示了组装步骤,并警告了可能遇到的内存消耗问题及其解决方案。
本文详细介绍了使用Falcon进行三代测序数据的基因组组装过程,包括准备数据、创建FOFN、配置文件的设定、运行程序以及评估组装结果。Falcon分为HGAP、Falcon和Falcon-Unzip三个部分,适用于不同类型的基因组组装。在实践中,通过调整参数和学习官方文档,可以优化组装效果。文章以E. coli为例,展示了组装步骤,并警告了可能遇到的内存消耗问题及其解决方案。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 2837
2837











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








